Introduction
The L1 cell adheesiomolekyyli geeni (L1CAM) on hermosolun adheesiomolekyyli, joka kuuluu immunoglobuliini-superperheeseen; sillä on keskeisiä toimintoja hermoston kehityksessä (Itoh and Fushiki, 2015). L1cam: n mutaatiot ovat liittyneet X-linkitettyihin neurologisiin oireyhtymiin, jotka on tiivistetty L1-sairauksiksi. Ne luokitellaan seuraavasti: X-linkitetty vesipää (XLH), joka johtuu Sylviuksen akveduktin ahtaumasta (HSAS), Masan oireyhtymästä (kehitysvammaisuus, afasia, laahustava kävely, addiktoidut peukalot), spastisesta parapareesista tyyppi 1 (SP1) ja corpus callosumin (ACC) X-linkitetystä ageneesista (Weller and Gartner, 2001; Itoh and Fushiki, 2015).
hgmd® Professional 2019.2-julkaisussa (https://portal.biobase-international.com/hgmd/pro/all.php) on raportoitu noin 282 tautia aiheuttavaa mutaatiota (DMs) L1CAM-geenissä. Muutokset L1CAM geeni ovat vaihtelevia; 282 potilaan mutaatioanalyysit paljastavat 51% missense-ja nonsense-mutaatioita, 25% poistoja, 5% lisäyksiä ja 19% liitoskohdan muutoksia, mutta l1cam: n Hiljaiset mutaatiot, joilla on patogeeninen potentiaali, olivat harvinaisia, ja hiljaiset mutaatiot jätettiin usein huomiotta erityisesti whole-exome sequencing (WES) – havaitsemisessa.
tässä tutkimuksessa seuloimme WESin avulla kiinalaisen raskaana olevan naisen sikiön DNA: ta, joka on ilmoittanut viidestä jatkuvasta raskaudesta, joilla on sikiön vesipää; l1cam-geenistä löytyi vain uusi Hiljainen mutaatio C.453g > T (P.Gly151=). Mielenkiintoista, lisäanalyysin kautta osoitimme, että hiljainen mutaatio loi potentiaalisen 5′: n liitoskohdan konsensussekvenssin, joka johtaisi 72 bp: n poistumiseen l1cam-proteiinin eksonista 5 ja 24 aminohaposta.
Tapausesittely
28-vuotias terve nainen ohjattiin meidän klinikallemme sen jälkeen, kun hän oli muissa sairaaloissa tehnyt neljä vapaaehtoista raskaudenkeskeytystä sikiön vesipään takia. Kaikki sikiöt olivat miehiä. Kun hän saapui sairaalaamme (Naistensairaala, School of Medicine, Zhejiangin yliopisto, Zhejiang, Kiina), hän oli jo viidennellä raskaudellaan 24.raskausviikolla sikiöllä hydrokefalus kuvatutkimusten perusteella. Geneettisen syyn selvittämiseksi sikiön verinäytteet otettiin 26 viikon raskausaikana. Tavanomaisia sytogeneettisiä tutkimuksia tehtiin sekä sikiönäytteille että vanhempainnäytteille, ja sikiönäyte analysoitiin edelleen yhden nukleotidin polymorfismiryhmällä (SNP) ja WES: llä.
tämä tutkimus toteutettiin Naistensairaalan eettisen komitean Zhejiangin yliopiston lääketieteellisen tiedekunnan suositusten mukaisesti, ja tietoon perustuva suostumus saatiin kaikilta tutkimukseen osallistuneilta Helsingin julistuksen mukaisesti. Tutkimusprotokolla hyväksyttiin Kiinan Zhejiangin yliopiston Naistensairaalan, School of Medicinen tarkastuslautakunnassa.
materiaalit ja menetelmät
karyotyyppi ja SNP-ryhmä
sikiön napanuoraveren ja perifeerisen napanuoraveren karyotyypit määritettiin tavanomaisella karyotyypityksellä vähintään 30 veren lymfosyyt, jotka kolkisiinit pysäyttivät metafaasissa. Viljeltyjen solujen g-banding-karyotyypit suoritettiin 320-400-kaistaisella tasolla noin 10 Mb: n resoluutiolla. SNP array suoritettiin cytoscan™ HD array (Affymetrix, USA) mukaan valmistajan ohjeiden, noin 2,600,000 markkereita mukaan lukien 750,000 SNP-koettimet ja 1,900,000 ei-polymorfism koettimet kattavan koko genomin kattavuus. Tiedot analysoitiin Chromosome Analysis Suite (Chas) – ohjelmistolla (Affymetrix, Santa Clara, CA), joka perustui grch37/hg19-kokoonpanoon. Kopionumerotuloksen ilmoituskynnykseksi asetettiin 500 kb merkkiluvun ollessa ≥50 voittojen osalta ja 200 kb merkkiluvun ollessa ≥50 tappioiden osalta.
koko-Eksome-sekvensointi
pääosan WES: stä toimitti Pekingin genomiikan instituutti. Genominen DNA uutettiin DNeasy Blood Kit (Qiagen, CA) ja sitten sirpaloitui Covaris LE220 (Massachusetts, USA) tuottaa pariloppuinen kirjasto (200-250 bp). Kaikki monistetut kirjastot suoritettiin bgiseq-500-alustalla, yksijuosteinen DNA sekoitettiin mgieasy™ DNA-kirjaston Prep Kit V1: een (BGI, Shenzhen, Kiina) ja sitten sekvensoitiin käyttäen 100SR-kemiaa bgiseq-500RS-suurinopeuksisella sekvensointipaketilla (BGI, Shenzhen, Kiina).
kohdennetusta sekvensoinnista ja suodattamisesta johdetut puhtaat lukemat (joiden pituus on 90 bp) yhdenmukaistettiin sitten ihmisen genomiviittauksen (hg19) kanssa Burrows-Wheeler Aligner (BWA)-Moninäköohjelmapaketin avulla (Li and Durbin, 2009). Linjauksen jälkeen, lähtötiedostoja käytettiin suorittamaan sekvensointi kattavuus ja syvyysanalyysi kohdealueen, yhden nukleotidin variantit (SNVs), ja indel calling, käytimme gatk ohjelmisto tunnistaa SNVs ja indels (McKenna et al., 2010), Kaikki SNVs ja indels suodatettiin ja arvioitiin useiden tietokantojen kautta, mukaan lukien National Center for Biotechnology Information (NCBI) Single-Nucleotide Polymorphism Database (dbSNP), HapMap, 1000 Genomes Project dataset, ja tietokanta 100 Kiinan terveitä aikuisia. Käytimme Condelia, SIFTIÄ, PolyPhen-2: ta, LRT: tä, Mutaatiomaistajaa ja Phylopia varianttien vaikutuksen ennustamiseen. Patogeeniset variantit arvioidaan American College of Medical Genetics and Genomics (ACMG) – järjestön (Richards et al., 2015). Mutaatioiden seulontaan käytettiin ihmisen Geenimutaatiotietokantaa (Human Gene Mutation Database, Hgmd). Kaikki mahdolliset patogeeniset variantit validoitiin Sanger-sekvensointimenetelmillä.
RNA-uutto, PCR ja sekvensointi
perifeerisen veren mononukleaarisolut (Pmbc) ja napanuoraveren mononukleaarisolut (CBMCs) eristettiin Ficollin tiheysgradienttierotuksella. Yhteensä RNA uutettiin Pmbc ja CBMCs käyttäen TRIzol (Takara, Japani). Uuttaa RNAS oli käänteinen-litteroitu käyttäen RT Kit (Takara, Japani). PCR suoritettiin käyttäen GoldStar Best Master Mixiä (Cwbio, Peking). Primer sekvenssit on lueteltu: L1CAM-DNA-5F, CCCACCCGTCCTTCCTA; L1CAM-DNA-5R, CGCTCGTCCTTGATGT; L1CAM-mRNA-4-6-F, GGTGTCCACTTCAAACCCAA; ja L1CAM-mRNA-4-6-R, GCGGCTCCTGTCAATCA. Sanger-sekvensointi suoritettiin ABI 3130-DNA-analysaattorilla.
tulokset
28-vuotias terve nainen ohjattiin klinikallemme sen jälkeen, kun hän oli tehnyt neljä vapaaehtoista raskaudenkeskeytystä sikiön vesipään takia. Kaikki sikiöt olivat uroksia (Kuva 1a). Sukutaulu näytti osoittavan XLH: ta. Hänellä oli jo viides raskaus 26. raskausviikolla. Sikiön ventriculomegalia havaittiin sikiön ultraäänitutkimuksella ja magneettikuvauksella, jotka osoittivat johdonmukaisesti hydrokefaluksen esiintymisen. He osoittivat, että molemminpuolinen aivokammio ja kolmas kammio olivat ilmeisesti laajentuneet, ja siellä oli vaikea vesipää aivosolujen intracerebral ja agenesis, corpus callosum (Kuva 1B).
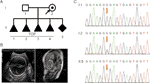
Kuva 1 A) perheen sukutaulu. TOP, raskauden keskeytys. B) sikiön kuvantamistutkimukset. Sikiön ultraäänitutkimus ja magneettikuvaus osoittivat, että sikiöllä oli vaikea vesipää. C) Sekvenssianalyysi genomisesta DNA: sta perheenjäseniltä. L1cam: n genotyypit olivat villityyppi, c. 453G > T Het, ja C. 453G > t Hom, I:1 (aviomies), I:2 (raskaana oleva nainen) ja II:5 (sikiö). Mutaatiosta kertovat punaiset nuolet.
mahdollisen geneettisen syyn selvittämiseksi teimme karyotyyppianalyysin ja SNP-järjestelmän analysoidaksemme sikiön verinäytteen emmekä löytäneet positiivisia löydöksiä. Choroidal neovaskularisaatio (CNV) tulos on talletettu geenin ilmentymä Omnibus (GEO); liittymisnumero on GSE133063, kuten alla (https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE133063).
sitten Wes havaitsi sikiön. Analyyttinen strategia patogeenisen variantin tunnistamiseksi on esitetty kuvassa S1. Varianttiluettelo (taulukko S1) saatiin seulomalla varianttitaajuudet, mutaatiotila ja periytymistila. Hydrokefalukseen liittyvät geenit (HP:0000238, http://compbio.charite.de/hpoweb/showterm?id=HP:0000238#id=HP_0000238) (taulukko S2) huomioon ottaen L1CAM-geenin ekson 5: ssä (NM_000425.3) ei havaittu muita merkittäviä mutaatioita lukuun ottamatta C.453G > T: n hiljaista mutaatiota. c.453G > T: tä ei raportoitu hgmd: ssä eikä Clinvarissa eikä sitä todettu dbSNP: ssä, gnomadissa eikä muissa tietokokonaisuuksissa. ACMG: N standardien ja ohjeiden mukaan (Richards et al., 2015), se ei ollut vielä saavuttanut kriteeriä ”patogeeninen” tai ”todennäköisesti patogeeninen”, mutta muita mahdollisia mutaatioita ei ollut; meillä ei ollut muuta vaihtoehtoa kuin tehdä lisäanalyysi löydetystä hiljaisesta mutaatiosta.
perinteisen ajattelun mukaan tämä emässubstituutio tapahtui kodonissa 151, joka koodaa glysiiniä, jolloin syntyy neutraali mutaatio (p.Gly151 = ). Tämä variantti vahvistettiin DNA: ssa, joka on otettu pariskunnan sikiön napanuoraverestä ja ääreisverestä Sanger-sekvensoinnilla (Kuva 1c). Nainen kantoi heterotsygoottista mutaatiota, ja hänen miehensä oli villin tyypin genotyyppi.
Mutaatiomaistajalla (http://www.mutationtaster.org/) c. 453G > t pisteytettiin ” tautia aiheuttavaksi.”Se osoitti, että proteiinin ominaisuudet saattavat vaikuttaa ja liitoskohtaa voidaan muuttaa; olimme uteliaita L1CAM-toiminnon mahdollisista liitosvaikutuksista tässä hiljaisessa mutaatiossa. Hiljaista mutaatiota testattiin seuraavilla online-ohjelmistotuotteilla: NetGene2 (http://www.cbs.dtu.dk/services/NetGene2/) ja NNSplice (http://www.fruitfly.org/seq_tools/splice.html); 5′ potentiaalisen liitospaikan ennustettiin syntyvän myös l1cam c: ssä.453g > T mutaatio ohjelmistotuotteilla (Kuva S2). Tulokset osoittivat, että tämä hiljainen mutaatio loi potentiaalisen 5′: n liitoskohdan 72 bp ylävirtaan normaalista eksoni 6/introni6: n liitoskohdasta (Kuva 2a). Jos näin on, voidaan löytää l1cam-lähetti-RNA:n (mRNA) pituusmuutos välillä I:2 ja II: 5 (Kuva 2a). RT-PCR suoritettiin käyttäen alukkeita, jotka oli suunniteltu vahvistamaan eksoneja 4-6 L1CAM mRNA: ssa. Itse asiassa tulokset osoittivat lyhyt bändi katkaisu sikiön cDNA PCR (II:5), kun taas bändi täydennetty l1cam mRNA sisälsi odotetun pitkä bändi aviomies cDNA PCR (I:1) ja pitkiä / lyhyitä nauhoja raskaana olevalla naisella cDNA PCR (I:2) (Kuva 2B). Monistetun fragmentin suora sekvensointi osoitti, että deleetio koski viimeisen 72 bp: n ekson 5: tä urospuolisessa sikiössä cDNA: ssa (nainen oli kantaja) (Kuva 2C). Meillä on ratkaisevat patogeeniset todisteet.
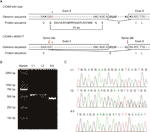
kuva 2 (A) Kaavamainen esitys ekson 5, intron 6, ja exon 6 organisaatio l1cam. (B) RT-PCR-analyysi l1cam cDNA: n eksoneista 5 ja 6 perifeerisen veren mononukleaarisoluista (pmbc) ja napanuoraveren Mononukleaarisoluista (CBMCs). RT-PCR-tuotteiden agaroosigeelielektroforeesi, joka on tuotettu I: 1:stä (aviomies), I:2: sta (raskaana oleva nainen) ja II: 5: stä (sikiö). (C) Sekvenssianalyysi RT-PCR tuotteen pmbc pari ja CBMCs sikiön.
tämä hiljainen mutaatio johti 24 aminohappoon L1CAM-proteiinia (jäämiä 151-174); Lys (K) korvattiin Glu (E): llä kodonissa 175 (Kuva 2a). Oli yhdenmukaistaminen useita l1cam proteiini sekvenssejä eri lajien ja säilyttäminen puuttuvat aminohapot l1cam poikki nisäkkäiden: Homo sapiens, Pan troglodytes, Bos taurus, Mus musculus ja Rattus norvegicus (Kuva 3A). Wild-tyypin ja c.453G > T-mutaation l1cam-proteiineja ennusti ohjelmisto CPHmodels-3.2-palvelin (http://www.cbs.dtu.dk/services/CPHmodels/) (kuva 3B). Immunoglobuliinin kaltainen (Ig-like) domeeni 2 (jäämät 134-230) villityyppisten ja silmukoivien l1cam-proteiinien mutaatio on esitetty kuvassa 3c. L1CAM c. 453G > t silmukoiva mutaatio muutti proteiinirakennetta, erityisesti Ig: n kaltainen domeeni 2
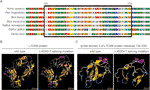
kuva 3 (A) useiden l1cam-proteiinisekvenssien kohdistaminen eri lajeihin. L1cam c.453 G > T johti siihen, että l1cam-proteiinin 24 aminohappoa (jäämiä 151-174) puuttui eri lajien säilyneeltä aminohappoalueelta. Mustassa pylväässä näkyvät puuttuvat aminohapot. (B) wild-tyypin ja c.453G > T-mutaation l1cam-proteiinin rakenteet, jotka ohjelmisto cphmodels-3.2-palvelin ennustaa. (C) immunoglobuliinin kaltaisen (Ig: n kaltaisen) domeenin 2 (jäämät 134-230) villin tyypin mutaation l1cam-proteiinin rakenteet.
Keskustelu
Wes havaitsi usein hiljaisia mutaatioita, mutta niihin ei ole kiinnitetty riittävästi huomiota, mikä johti DMs: n puuttumiseen. Tässä tutkimuksessa, palkkasimme WESin tutkimaan geneettistä syytä kiinalaisen perheen kanssa vesipää, mutta vain löytyi Uusi Hiljainen mutaatio L1CAM, joka pakotti meidät tekemään lisäanalyysin. Onneksi todistimme, että hiljainen mutaatio loi uuden 5 ’ liitospaikan ja oli DM.
L1CAM – mutaatiot voivat aiheuttaa X-linkitetyn L1-taudin, mutta kliiniset oireet ovat vaihtelevia; mutaatiot tuottavat odottamattomia fenotyyppejä. Tutkimuksessa viisi kärsivää sikiötä ovat kaikki miehiä, mikä sopii perintökuvioon. Sikiön ultraäänitutkimuksessa ja magneettikuvauksessa näkyy tyypillinen L1-tauti, johon kuuluvat muun muassa keltarauhasen XLH ja ageneesi. Se parantaa ymmärrystämme l1cam: n genotyyppi–fenotyyppikorrelaatiosta.
L1CAM c.453g > T: llä (P.Gly151 = ) ei aluksi arveltu olevan vaikutusta proteiinisekvenssiin. Mutta muut Hiljaiset mutaatiot, c.924C > T (P.Gly308 = ) ja c.645C > T (P.Gly215 = ), L1CAM-geenissä on raportoitu olevan DMs (Du et al., 1998; Vos et al., 2010). N.924c > T mutaatio johti uuden liitoskohdan aktivoitumiseen 69 bp 5′ normaaliin ekson 8/intron 8 luovuttajan liitoskohtaan, ja se on julistettu hydrokefalusta aiheuttavaksi alueeksi (Du et al., 1998). C. 645C > t l1cam: ssa 51 bp poistettiin uuden ekson 6/intron 6-luovuttajan liitoksen aktivoituessa (Vos et al., 2010). Nykyinen tutkimuksemme oli samanlainen; C.453G > T: n mutaatio loi potentiaalisen 5′: n liitoskohdan yläjuoksulle normaalista Exon 5/intron 5: n liitoskohdasta. Kaikki nämä Hiljaiset mutaatiot loivat uusia luovuttajien liitospaikkoja, jolloin eksoni ohitettiin. Se muistutti meitä kiinnittämään erityistä huomiota näihin hiljaisiin mutaatioihin,jotka voivat vaikuttaa proteiinien sekoittumiseen.
transmembraaniglykoproteiinina ja solujen adheesiomolekyylien immunoglobuliini-superperheen jäsenenä l1cam-proteiini voi olla vuorovaikutuksessa solun pinnalla useiden eri glykoproteiinien kanssa, ja homofiilinen sitoutuminen on luultavasti sen pääasiallinen vuorovaikutustapa (Wei and Ryu, 2012). IG: n kaltaisten domeenien 1-4 kiderakennetta neurofassiinissa koskevat tutkimukset viittasivat siihen, että monet patologiset L1-mutaatiot vaikuttavat näiden domeenien säilyneisiin aminohappojäämiin ja häiritsevät homofiilisia vuorovaikutuksia (Liu et al., 2011), erityisesti todennettuna funktiotutkimus Ig-like domain 2 (Zhao et al., 1998). Tutkimuksessamme spekuloimme, että L1CAM C.453g > t muutti Ig: n kaltaista domain 2: ta l1cam-proteiinin solunulkoisessa osassa, mikä johti epänormaaliin solunulkoiseen vuorovaikutukseen, jolloin signalointireitin aleneminen ei alkanut. Tämän l1cam-muunnoksen massaspektrometrialle omistettu lisätutkimus selventäisi erityisesti sitä, mitä molekyylikokonaisuutta solussa tuotetaan.
yhteenvetona WESin kautta raportoimme uudesta hiljaisesta mutaatiosta c. 453G > T L1CAM: ssa, joka tuottaa 5′: n liitoskohdan, joka vastaa vesipäätä. Tämän epänormaalin proteiinimuunnoksen ennustettiin muuttavan Ig: n kaltaista domeeni 2: ta, mikä saattaa vaikuttaa l1cam-proteiinin homofiiliseen sitoutumiseen. Lisäksi teimme raskausdiagnoosin raskaana olevalle naiselle, joka ilmoitti viidestä jatkuvasta vesipääraskaudesta. Sillä välin se ehdotti, että joitakin wesissä havaittuja hiljaisia mutaatioita ei tulisi jättää huomiotta; näiden mutaatioiden liittäminen toisiinsa oli välttämätöntä. Se tarjosi uuden geneettisen perustan sikiödiagnoosille ja ennen implantaatiota tapahtuvalle vesipäädiagnoosille.
tietojen saatavuus
julkisesti saatavilla olevat aineistot analysoitiin tässä tutkimuksessa. Tiedot löytyvät täältä: GSE133063 (https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE133063).
eettinen lausunto
tutkimukset, joihin osallistui ihmisiä, tarkasteli ja hyväksyi Kiinan Zhejiangin yliopiston Naistensairaalan tarkastuslautakunta. Osallistujien laillinen huoltaja/lähiomainen antoi kirjallisen suostumuksen osallistua tähän tutkimukseen. Kirjallinen tietoon perustuva suostumus saatiin yksityishenkilöiltä ja alaikäisiltä laillisilta huoltajilta/lähisukulaisilta tämän artikkelin mahdollisesti tunnistettavien kuvien tai tietojen julkaisemiseksi.
Tekijäosuudet
ys, YLi, MC, YLu, YQ ja YY tekivät kokeita. YS valmisteli luvut. MC ja YLi analysoivat WESin tiedot. YLu ja YQ suorittivat karyotype analysis ja SNP array. Otit näytteitä. HL ja FL toimittivat kuvantamistutkimuksia. Ys ja MD kirjoittivat käsikirjoituksen. Kaikki kirjoittajat lukivat ja hyväksyivät lopullisen käsikirjoituksen.
Rahoitus
tätä tutkimusta tuki Kiinan kansallinen Luonnontieteellinen säätiö (apuraha nro 81801441), joka on Zhejiangin maakunnan keskeinen tutkimus-ja kehitysohjelma (apuraha nro. 2019C03025), Kiinan kansallinen keskeinen tutkimus-ja kehitysohjelma (Grant No. 2016yfc1000703) ja Zhejiangin maakunnan lääketieteellinen Tieteellinen tutkimussäätiö (Grant No. 2014kya246).
Eturistiriitalausunto
kirjoittajat toteavat, että tutkimus tehtiin ilman kaupallisia tai taloudellisia suhteita, joita voitaisiin pitää mahdollisena eturistiriitana.
kiitokset
kiitämme tutkimukseen osallistuneita potilaita. Kiitämme Tri. Jiong Gao (BGI Genomics, BGI-Shenzhen, Shenzhen 518083, Kiina) avustaan tämän käsikirjoituksen valmistelun aikana.
Täydennysaineisto
tämän artikkelin Täydennysaineisto löytyy verkosta osoitteesta: https://www.frontiersin.org/articles/10.3389/fgene.2019.00817/full#supplementary-material
Kuva S1 / analyyttinen strategia todennäköisen patogeenisen variantin tunnistamiseksi WES: n avulla.
Kuva S2 / luovuttajan liitospaikat netgene2: n ja Nnsplicen ennustamana.
taulukko S1 / varianttien luettelo seulomalla varianttien taajuudet, mutaatiotilanne ja periytymistila.
taulukko S2 / luettelo hydrokefalukseen liittyvistä geeneistä (HP:0000238: n vienti).
Du, Y. Z., Dickerson, C., Aylsworth, A. S., Schwartz, C. E. (1998). Hiljainen mutaatio, C924T (G308G), l1cam-geenissä aiheuttaa X-linkitetyn vesipään (HSAS). J. Med. Genet. 35 (6), 456–462. doi: 10.1136 / jmg.35.6.456
PubMed Abstract | CrossRef Full Text | Google Scholar
Itoh, K., Fushiki, S. (2015). Rooli L1cam hiiren kortikogeneesi,ja patogeneesi vesipää. Pathol. Int. 65 (2), 58–66. doi: 10.1111/pin.12245
PubMed Abstract | CrossRef Full Text | Google Scholar
Li, H., Durbin, R. (2009). Nopea ja tarkka lyhyt lukusuuntaus Burrows-Wheeler-muunnoksella. Bioinformatiikka 25 (14), 1754-1760. doi: 10.1093 / bioinformatiikka / btp324
PubMed Abstract | CrossRef Full Text | Google Scholar
Liu, H., Focia, P. J., He, X. (2011). Neurofasciinin homofiilinen adheesiomekanismi, joka kuuluu hermosolujen adheesiomolekyylien L1-perheeseen. J. Biol. Kemiaa. 286 (1), 797–805. doi: 10.1074 / jbc.M110.180281
PubMed Abstract | CrossRef Full Text | Google Scholar
McKenna, A., Hanna, M., Banks, E., Sivatšenko, A., Cibulskis, K., Kernytsky, A., et al. (2010). The Genome Analysis Toolkit: a MapReduce framework for analysing next-generation DNA sequencing data. Genome Res. 20 (9), 1297-1303. doi: 10.1101 / gr.107524.110
PubMed Abstract | CrossRef Full Text | Google Scholar
Richards, S., Aziz, N., Bale, S., Bick, D., Das, S., Gaster-Foster, J., et al. (2015). Standards and guidelines for the interpretation of sequence variants: a joint consensus recommendation of the American College of Medical Genetics and Genomics and the Association for Molecular Pathology. Genet. Med. 17 (5), 405–424. doi: 10.1038 / gim.2015.30
PubMed Abstract | CrossRef Full Text | Google Scholar
Vos, Y. J., de Walle, H. E. K., Bos, K. K., Stegeman, J. A., Berge, A. M., Bruining, M., et al. (2010). Genotype-phenotype correlations in L1 syndrome: a guide for genetic counciling and mutation analysis. J. Med. Genet. 47 (3), 169–175. doi: 10.1136 / jmg.2009.071688
PubMed Abstract | CrossRef Full Text | Google Scholar
Wei, C. H., Ryu, S. E. (2012). Solun adheesiomolekyylien L1-perheen homofiilinen vuorovaikutus. Käyt.viim. Mol. Med. 44 (7), 413–423. doi: 10,3858 / emm.2012.44.7.050
PubMed Abstract | CrossRef Full Text | Google Scholar
Weller, S., Gartner, J. (2001). Geneettiset ja kliiniset näkökohdat x-linkitetty vesipää (L1 tauti): mutaatiot L1CAM geeni. Hum. Mutat. 18 (1), 1–12. doi: 10.1002 / humu.1144
PubMed Abstract | CrossRef Full Text | Google Scholar
Zhao, X., Yip, P. M., Siu, C. H. (1998). Identification of a homophilic binding site in immunoglobulin-like domain 2 of the cell adhesion molecule L1. J. Neurochem. 71 (3), 960–971 doi: 10.1046/j.1471-4159.1998.71030960.x.
PubMed Abstract | CrossRef Full Text | Google Scholar