- Introducere
- prezentarea cazului
- materiale și metode
- cariotipul și matricea SNP
- secvențierea întregului exom
- extracția ARN, PCR și secvențierea
- rezultate
- discuție
- disponibilitatea datelor
- declarație de etică
- contribuțiile autorului
- finanțare
- Declarație privind conflictul de interese
- mulțumiri
- material suplimentar
Introducere
gena moleculei de adeziune a celulelor L1 (L1CAM) este o moleculă de adeziune a celulelor neuronale aparținând superfamiliei imunoglobulinei; posedă funcții cheie în dezvoltarea sistemului nervos (Itoh și Fushiki, 2015). Mutațiile din L1CAM au fost legate de sindroamele neurologice legate de X, care sunt rezumate ca boli L1. Acestea sunt clasificate după cum urmează: Hidrocefalie legată de X (XLH) datorită stenozei apeductului Sylvius (HSAS), sindromului MASA (dizabilitate intelectuală, afazie, mers amestecat, degete aductibile), parapareză spastică de tip 1 (SP1) și ageneză legată de X corpus callosum (ACC) (Weller și Gartner, 2001; Itoh și Fushiki, 2015).
aproximativ 282 de mutații cauzatoare de boală (DMs) în gena L1CAM au fost raportate în hgmd XV professional 2019.2 (https://portal.biobase-international.com/hgmd/pro/all.php). Modificările genei L1CAM sunt variate; analizele datelor privind mutațiile de la 282 de pacienți au evidențiat 51% mutații missense și nonsens, 25% ștergeri, 5% inserții și 19% Modificări ale locului de îmbinare, dar mutațiile silențioase în L1CAM cu potențial patogen au fost rare, iar mutațiile silențioase au fost adesea ignorate în special în detectarea secvențierii întregului exom (WES).
în acest studiu, folosind WES, am analizat ADN-ul fetal al unei femei însărcinate chineze care a raportat cinci sarcini continue cu hidrocefalie fetală; am găsit doar o nouă mutație tăcută c.453g > T (P.Gly151 = ) în gena L1CAM. Interesant, printr-o analiză suplimentară, am indicat mutația silențioasă a creat o potențială secvență de consens a site-ului de îmbinare 5′, ceea ce ar duce la o ștergere în cadru a 72 bp din exonul 5 și 24 aminoacizi ai proteinei L1CAM.
prezentarea cazului
o femeie sănătoasă de 28 de ani a fost trimisă la clinica noastră după patru întreruperi voluntare ale sarcinii din cauza hidrocefaliei fetale la alte spitale. Toți fetușii erau bărbați. Când a ajuns la spitalul nostru (Spitalul de femei, școala de Medicină, Universitatea Zhejiang, Zhejiang, China), ea era deja la a cincea sarcină la 24 de săptămâni de gestație, cu un hidrocefalie fetală prin examinări de imagine. Pentru a explora cauza genetică, prelevarea de sânge fetal a fost efectuată la 26 de săptămâni de vârstă gestațională. Studiile citogenetice convenționale au fost efectuate atât pentru probele fetale, cât și pentru cele parentale, iar proba fetală a fost analizată în continuare prin matrice de polimorfism cu un singur nucleotid (SNP) și Wes.
acest studiu a fost realizat în conformitate cu recomandările Comitetului de etică al Spitalului de femei, școala de Medicină Universitatea Zhejiang, iar consimțământul informat a fost obținut de la toți participanții la acest studiu în conformitate cu declarația de la Helsinki. Protocolul de studiu a fost aprobat de Comisia de revizuire a Spitalului de femei, școala de Medicină, Universitatea Zhejiang din China.
materiale și metode
cariotipul și matricea SNP
cariotipurile sângelui din cordonul ombilical fetal și ale sângelui din cordonul ombilical periferic au fost determinate prin cariotiparea convențională a cel puțin 30 de limfocite din sânge, care au fost oprite la metafază de colchicină. Cariotipurile de bandă G ale celulelor cultivate au fost efectuate la nivelul benzii 320-400 cu o rezoluție de aproximativ 10 Mb. SNP array a fost realizat de cytoscan HD array (Affymetrix, SUA) conform instrucțiunilor producătorului, cu aproximativ 2.600.000 de markeri, inclusiv 750.000 de sonde SNP și 1.900.000 de sonde non-polimorfice pentru o acoperire completă a genomului întreg. Datele au fost analizate de software-ul cromozomial Analysis Suite (ChAS) (Affymetrix, Santa Clara, CA) pe baza ansamblului GRCh37/hg19. Pragul de raportare al rezultatului numărului de exemplare a fost stabilit la 500 kb cu un număr de Marcatori de 50 la sută pentru câștiguri și la 200 kb cu un număr de Marcatori de 50 la sută pentru pierderi.
secvențierea întregului exom
partea principală a WES a fost furnizată de Institutul de Genomică din Beijing. ADN-ul Genomic a fost extras de un kit de sânge DNeasy (Qiagen, CA) și apoi a fost fragmentat de Covaris LE220 (Massachusetts, SUA) pentru a genera o bibliotecă pereche (200-250 bp). Toate bibliotecile amplificate au fost efectuate pe platforma BGISEQ-500, ADN-ul cu o singură catenă a fost amestecat cu kitul de pregătire a Bibliotecii ADN mgieasy v1 (BGI, Shenzhen, China) și apoi secvențiat folosind chimia 100SR cu kitul de secvențiere cu randament ridicat BGISEQ-500rs (BGI, Shenzhen, China).
citirile curate (cu o lungime de 90 bp) derivate din secvențierea și filtrarea vizate au fost apoi aliniate la referința genomului uman (hg19) folosind pachetul software multi-Vision Burrows-Wheeler Aligner (BWA) (Li și Durbin, 2009). După aliniere, fișierele de ieșire au fost utilizate pentru a efectua acoperirea secvențială și analiza aprofundată a regiunii țintă, variantele cu un singur nucleotid (SNVs) și apelarea indel, am folosit software-ul GATK pentru a detecta SNVs și indels (McKenna și colab., 2010), toate SNV-urile și Indel-urile au fost filtrate și estimate prin mai multe baze de date, inclusiv Centrul Național pentru Informații Biotehnologice (NCBI) baza de date cu polimorfism cu un singur nucleotid (DBSNP), HapMap, 1000 genomes Project set de date și baza de date a 100 de adulți sănătoși chinezi. Am folosit Condel, SIFT, PolyPhen-2, LRT, Mutation Taster și PhyloP pentru a prezice efectul variantelor. Variantele patogene sunt evaluate în conformitate cu protocolul emis de Colegiul American de Genetică Medicală și genomică (ACMG) (Richards și colab., 2015). Baza de date a mutațiilor genetice umane (Hgmd) a fost utilizată pentru a examina mutațiile. Toate variantele potențiale patogene au fost validate folosind metode de secvențiere Sanger.
extracția ARN, PCR și secvențierea
celulele mononucleare din sângele periferic (Pmbc) și celulele mononucleare din sângele din cordonul ombilical (Cbmc) au fost izolate prin separarea gradientului densității Ficoll. ARN-ul Total a fost extras din Pmbc-uri și Cbmc-uri folosind TRIzol (Takara, Japonia). ARN-urile totale extrase au fost transcrise invers folosind RT Kit (Takara, Japonia). PCR a fost realizat folosind GoldStar Best Master Mix (Cwbio, Beijing). Secvențele de grund sunt enumerate: L1CAM-ADN-5F, CCCACCCGTCCTTTCCTA; L1CAM-ADN-5R, CGCTCGTCCTGCTTGATGT; L1CAM-ARNm-4-6-F, GGTGTCCACTTCAAACCCAA; și l1cam-ARNm-4-6-R, GCGGCTTCCTGTCAATCA. Secvențierea Sanger a fost efectuată de un analizor de ADN ABI 3130.
rezultate
o femeie sănătoasă de 28 de ani a fost trimisă la clinica noastră după patru întreruperi voluntare ale sarcinii din cauza hidrocefaliei fetale. Toți fetușii erau bărbați (figura 1A). Pedigree-ul familial părea să arate XLH. Era deja aici a cincea sarcină la 26 de săptămâni de gestație. Ventriculomegalia fetală a fost detectată prin ecografie fetală și RMN, care a demonstrat în mod constant prezența hidrocefaliei. Ei au arătat că ventriculul cerebral bilateral și cel de-al treilea ventricul au fost evident dilatate și a existat hidrocefalie severă în intracerebral și ageneză a corpului calos (figura 1B).
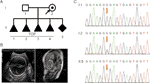
Figura 1 (A) Pedigree-ul familiei. Sus, întreruperea sarcinii. (B) examinări imagistice ale fătului. Ecografia fetală și RMN-ul fetal au arătat că există hidrocefalie severă la făt. (C) analiza secvenței ADN-ului genomic de la membrii familiei. Genotipurile L1CAM au fost de tip sălbatic, c. 453G > T Het și C. 453G > T Hom, în I:1 (soț), I:2 (femeie însărcinată) și II:5 (făt). Mutația este indicată de săgețile roșii.
pentru a explora posibila cauză genetică, am efectuat analiza cariotipului și matricea SNP pentru a analiza prelevarea de sânge fetal și nu am găsit rezultate pozitive. Rezultatul neovascularizării coroidiene (CNV) a fost depus în expresia genică Omnibus (GEO); numărul de aderare este GSE133063, așa cum este anexat mai jos (https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE133063).
apoi, am detectat fătul de către WES. Strategia analitică de identificare a variantelor patogene probabile a fost prezentată în figura S1. O listă de variante (tabelul S1) au fost obținute prin screening-ul frecvențelor variantei, starea mutației și modul de moștenire. Luând în considerare genele asociate hidrocefaliei (HP:0000238, http://compbio.charite.de/hpoweb/showterm?id=HP:0000238#id=HP_0000238) (tabelul S2), nu a existat nicio mutație notabilă suplimentară, cu excepția mutației silențioase A c.453g > T în exonul 5 al genei L1CAM (NM_000425.3). c. 453G > T nu a fost raportat în HGMD și ClinVar și nu a fost găsit în dbsnp, gnomAD și alte seturi de date. În conformitate cu standardele și liniile directoare ale ACMG (Richards și colab., 2015), nu a atins încă criteriul „patogen” sau „probabil patogen”, dar nu au existat alte mutații potențiale; nu am avut de ales decât să facem o analiză suplimentară a mutației silențioase găsite.
conform gândirii tradiționale, această substituție de bază a avut loc în a treia bază din codonul 151, care codifică o glicină, creând astfel o mutație neutră (P.Gly151 = ). Această variantă a fost confirmată în ADN extras din sângele din cordonul ombilical fetal și din sângele periferic în cuplu prin secvențierea Sanger (figura 1C). Femeia purta mutația heterozigotă, iar soțul ei era un genotip de tip sălbatic.
cu mutație degustător (http://www.mutationtaster.org/), c.453G > T a fost marcat ca „boala cauzatoare.”A arătat că caracteristicile proteinelor ar putea fi afectate și site-ul de îmbinare ar putea fi schimbat; am fost curioși cu privire la potențialele efecte de îmbinare ale funcției L1CAM în această mutație silențioasă. Mutația silențioasă a fost testată folosind următoarele produse software online: NetGene2 (http://www.cbs.dtu.dk/services/NetGene2/) și NNSplice (http://www.fruitfly.org/seq_tools/splice.html); site-ul potențial de îmbinare 5′ a fost, de asemenea, prevăzut să fie creat în l1cam c.453g > mutația T utilizând produsele software (figura s2). Rezultatele au arătat că această mutație silențioasă a creat un potențial situs de îmbinare 5′ 72 bp în amonte de situsul normal de îmbinare exon 6/intron 6 (Figura 2a). Dacă acesta este cazul, putem găsi schimbarea lungimii ARN-ului mesager L1CAM (ARNm) între I:2 și II:5 (Figura 2a). RT-PCR a fost realizat folosind primeri concepuți pentru a amplifica exonii 4-6 în ARNm L1CAM. Într-adevăr, rezultatele au arătat o bandă scurtă de trunchiere în PCR ADNc fetal (II: 5), în timp ce banda amplificată din ARNm L1CAM conținea banda lungă așteptată în PCR ADNc soț (I:1) și benzi lungi/scurte la femeia gravidă ADNc PCR (I:2) (Figura 2b). Secvențierea directă a fragmentului amplificat a arătat că ștergerea a implicat ultimele 72 bp de exon 5 în ADNc fetal masculin (femeia a fost purtătoare) (figura 2c). Avem dovezile patogene cruciale.
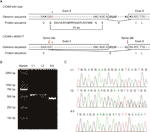
Figura 2 (a) Reprezentarea schematică a organizației exon 5, intron 6 și exon 6 în l1cam. (B) analiza RT-PCR a exonilor 5 și 6 ai ADNc L1CAM din celulele mononucleare din sângele periferic (Pmbc) și celulele mononucleare din sângele din cordonul ombilical (Cbmc). Electroforeza în gel de agaroză a produselor RT-PCR generate de I: 1 (soț), I:2 (femeie însărcinată) și II:5 (făt). (C) analiza secvențială a produsului RT-PCR din Pmbc-urile cuplului și Cbmc-urile fătului.
această mutație silențioasă a dus la 24 de aminoacizi ai proteinei L1CAM (reziduuri 151-174); Lys (K) a fost înlocuit cu Glu (E) la codonul 175 (figura 2a). Au existat alinierea mai multor secvențe de proteine L1CAM pe mai multe specii și conservarea aminoacizilor lipsă din l1cam la mamifere: Homo sapiens, Pan troglodytes, Bos taurus, mus musculus și Rattus norvegicus (figura 3A). Tipul sălbatic și C.453g > proteinele t splicing mutation l1cam au fost prezise de serverul software CPHmodels-3.2 (http://www.cbs.dtu.dk/services/CPHmodels/) (figura 3b). Domeniul 2 de tip imunoglobulină (Ig-like) (reziduuri 134-230) al mutației de tip sălbatic și de îmbinare a proteinelor l1cam este prezentat în figura 3C. L1CAM c. 453g > t mutația de îmbinare a modificat structura proteinei ,în special domeniul Ig-like 2
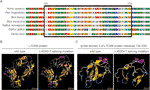
Figura 3 (a) alinierea mai multor secvențe de proteine L1CAM între specii. L1CAM c.453G > T a dus la lipsa a 24 de aminoacizi ai proteinei L1CAM (reziduuri 151-174) în regiunea aminoacizilor conservați la diferite specii. Coloana neagră arată aminoacizii lipsă. (B) structurile de tip sălbatic și c.453g > t splicing proteina mutație l1cam așa cum a prezis de software-ul CPHmodels-3.2 Server. (C) structurile domeniului 2 de tip imunoglobulină (IG-like) (reziduuri 134-230) ale proteinei l1cam de tip sălbatic și mutație de îmbinare.
discuție
mutațiile silențioase au fost adesea detectate de WES, dar s-a acordat o atenție insuficientă, ceea ce a dus la omiterea DMs. În acest studiu, l-am angajat pe WES pentru a explora cauza genetică a unei familii chineze cu hidrocefalie, dar am găsit doar o nouă mutație tăcută în L1CAM, care ne-a forțat să facem o analiză suplimentară. Din fericire, am dovedit că mutația silențioasă a creat un nou site de îmbinare 5′ și a fost un DM.
mutațiile L1CAM pot provoca o boală L1 legată de X, dar simptomele clinice sunt variabile; mutațiile produc fenotipuri neașteptate. În cadrul studiului, cei cinci fetuși suferinzi sunt toți bărbați, ceea ce este în concordanță cu un model de moștenire. Ecografia fetală și RMN prezintă o boală tipică L1, incluzând XLH și ageneza corpului calos. Îmbunătățește înțelegerea noastră asupra corelației genotip–fenotip a L1CAM.
L1CAM c.453g > T (P.Gly151 = ) s-a crezut inițial că nu are niciun efect asupra secvenței proteice. Dar alte mutații silențioase, c. 924C > T (P.Gly308 = ) și C.645c > T (p.Gly215 = ), în gena L1cam au fost raportate a fi DMs (Du și colab., 1998; Vos și colab., 2010). C.924c > mutația T a dus la activarea unui nou situs de îmbinare 69 bp 5′ la situsul normal de îmbinare a donatorului exon 8/intron 8 și a fost declarat ca un situs „cauzator de boli” pentru hidrocefalie (Du și colab., 1998). Pentru C. 645C > T în L1CAM, 51 bp a fost șters odată cu activarea unei noi îmbinări donatoare exon 6/intron 6 (Vos și colab., 2010). Studiul nostru de față a fost similar; mutația c.453g > T a creat un potențial situs de îmbinare 5′ în amonte de situsul normal de îmbinare exon 5/intron 5. Toate aceste mutații silențioase au creat noi site-uri de îmbinare a donatorilor, rezultând că exonul a fost omis. Ne-a amintit să acordăm o atenție deosebită acestor mutații tăcute, care pot afecta îmbinarea proteinelor.
ca glicoproteină transmembranară și membru al superfamiliei imunoglobulinelor moleculelor de adeziune celulară, proteina L1CAM poate interacționa la suprafața celulei cu un număr de glicoproteine diferite, iar legarea homofilă este probabil principalul său mod de interacțiune (Wei și Ryu, 2012). Studiile privind structura cristalină a domeniilor asemănătoare Ig 1-4 Din neurofascină au sugerat că multe mutații patologice L1 afectează reziduurile de aminoacizi conservate din aceste domenii și interferează cu interacțiunile homofile (Liu și colab., 2011), mai ales după cum a fost verificat de cercetarea funcțiilor domeniului 2 asemănător Ig (Zhao și colab., 1998). În studiul nostru, am speculat că L1CAM c. 453g > t a modificat domeniul Ig-like 2 în partea extracelulară a proteinei L1CAM, ducând la interacțiunea extracelulară anormală, nereușind să înceapă inițierea în aval a căii de semnalizare. Un studiu suplimentar dedicat spectrometriei de masă a acestei variante L1CAM ar clarifica în mod specific ce ansamblu molecular este produs în celulă.
în rezumat, prin WES, am raportat o nouă mutație silențioasă c.453g > T în L1CAM care produce un site de îmbinare de 5′ responsabil pentru hidrocefalie. S-a prezis că această variantă proteică anormală va modifica domeniul 2 asemănător Ig, ceea ce ar putea afecta legarea homofilă a proteinei L1CAM. În plus, am efectuat diagnosticul genetic prenatal pentru femeia gravidă care a raportat cinci sarcini continue cu hidrocefalie. Între timp, a sugerat că unele mutații silențioase detectate în WES nu ar trebui ignorate; predicțiile de îmbinare ale acestor mutații erau necesare. Acesta a oferit o nouă bază genetică pentru diagnosticul prenatal și diagnosticul prenatal pre-implantare de hidrocefalie.
disponibilitatea datelor
seturile de date disponibile publicului au fost analizate în acest studiu. Aceste date pot fi găsite aici: GSE133063(https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE133063).
declarație de etică
studiile care au implicat participanți umani au fost revizuite și aprobate de Comisia de revizuire a Spitalului de femei, școala de Medicină, Universitatea Zhejiang din China. Consimțământul scris în cunoștință de cauză de a participa la acest studiu a fost oferit de tutorele legal/rudele apropiate ale participanților. Consimțământul scris în cunoștință de cauză a fost obținut de la persoana(persoanele) și de la tutorele legal al minorului(minorilor) pentru publicarea oricăror imagini sau date potențial identificabile incluse în acest articol.
contribuțiile autorului
YS, YLi, MC, YLu, YQ și YY au efectuat experimente. YS pregătit cifrele. MC și YLi au analizat datele WES. YLu și YQ au efectuat analiza cariotipului și matricea SNP. YY recrutat probe. HL și FL au furnizat examinări imagistice. YS și MD au scris manuscrisul. Toți autorii au citit și aprobat manuscrisul final.
finanțare
acest studiu a fost susținut de Fundația Națională de științe Naturale din China (grantul nr.81801441), Programul cheie de cercetare și dezvoltare al provinciei Zhejiang (grantul nr. 2019c03025), Programul Național de cercetare și dezvoltare cheie al Chinei (Grant No. 2016yfc1000703) și Fundația de cercetare științifică Medicală din provincia Zhejiang (Grant No.2014kya246).
Declarație privind conflictul de interese
autorii declară că cercetarea a fost realizată în absența oricăror relații comerciale sau financiare care ar putea fi interpretate ca un potențial conflict de interese.
mulțumiri
mulțumim pacienților înscriși în această cercetare. Îi mulțumim Dr. Jiong Gao (BGI Genomics, BGI-Shenzhen, Shenzhen 518083, China) pentru asistența sa în timpul pregătirii acestui manuscris.
material suplimentar
materialul suplimentar pentru acest articol poate fi găsit online la: https://www.frontiersin.org/articles/10.3389/fgene.2019.00817/full#supplementary-material
figura S1 / strategia analitică pentru identificarea probabilei variante patogene de către WES.
figura S2 | Site-urile de îmbinare donatoare prezise de NetGene2 și NNSplice.
tabelul S1 | lista variantelor prin screening-ul frecvențelor variantelor, starea mutației și modul de moștenire.
tabelul S2 | lista genelor asociate hidrocefaliei (Export pentru HP:0000238).
Du, Y. Z., Dickerson, C., Aylsworth, A. S., Schwartz, C. E. (1998). O mutație silențioasă, C924T (G308G), în gena L1CAM are ca rezultat hidrocefalie legată de X (HSAS). J. Med. Genet. 35 (6), 456–462. doi: 10.1136/jmg.35.6.456
Rezumat PubMed / CrossRef Text Complet / Google Scholar
Itoh, K., Fushiki, S. (2015). Rolul L1cam în corticogeneza murină și patogeneza hidrocefaliei. Pathol. Int. 65 (2), 58–66. doi: 10.1111/bolț.12245
Rezumat PubMed / CrossRef Text Complet / Google Scholar
Li, H., Durbin, R. (2009). Aliniere rapidă și precisă de citire scurtă cu transformarea Burrows-Wheeler. Bioinformatica 25 (14), 1754-1760. doi: 10.1093 / Bioinformatica / btp324
PubMed rezumat / CrossRef textul integral / Google Scholar
Liu, H., Focia, P. J., El, X. (2011). Mecanismul de adeziune homofilă al neurofascinei, membru al familiei L1 de molecule de adeziune a celulelor neuronale. J. Biol. Chem. 286 (1), 797–805. doi: 10.1074 / jbc.M110.180281
rezumat PubMed / CrossRef Text Complet / Google Scholar
McKenna, A., Hanna, M., bănci, E., Sivachenko, A., Cibulskis, K., Kernytsky, A. și colab. (2010). Setul de instrumente de analiză a genomului: un cadru MapReduce pentru analiza datelor de secvențiere a ADN-ului de generație următoare. Genomul Res. 20 (9), 1297-1303. doi: 10.1101 / gr.107524.110
rezumat PubMed / CrossRef Text Complet / Google Scholar
Richards, S., Aziz, N., Bale, S., Bick, D., Das, S., Gastier-Foster, J. și colab. (2015). Standarde și orientări pentru interpretarea variantelor de secvență: o recomandare comună de consens a Colegiului American de Genetică Medicală și genomică și a Asociației pentru Patologie Moleculară. Genet. Med. 17 (5), 405–424. doi: 10.1038 / gim.2015.30
rezumat PubMed / CrossRef Text Complet / Google Scholar
Vos, Y. J., de Walle, H. E. K., Bos, K. K., Stegeman, J. A., Berge, A. M., Bruining, M. și colab. (2010). Corelațiile genotip-fenotip în sindromul L1: un ghid pentru consilierea genetică și analiza mutațiilor. J. Med. Genet. 47 (3), 169–175. doi: 10.1136 / jmg.2009.071688
Rezumat PubMed / CrossRef Text Complet / Google Scholar
Wei, C. H., Ryu, S. E. (2012). Interacțiunea homofilă a familiei L1 de molecule de adeziune celulară. Exp. Mol. Med. 44 (7), 413–423. doi: 10.3858 / emm.2012.44.7.050
PubMed Rezumat / CrossRef Text Integral / Google Scholar
Weller, S., Gartner, J. (2001). Aspecte genetice și clinice ale hidrocefaliei legate de X (boala L1): mutații ale genei L1CAM. Zumzet. Mutatul. 18 (1), 1–12. doi: 10.1002 / humu.1144
PubMed Abstract | CrossRef Full Text | Google Scholar
Zhao, X., Yip, P. M., Siu, C. H. (1998). Identification of a homophilic binding site in immunoglobulin-like domain 2 of the cell adhesion molecule L1. J. Neurochem. 71 (3), 960–971 doi: 10.1046/j.1471-4159.1998.71030960.x.
PubMed Abstract | CrossRef Full Text | Google Scholar