Variationen von Kinetoplastennetzwerken wurden ebenfalls beobachtet und werden durch die Anordnung und Position ihrer kDNA beschrieben.
- Ein Pro-kDNA-Kinetoplast ist eine bündelartige Struktur, die in der Mitochondrienmatrix proximal zum flagellären Basalkörper gefunden wird. Im Gegensatz zum herkömmlichen kDNA-Netzwerk enthält ein Pro-kDNA-Kinetoplast sehr wenig Verkettung und seine Maxicircles und Minicircles sind entspannt statt supercoiled. Pro-kDNA wurde bei Bodo saltans, Bodo designis, Procryptobia sorokini syn. Bodo sorokini, Rhynchomonas nasuta und Cephalothamnium cyclopi.
- Ein Poly-kDNA-Kinetoplast ähnelt in seiner kDNA-Struktur einem Pro-kDNA-Kinetoplast. Es enthält wenig catenation und kein Supercoiling. Die Besonderheit von Poly-kDNA besteht darin, dass die Poly-kDNA nicht wie bei Pro-kDNA aus einem einzigen kugelförmigen Bündel besteht, sondern auf verschiedene diskrete Herde im gesamten Mitochondrienlumen verteilt ist. Poly-kDNA wurde in Dimastigella trypaniformis (ein Kommensal im Darm einer Termite), Dimastigella mimosa (ein frei lebendes Kinetoplastid) und Cruzella marina (ein Parasit des Darms einer Seespritze) beobachtet.
- Ein Pan-kDNA-Kinetoplast, wie Poly-kDNA und Pro-kDNA, enthält einen geringeren Grad an Verkettung, aber es enthält Minicircles, die supercoiled sind. Pan-kDNA-Kinetoplasten füllen den größten Teil der Mitochondrienmatrix und sind nicht auf diskrete Herde wie Poly-kDNA beschränkt. Pan-kDNA wurde bei Cryptobia helicis (einem Parasiten des Receptaculum seminis von Schnecken), Bodo caudatus und Cryptobia branchialis (einem Parasiten von Fischen) beobachtet.
- Ein Mega-kDNA-Kinetoplast ist ziemlich gleichmäßig in der Mitochondrienmatrix verteilt, enthält jedoch keine Minicircles. Stattdessen werden Sequenzen von kDNA, die in der Sequenz anderen Kinetoplast-Minicircles ähnlich sind, im Tandem zu größeren Molekülen von etwa 200 kb Länge verbunden. Mega-kDNA (oder Mega-kDNA ähnliche Strukturen) wurden bei Trypanoplasma borreli (einem Fischparasiten) und Jarrellia sp. (ein Walparasit).
Das Vorhandensein dieser Vielfalt von kDNA-Strukturen verstärkt die evolutionäre Beziehung zwischen den Arten von Kinetoplastiden. Da Pan-kDNA am ehesten einem DNA-Plasmid ähnelt, kann es sich um die angestammte Form von kDNA handeln.
Replikationbearbeiten

Die Replikation des Kinetoplasten erfolgt gleichzeitig mit der Duplikation des benachbarten Flagellums und unmittelbar vor der nuklearen DNA-Replikation. In einem traditionellen Crithidia fasciculata kDNA-Netzwerk wird die Initiierung der Replikation durch die Verknüpfung von kDNA-Minicircles über Topoisomerase II gefördert. Die freien minicircles werden in eine Region zwischen dem Kinetoplast und der mitochondrialen Membran freigegeben, die kinetoflagellar Zone (KFZ) genannt wird. Nach der Replikation wandern die Minicircles durch unbekannte Mechanismen zu den antipodischen Proteinkomplexen, die mehrere Replikationsproteine enthalten, einschließlich einer Endonuklease, Helikase, DNA-Polymerase, DNA-Primase und DNA-Ligase, die die Reparatur verbleibender Diskontinuitäten in den neu replizierten Minicircles initiieren.
Dieser Vorgang findet jeweils in einem Minicircle statt, und nur eine kleine Anzahl von Minicircles wird zu einem bestimmten Zeitpunkt nicht verknüpft. Um zu verfolgen, welche Minicircles repliziert wurden, verbleibt beim Wiedereintritt in das kDNA-Netzwerk eine kleine Lücke in den entstehenden Minicircles, die sie als bereits repliziert identifiziert. Minicircles, die noch nicht repliziert wurden, sind immer noch kovalent geschlossen. Unmittelbar nach der Replikation wird jeder Nachkomme an das kDNA-Netzwerk proximal der antipodalen Proteinkomplexe gebunden und die Lücken werden teilweise repariert.
Wenn die Minicircle-Replikation fortschreitet, dreht sich das gesamte kDNA-Netzwerk um die zentrale Achse der Festplatte, um den Aufbau neuer Minicircles zu verhindern. Es wird angenommen, dass die Rotation direkt mit der Replikation des benachbarten Flagellums verbunden ist, da sich der Tochterbasalkörper auch um den Mutterbasalkörper in einem Timing und einer ähnlichen Weise wie die Rotation des Kinetoplasten dreht. Durch Drehen werden die Minicircles des Tochterkinetoplast spiralförmig zusammengesetzt und beginnen sich nach innen in Richtung der Mitte der Scheibe zu bewegen, wenn neue Minicircles entkoppelt und zur Replikation in das KFZ bewegt werden.
Während die genauen Mechanismen für Maxicircle kDNA noch im selben Detail wie für Minicircle kDNA bestimmt werden müssen, wird eine Struktur namens Nabelschnur (deutsch für „Nabelschnur“) beobachtet, die die Tochter-kDNA-Netzwerke anbindet, aber schließlich während der Trennung bricht. Unter Verwendung der Fischsonden, zum des Nabelschnurziels, ist es gefunden worden, um maxicircle kDNA zu enthalten.
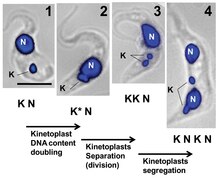
Die Kinetoplastenreplikation wird als in fünf Stufen auftretend beschrieben, jeweils in Bezug auf die Replikation des benachbarten Flagellums.
- Stufe I: Der Kinetoplast hat die Replikation noch nicht initiiert, enthält keine antipodischen Proteinkomplexe und ist relativ zu einem einzelnen flagellären Basalkörper positioniert.
- Stadium II: Der Kinetoplast beginnt antipodische Proteinkomplexe zu zeigen. Der flagelläre Basalkörper beginnt ebenso wie der Kinetoplast mit der Replikation. Die Assoziation des replizierenden Kinetoplasten mit den beiden Basalkörpern bewirkt, dass er ein gewölbtes Aussehen entwickelt.
- Stadium III: Das neue Flagellum beginnt sich zu trennen und der Kinetoplast nimmt eine zweilappige Form an.
- Stufe IV: Die Kinetoplasten erscheinen als separate Scheiben, bleiben aber durch die Nabelschnur verbunden.
- Stadium V: Die Tochterkinetoplasten sind vollständig getrennt, da die Nabelschnur gebrochen ist. Ihre Struktur ist identisch mit der in Stufe I.