zaobserwowano również odmiany sieci kinetoplast i są one opisane przez rozmieszczenie i lokalizację ich kDNA.
- kinetoplast pro-kDNA jest strukturą podobną do wiązki znajdującą się w matrycy mitochondrialnej bliższej do flagellar basal body. W przeciwieństwie do konwencjonalnej sieci KDNA, kinetoplast pro-KDNA zawiera bardzo mało katenacji, a jego maxicircles i minicircles są zrelaksowane zamiast supercoiled. Pro-kDNA zaobserwowano u Bodo saltans, Bodo designis, Procryptobia sorokini syn. Bodo sorokini, Rhynchomonas nasuta i Cephalothamnium cyclopi.
- kinetoplast Poli-kDNA jest podobny w strukturze kDNA do kinetoplastu pro-kDNA. Zawiera niewiele katenacji i nie supercoiling. Charakterystyczną cechą poly-kDNA jest to, że zamiast składać się z pojedynczego wiązki kulistej, jak w pro-KDNA, poly-KDNA jest rozprowadzany wśród różnych dyskretnych ognisk w całym świetle mitochondrialnym. Poli-kDNA zaobserwowano u Dimastigella trypaniformis (komensalu w jelicie Termita), Dismastigella mimosa (wolnożyjącego kinetoplastyda) i Cruzella marina (pasożyta jelita morskiego).
- kinetoplast pan-kDNA, podobnie jak Poli-kDNA i pro-KDNA, zawiera mniejszy stopień katenacji, ale zawiera minicircles, które są supercoiled. Kinetoplasty Pan-kDNA wypełniają większość macierzy mitochondrialnej i nie ograniczają się do dyskretnych ognisk takich jak Poli-KDNA. Pan-kDNA zaobserwowano u Cryptobia helicis (pasożyt receptaculum seminis ślimaków), Bodo caudatus i Cryptobia branchialis (pasożyt ryb).
- kinetoplast mega-kDNA jest rozmieszczony dość równomiernie w matrycy mitochondrialnej, ale nie zawiera minikomórek. Zamiast tego, sekwencje KDNA podobne w sekwencji do innych kinetoplastów są połączone tandemowo w większe cząsteczki o długości około 200KB. Mega-kDNA (lub struktury podobne do mega-KDNA) obserwowano u Trypanoplasme borreli (pasożyta ryb) i Jarrellia sp. (pasożyt wieloryba).
obecność tej odmiany struktur kDNA wzmacnia ewolucyjne pokrewieństwo między gatunkami kinetoplastydów. Ponieważ pan-KDNA najbardziej przypomina plazmid DNA, może być przodkową formą kDNA.
Replikacjedytuj

replikacja kinetoplastu zachodzi równocześnie z duplikacją sąsiedniego wiciowca i tuż przed replikacją jądrowego DNA. W tradycyjnej sieci KDNA crithidia fasciculata inicjacja replikacji jest promowana przez rozłączanie miniklingów kDNA przez topoizomerazę II. Wolne minikulatury uwalniane są do obszaru między kinetoplastem a błoną mitochondrialną zwanego strefą kinetoflagellarną (KFZ). Po replikacji minikulki migrują przez nieznane mechanizmy do antypodalnych kompleksów białkowych, które zawierają kilka białek replikacyjnych, w tym endonukleazę, helikazę, polimerazę DNA, prymazę DNA i ligazę DNA, które inicjują naprawę pozostałych nieciągłości w nowo replikowanych minikulkach.
ten proces odbywa się jeden minicrcle na raz, a tylko niewielka liczba minicrcle są odłączone w danym momencie. Aby śledzić, które minicircles zostały zreplikowane, po ponownym dołączeniu do sieci kDNA pozostaje niewielka luka w powstających minicircles, która identyfikuje je jako już zreplikowane. Minicircles, które nie zostały jeszcze zreplikowane, są nadal kowalencyjnie zamknięte. Natychmiast po replikacji każde potomstwo jest przyłączone do sieci KDNA bliższej antypodalnym kompleksom białkowym i luki są częściowo naprawiane.
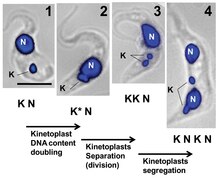
w miarę postępu replikacji minicircle, aby zapobiec powstawaniu nowych minicircle, cała sieć KDNA będzie obracać się wokół centralnej osi dysku. Uważa się, że rotacja jest bezpośrednio związana z replikacją sąsiedniego wici, ponieważ podstawowe ciało córki będzie również obracać się wokół podstawowego ciała matki w czasie i sposób podobny do rotacji kinetoplastu. Przez obracanie, minicircles kinetoplast córki są montowane w sposób spiralny i zaczynają poruszać się do wewnątrz w kierunku środka dysku, jak nowe minicircles są odłączone i przeniesione do KFZ do replikacji.
chociaż dokładne mechanizmy maxicircle kDNA nie zostały jeszcze określone w tych samych szczegółach, co minicircle kDNA, zaobserwowano strukturę zwaną nabelschnur (po niemiecku „pępowina”), która łączy sieci potomne kDNA, ale ostatecznie pęka podczas separacji. Wykorzystując sondy rybne do celowania w nabelschnur, stwierdzono, że zawiera maxicircle kDNA.
replikacja Kinetoplastów jest opisana jako występująca w pięciu etapach, każdy w odniesieniu do replikacji sąsiedniego wici.
- Etap I: Kinetoplast nie rozpoczął jeszcze replikacji, nie zawiera antypodalnych kompleksów białkowych i jest umiejscowiony w stosunku do pojedynczego flagellar basal body.
- Etap II: kinetoplast zaczyna wykazywać antypodowe kompleksy białkowe. Ciało podstawne flagellar rozpoczyna replikację, podobnie jak kinetoplast. Związek replikującego kinetoplastu z dwoma ciałami podstawnymi powoduje, że rozwija się kopulasty wygląd.
- Etap III: nowe flagellum zaczyna się rozdzielać, a kinetoplast nabiera bilobedowego kształtu.
- Etap IV: Kinetoplasty pojawiają się jako oddzielne dyski, ale pozostają połączone przez nabelschnura.
- etap V: kinetoplasty córki są całkowicie oddzielone, gdy nabelschnur jest uszkodzony. Ich struktura jest identyczna jak w etapie I.