y se describen por la disposición y ubicación de su kDNA.
- Un cinetoplasto pro-kDNA es una estructura similar a un haz que se encuentra en la matriz mitocondrial proximal al cuerpo basal flagelar. En contraste con la red convencional de kDNA, un cinetoplasto pro-kDNA contiene muy poca catenación y sus círculos máximos y mínimos están relajados en lugar de súper enrollados. Se ha observado Pro-kDNA en Bodo saltans, Bodo designis, Procryptobia sorokini syn. Bodo sorokini, Rhynchomonas nasuta y Cephalothamnium cyclopi.
- Un cinetoplasto de poli-kDNA es similar en estructura de kDNA a un cinetoplasto pro-kDNA. Contiene poca encadenamiento y no superenrollamiento. La característica distintiva del poli-kDNA es que en lugar de estar compuesto de un único haz globular como en el pro-kDNA, el poli-kDNA se distribuye entre varios focos discretos a lo largo de la luz mitocondrial. Se ha observado poli-kDNA en Dimastigella trypaniformis (un comensal en el intestino de una termita), Dismastigella mimosa (un cinetoplástido de vida libre) y Cruzella marina (un parásito del intestino de un chorro de mar).
- pan-kDNA cinetoplasto, como poli-kDNA y pro-kDNA, contiene un menor grado de encadenamiento pero contiene minicircles que se superenrollado. Los cinetoplastos Pan-kDNA llenan la mayor parte de la matriz mitocondrial y no se limitan a focos discretos como el poli-kDNA. Se ha observado Pan-kDNA en Cryptobia helicis (un parásito del receptaculum seminis de los caracoles), Bodo caudatus y Cryptobia branchialis (un parásito de los peces).
- Un cinetoplasto mega-kDNA se distribuye de manera bastante uniforme por toda la matriz mitocondrial, pero no contiene minicirculares. En cambio, secuencias de kDNA similares en secuencia a otros minicirculos de cinetoplasto se conectan en tándem en moléculas más grandes de aproximadamente 200 kb de longitud. Se han observado mega-kDNA (o estructuras similares a mega-kDNA) en Trypanoplasme borreli (un parásito de peces) y Jarrellia sp. (un parásito de ballena).
La presencia de esta variedad de estructuras de kDNA refuerza la relación evolutiva entre las especies de cinetoplástidos. Como pan-kDNA se parece más a un plásmido de ADN, puede ser la forma ancestral de kDNA.
Replicacióneditar

La replicación del cinetoplasto ocurre simultáneamente a la duplicación del flagelo adyacente y justo antes de la replicación del ADN nuclear. En una red tradicional de kDNA de Crithidia fasciculata, el inicio de la replicación es promovido por la desvinculación de los minicirculares de kDNA a través de la topoisomerasa II. Los minicirculares libres se liberan en una región entre el cinetoplasto y la membrana mitocondrial llamada zona cinetoflagelar (KFZ). Después de la replicación, los minicirculares migran por mecanismos desconocidos a los complejos de proteínas antipodales que contienen varias proteínas de replicación, incluyendo una endonucleasa, helicasa, ADN polimerasa, ADN primasa y ADN ligasa, que inician la reparación de las discontinuidades restantes en los minicirculares recién replicados.
Este proceso ocurre un minicircular a la vez, y solo un pequeño número de minicirculares se desvinculan en un momento dado. Para realizar un seguimiento de los minicirculares que se han replicado, al volver a unirse a la red kDNA, queda un pequeño espacio en los minicirculares nacientes, que los identifica como que ya se han replicado. Los minicirculares que aún no se han replicado siguen cerrados covalentemente. Inmediatamente después de la replicación, cada progenie se une a la red kDNA proximal a los complejos proteicos antípodas y los huecos se reparan parcialmente.
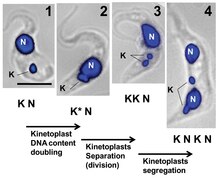
A medida que avanza la replicación de minicirculares, para evitar la acumulación de nuevos minicirculares, toda la red kDNA girará alrededor del eje central del disco. Se cree que la rotación está directamente conectada a la replicación del flagelo adyacente, ya que el cuerpo basal de la hija también girará alrededor del cuerpo basal de la madre en un tiempo y manera similar a la rotación del cinetoplasto. Al girar, los minicirculares del kinetoplasto hija se ensamblan en forma de espiral y comienzan a moverse hacia el interior hacia el centro del disco a medida que los nuevos minicirculares se desvinculan y se mueven hacia la KFZ para su replicación.
Mientras que los mecanismos exactos para el kDNA de círculo máximo aún no se han determinado con el mismo detalle que el kDNA de círculo mínimo, se observa una estructura llamada nabelschnur (en alemán, «cordón umbilical») que ata las redes de kDNA hija, pero finalmente se rompe durante la separación. Usando sondas de PECES para apuntar al nabelschnur, se ha encontrado que contiene kDNA de círculo máximo.
La replicación de cinetoplastos se describe en cinco etapas, cada una en relación con la replicación del flagelo adyacente.
- Estadio I: El cinetoplasto aún no ha iniciado la replicación, no contiene complejos de proteínas antípodas y está posicionado en relación con un solo cuerpo basal flagelar.
- Estadio II: El cinetoplasto comienza a mostrar complejos proteicos antípodas. El cuerpo basal flagelar comienza la replicación, al igual que el cinetoplasto. La asociación del cinetoplasto replicante a los dos cuerpos basales hace que desarrolle una apariencia abovedada.
- Etapa III: El nuevo flagelo comienza a separarse y el cinetoplasto toma una forma bilobulada.
- Estadio IV: Los cinetoplastos aparecen como discos separados, pero permanecen conectados por el nabelschnur.
- Etapa V: Los cinetoplastos hija están completamente separados al romperse el nabelschnur. Su estructura es idéntica a la que se ve en la etapa I.