výsledky a diskuse
amplifikovali jsme 307 BP fragment genů mitochondriálního cytochromu b z vydry Euroasijské Lutra lutra (Lotyšsko), L.lutra (Čína), l lutra (Evropa) a L. lutra (Asahikawa), asijské vydry malé drápy aonyx cinerea a lasice Mustela sibirica a Mustela itatsi, subklonovali amplifikované produkty v pUC18 a určili jejich sekvence. Sekvence L. lutra (Evropa) a L.lutra (Asahikawa) zcela souhlasili s l lutra (Čína) a L lutra (Lotyšsko). Sekvence dvou jedinců Mustela itatsi byly identické, ale lišily se ve třech pozicích od nedávno hlášených (Masuda a Yoshida, 1994), v sekvenčních pozicích 1-224(viz obr. 1).
obr. 1.
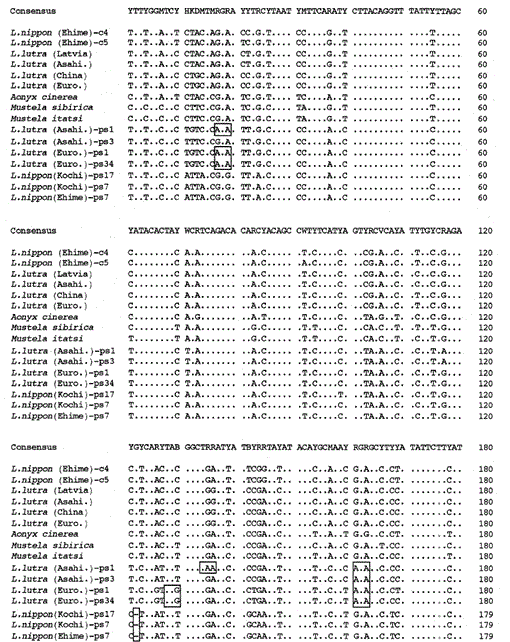
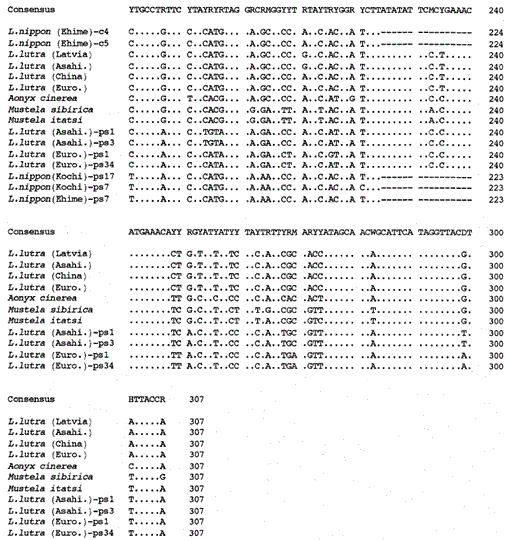
porovnání nukleotidových sekvencí mitochondriálního cytochromu b a cytochromu B podobných sekvencí Lutra nippon (Ehime a Kochi) s sekvencemi l lutra (Lotyšsko, Čína, Evropa a Asahikawa), aonyx cinerea, Mustela sibirica a M. itatsi. Délka sekvence, 224 bp pro L. nippon a 307 bp pro ostatní. Je ukázáno několik zástupců sekvencí podobných cytochromu b (ps). Pozice delečního a terminačního kodonu v sekvencích podobných cytochromu b jsou boxovány. Euro., Evropa; Asahi., Asahikawa.
obr. 1.
pokračování.
na druhé straně bylo provedeno mnoho pokusů o amplifikaci fragmentu DNA genu cytochromu B Lutra nippon pomocí různých 10-50 let starých vzorků, jako jsou játra, srdce a kůže konzervované ve formalinu, opálená kůže a chlupy vycpaného vzorku, ale všechny pokusy byly neúspěšné.
podařilo se nám amplifikovat fragment 224 bp genu mitochondriálního cytochromu b z 30letého mumie podobného vzorku Lutry nippon z Ehime (obr. 1). Přímé sekvenování amplifikovaných produktů naznačovalo přítomnost více než dvou typů sekvencí. Proto byly produkty subklonovány v pUC18 a 30 klonů bylo sekvenováno. Mezi nimi bylo nejednoznačně identifikováno devět klonů jako sekvence mitochondriálního cytochromu B Lutra nippon, protože sekvence byla zjevně podobná mitochondriálním sekvencím z jiných druhů Lutra, z nichž osm klonů mělo stejnou nukleotidovou sekvenci (reprezentovanou sekvencí L. nippon (Ehime) – c4 na obr. 1) ale zbývající (sekvence L. nippon (Ehime) – c5 na obr. 1) byl odlišný ve dvou polohách (21: C→T A 92: T→A). Jedenáct klonů mělo sekvence podobné cytochromu B, jak je popsáno níže. Zbývajících 10 klonů mělo neočekávaně téměř identickou sekvenci se sekvencí prasečího cytochromu b, což naznačuje, že během skladování vzorku podobného mumii došlo k vážné kontaminaci DNA.
zjistili jsme, že DNA z mumie podobné Lutra nippon (Ehime) obsahuje sekvenci podobnou cytochromu B. Překvapivě všechny amplifikované produkty z jiného Lutra nippon z Kochi (nejméně 30letý vycpaný vzorek) odpovídaly sekvencím podobným cytochromu b a nebyl amplifikován Žádný fragment mitochondriálního cytochromu b. Kromě toho byly amplifikovány fragmenty podobné cytochromu B 307 bp s výtěžkem 30-40% celkových klonů z DNAs připravených z čerstvých jater nebo chlupů vydry Euroasijské L. Lutry (Evropa a Asahikawa), ale ne z DNAs z L Lutry (Lotyšsko a Čína). Přítomnost sekvence podobné cytochromu b tedy není artefaktem během dlouhého skladování DNA. Sekvence podobná cytochromu b se také nachází v jaderné DNA u hlodavce (Smith et al ., 1992). Sekvence Lutry podobné cytochromu B jsou charakterizovány přítomností delece, která způsobuje posun čtecího rámce a terminační kodony (obr. 1), přítomností několika typů sekvencí s menšími odchylkami (obr. 1) a zjevně rychlejší evoluční rychlostí ve srovnání se sekvencí mitochondriálního cytochromu b (viz obr. 2). Zdá se tedy, že sekvence podobná cytochromu b nefunguje a je přítomna jako pseudoforma. Nezkoumali jsme, zda je sekvence Lutra podobná cytochromu B přítomna v mitochondriální DNA nebo v jaderné DNA.
obr. 2.
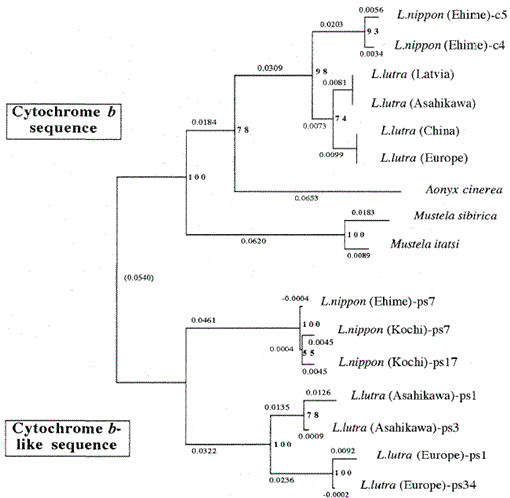
fylogenetický strom vytvořený z zarovnání sekvence (na základě sekvence 1-224 na obr. 1) sekvencí mitochondriálního cytochromu b a cytochromu B s metodou sousedního spojení. Čísla v každém větvícím bodě ukazují hodnoty bootstrapu (100 replikací), ps představuje sekvenci podobnou cytochromu b.
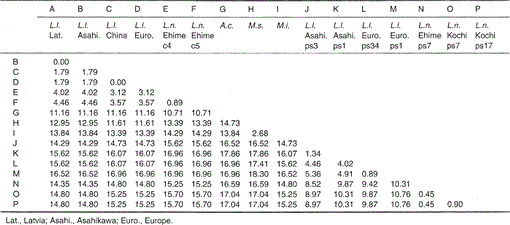
nukleotidové sekvence mitochondriálního cytochromu b a cytochromu B-like genů Lutra nippon jsou zarovnány s sekvencemi L. Lutry (Lotyšsko, Čína, Evropa a Asahikawa), aonyx cinerea, Mustela sibirica a Mustela itatsi, na obr. 1. Ze sekvence 1 až 224 byla Lutra nippon (C4) odlišná od L lutra (Lotyšsko) a L.lutra (Čína) v 9 a 7 nukleotidech, z nichž všechny byly umístěny na třetí pozici kodonu a identifikovány jako přechodné rozdíly a↔G nebo C T. T. rozdíl v Lutra lutra byl 4 nukleotidy. Tyto substituce nukleotidů nezpůsobují náhradu aminokyselin. Na druhé straně existuje 6 nukleotidů rozdíl ve dvou kongenerických lasicích Mustela sibirica a Mustela itatsi v této oblasti. Rozdíl sekvence mezi Lutra nippon a Lutra lutra (7-9 nukleotidů: v průměru 3, 6%) je tedy větší než rozdíl (6 nukleotidů) mezi dvěma druhy Mustela. To je také v souladu s myšlenkou, že u druhů Mustelidae může více než 3,5% rozdíl v sekvenci genu cytochromu b odpovídat rozdílu odlišných druhů (Masuda a Yoshida, 1994). Procentní rozdíl mezi nukleotidovými sekvencemi je uveden v tabulce 1.
Tabulka 1.
procentní rozdíl mezi nukleotidovými sekvencemi (224 bp) znázorněnými na obr. 1
Obrázek 2 ukazuje fylogenetický strom vytvořený ze sekvenčních dat získaných v této studii metodou sousedního spojování ve verzi phylip package 3.5 c (Felsenstein, 1993). Stejná topologie byla rekonstruována metodou Fitch a Margoliash (Felsenstein, 1993). Sekvence byly rozděleny do dvou hlavních shluků, jeden je pro geny mitochondriálního cytochromu b a druhý pro sekvence podobné cytochromu b. Jak bylo uvedeno výše, sekvence podobné cytochromu B byly amplifikovány z dna Lutra nippon (2 jedinci, z Kochi a Ehime), L. lutra (Evropa) a L. lutra (Asahikawa). Větvení sekvencí podobných cytochromu b dvou druhů Lutra bylo stejné jako v genech mitochondriálního cytochromu b, ale evoluční rychlost pro sekvence podobné cytochromu b byla alespoň dvakrát rychlejší, v souladu s obecnými charakteristikami pseudo-genů. Strom také jasně ukazuje, že existuje velký genetický rozdíl mezi Lutra nippon a Lutra lutra, a tento výsledek je v souladu s nedávnou morfologickou studií, že japonská říční vydra není poddruhem vydry Euroasijské Lutra lutra, ale zřetelným druhem Lutra nippon (Imaizumi a Yoshiyuki, 1989). Aby však bylo možné získat přesvědčivé důkazy o fylogenetickém vztahu mezi těmito dvěma druhy, musí být stanovena delší sekvence DNA Lutra nippon. To by bylo provedeno, když byl získán čerstvý vzorek japonské říční vydry.