RESULTS AND DISCUSSION
we hebben het 307 BP fragment van de mitochondriale cytochroom B genen van de Euraziatische otters Lutra lutra (Letland), L. lutra (China), l lutra (Europa) en L. lutra (Asahikawa), de Aziatische kleingeklauwde otter Aonyx cinerea en de wezels Mustela sibirica en Mustela itatsi, gesubcloneerd de versterkte producten in pUC18 en bepaald hun sequenties. De sequenties van L. lutra (Europa) en L. lutra (Asahikawa) waren het volledig eens met die van L lutra (China) en L lutra (Letland), respectievelijk. De sequenties van twee individuen van Mustela itatsi waren identiek, maar verschillend in drie posities van die recent gerapporteerd (Masuda en Yoshida, 1994), in de sequentie posities 1-224 (zie Fig. 1).
Fig. 1.
vergelijking van de nucleotidesequenties van het mitochondriale cytochroom B en cytochroom b-achtige sequenties van Lutra nippon (Ehime en Kochi) met die van L lutra (Letland, China, Europa en Asahikawa), Aonyx cinerea, Mustela sibirica en M. itatsi. De sequentie lengte, 224 bp voor L. nippon en 307 bp voor de anderen. Verschillende vertegenwoordigers van de cytochroom b-achtige sequenties (ps) worden getoond. Posities van de schrapping en beëindiging codon in de cytochroom b-achtige sequenties zijn boxed. Eurozone., Europa; Asahi. Asahikawa.
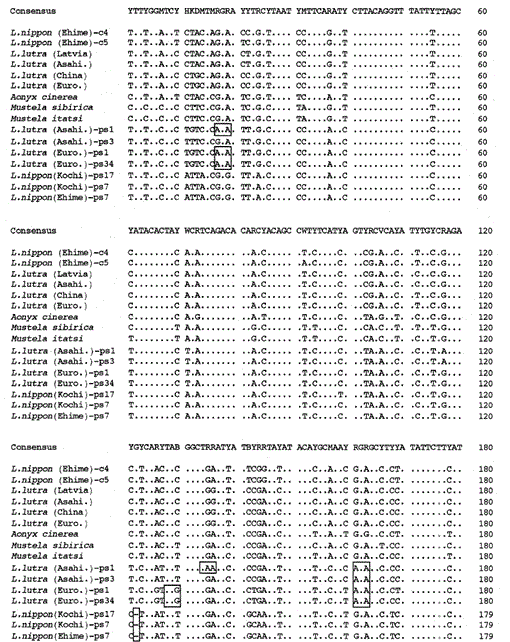
Fig. 1.
voortgezet.
aan de andere kant werden veel pogingen gedaan om het DNA-fragment van het cytochroom B-gen van Lutra nippon te versterken met behulp van verschillende 10-50 jaar oude monsters, zoals de lever, het hart en de huid die in formaline zijn bewaard, het gelooide leer en de haren van de gevulde exemplaren, maar alle pogingen waren niet succesvol.
we zijn erin geslaagd het 224 bp fragment van het mitochondriale cytochroom B gen te versterken van een 30 jaar oud mummieachtig specimen van Lutra nippon uit Ehime (Fig. 1). Het directe rangschikken van de versterkte producten stelde de aanwezigheid van meer dan twee soorten opeenvolging voor. Daarom werden de producten gesubkloneerd in pUC18 en werden 30 klonen gesequenced. Onder hen werden negen klonen dubbelzinnig geïdentificeerd als de mitochondriale cytochroom B-sequentie van Lutra nippon omdat de sequentie blijkbaar vergelijkbaar was met de mitochondriale sequenties van andere Lutra-soorten, waarvan acht klonen dezelfde nucleotidesequentie hadden (vertegenwoordigd door de sequentie van L. nippon (Ehime) – c4 in Fig. 1) maar de resterende (de volgorde van L. nippon (Ehime) – c5 in Fig. 1) was verschillend in twee posities (21: C→T en 92: T→A). De elf klonen hadden de cytochroom b-achtige sequenties zoals hieronder beschreven. De resterende 10 klonen hadden onverwacht de bijna identieke sequentie als de varkenscytochroom B-sequentie, wat suggereert dat er een ernstige DNA-besmetting plaatsvond tijdens de opslag van het mummieachtige Monster.
we vonden dat het DNA van een mummieachtige Lutra nippon (Ehime) een cytochroom-B-achtige sequentie bevat. Verrassend, alle van de versterkte producten van een andere Lutra nippon van Kochi (ten minste 30-jaar-oude gevulde specimen) overeen met de cytochroom b-achtige sequenties, en geen mitochondriale cytochroom B fragment werd versterkt. Bovendien werden de cytochroom B-achtige fragmenten van 307 bp versterkt, met een opbrengst van 30-40% van de totale klonen, uit de DNAs bereid uit de verse lever of haren van de Euraziatische otters L. lutra (Europa en Asahikawa), maar niet uit de DNAs van L lutra (Letland en China). Aldus is de aanwezigheid van cytochroom B-als opeenvolging geen artefact tijdens een lange opslag van DNA. De cytochroom b-achtige sequentie worden ook gevonden in het nucleaire DNA in een knaagdier (Smith et al., 1992). De cytochroom B-achtige opeenvolgingen van Lutra worden gekenmerkt door de aanwezigheid van een schrapping om een verschuiving van het leesframe en beëindigingscodon (Fig. 1), door de aanwezigheid van verschillende soorten sequenties met kleine variaties (Fig. 1), en door de blijkbaar snellere evolutionaire snelheid vergeleken met de mitochondriale cytochroom B-sequentie (zie Fig. 2). De cytochroom B-achtige sequentie lijkt dus niet te functioneren en is aanwezig als pseudo-vorm. We hebben niet onderzocht of de cytochroom b-achtige sequentie van Lutra aanwezig is in het mitochondriale DNA of in het nucleaire DNA.
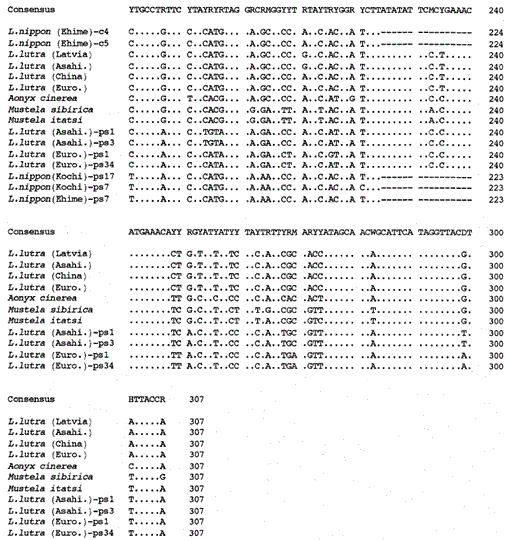
Fig. 2.
een fylogenetische boom opgebouwd uit de sequentieuitlijning (gebaseerd op sequentie 1-224 in Fig. 1) van de mitochondriale cytochroom B en cytochroom b-achtige sequenties met de Neighbor-Joining methode. De getallen op elk branching point tonen bootstrap waarden (100 replicaties), ps vertegenwoordigt de cytochroom b-achtige sequentie.
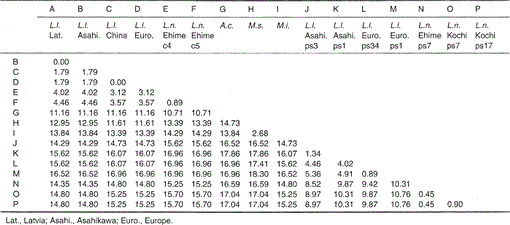
de nucleotide sequenties van de mitochondriale cytochroom B en cytochroom b-achtige genen van Lutra nippon zijn afgestemd op die van L. lutra (Letland, China, Europa en Asahikawa), Aonyx cinerea, Mustela sibirica en Mustela itatsi, in Fig. 1. Van de sequentie 1 tot 224 was Lutra nippon (C4) verschillend van die van L lutra (Letland) en L. lutra (China) in respectievelijk 9 en 7 nucleotiden, die allemaal op de derde positie van een codon lagen en geïdentificeerd werden als overgangsverschillen A↔G of C↔T. Het verschil binnen Lutra lutra was 4 nucleotiden. Deze nucleotide substituties veroorzaken geen aminozuurvervanging. Aan de andere kant, er zijn 6 nucleotiden verschil in de twee congeneren wezels Mustela sibirica en Mustela itatsi in dit gebied. Zo is het sequentieverschil tussen Lutra nippon en Lutra lutra (7-9 nucleotiden: gemiddeld 3,6%) groter dan dat (6 nucleotiden) tussen de twee Mustela-soorten. Dit is ook consistent met het idee dat bij de Mustelidae-soorten, meer dan 3,5% verschil in volgorde van cytochroom B-gen kan overeenkomen met het verschil van verschillende soorten (Masuda and Yoshida, 1994). Het procentuele verschil tussen de nucleotidesequenties wordt weergegeven in Tabel 1.
Tabel 1.
Percentage verschil tussen de nucleotidesequenties (224 bp) weergegeven in Fig. 1
Figuur 2 toont een fylogenetische boom opgebouwd uit de sequentiegegevens verkregen in deze studie met de Neighbor-Joiningmethode in het FYLIPPAKKET Versie 3.5 c (felsenstein, 1993). Dezelfde topologie werd gereconstrueerd met de Fitch en Margoliash methode (felsenstein, 1993). De opeenvolgingen werden verdeeld in twee hoofdclusters, is één voor de mitochondrial cytochroom B genen en andere voor cytochroom B-als opeenvolgingen. Zoals hierboven vermeld, werden de cytochroom B-achtige sequenties versterkt uit de DNAs van Lutra nippon (2 individuen, van Kochi en Ehime), L. lutra (Europa) en L. lutra (Asahikawa). Het vertakkingspatroon van de cytochroom B-achtige sequenties van de twee Lutra-soorten was hetzelfde als in de mitochondriale cytochroom B-genen, maar de evolutionaire snelheid voor de cytochroom b-achtige sequenties was minstens twee keer sneller, in overeenstemming met de Algemene kenmerken van pseudogenen. De boom toont ook duidelijk aan dat er een groot genetisch verschil is tussen Lutra nippon en Lutra lutra, en dit resultaat is consistent met de recente morfologische studie dat de Japanse rivierotter geen ondersoort is van de otter Lutra lutra, maar een aparte soort, Lutra nippon (Imaizumi and Yoshiyuki, 1989). Echter, om een sluitend bewijs voor de fylogenetische relatie tussen de twee soorten te krijgen, langere DNA-sequentie van Lutra nippon moet worden bepaald. Dit zou gebeuren wanneer het verse monster van de Japanse rivierotter werd verkregen.