rezultate și discuții
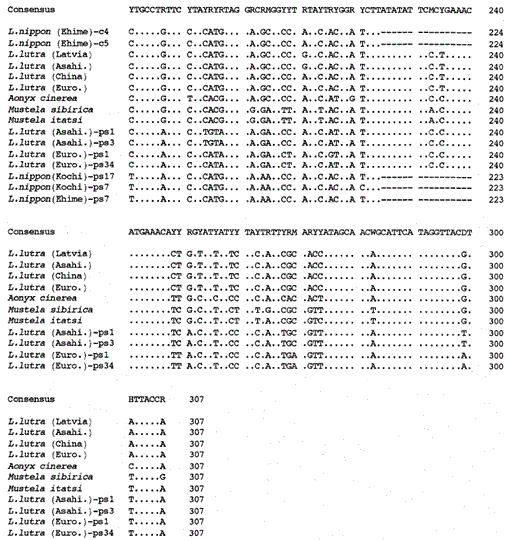
am amplificat fragmentul de 307 bp al genelor citocromului B mitocondrial de la vidrele Eurasiatice Lutra lutra (Letonia), L. lutra (China), L lutra (Europa) și L. lutra (Asahikawa), Vidra asiatică cu gheare mici aonyx cinerea și nevăstuicile Mustela sibirica și Mustela itatsi, au subclonat produsele amplificate în puc18 și a determinat secvențele lor. Secvențele lui L. lutra (Europa) și L. lutra (Asahikawa) au fost de acord în totalitate cu cele ale lutra (China) și, respectiv, lutra (Letonia). Secvențele a doi indivizi din Mustela itatsi au fost identice, dar diferite în trei poziții față de cele raportate recent (Masuda și Yoshida, 1994), în pozițiile secvenței 1-224 (vezi Fig. 1).
Fig. 1.
Compararea secvențelor nucleotidice ale citocromului mitocondrial B și secvențelor asemănătoare citocromului B ale Lutra Nippon (Ehime și Kochi) cu cele ale L lutra (Letonia, China, Europa și Asahikawa), aonyx cinerea, Mustela sibirica și M. itatsi. Lungimea secvenței, 224 bp pentru L. nippon și 307 bp pentru ceilalți. Sunt prezentați mai mulți reprezentanți ai secvențelor asemănătoare citocromului b (ps). Pozițiile codonului de ștergere și terminare în secvențele asemănătoare citocromului b sunt cutie. Euro., Europa; Asahi., Asahikawa.
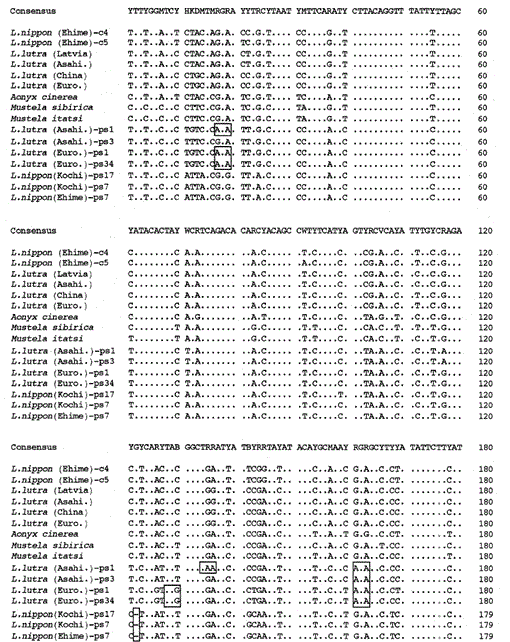
Fig. 1.
continuare.
pe de altă parte, s-au făcut multe încercări de amplificare a fragmentului ADN al genei citocromului B A Lutra nippon folosind diferite probe vechi de 10-50 de ani, cum ar fi ficatul, inima și pielea conservate în formalină, pielea tăbăcită și firele de păr ale specimenului umplut, dar toate încercările nu au avut succes.
am reușit să amplificăm fragmentul de 224 bp al genei mitocondriale a citocromului B dintr-un specimen de Lutra nippon de 30 de ani de la Ehime (Fig. 1). Secvențierea directă a produselor amplificate a sugerat prezența a mai mult de două tipuri de secvențe. Prin urmare, produsele au fost subclonate în pUC18 și 30 de clone au fost secvențiate. Dintre acestea, nouă clone au fost identificate ambiguu ca fiind secvența citocromului B mitocondrial a Lutra nippon, deoarece secvența era aparent similară cu secvențele mitocondriale din alte specii Lutra, dintre care opt clone aveau aceeași secvență nucleotidică (reprezentată de secvența lui L. nippon (Ehime)-c4 în Fig. 1) dar restul (secvența lui L. nippon (Ehime)-c5 în Fig. 1) a fost diferit în două poziții (21: C→T și 92: T→O). Cele unsprezece clone au avut secvențe asemănătoare citocromului b, așa cum este descris mai jos. Restul de 10 clone, în mod neașteptat, au avut secvența aproape identică cu secvența citocromului B de porc, sugerând că s-a produs o contaminare gravă a ADN-ului în timpul depozitării specimenului asemănător mumiei.
am descoperit că ADN-ul unei Lutra Nippon (Ehime) asemănătoare unei mumii conține o secvență asemănătoare citocromului B. În mod surprinzător, toate produsele amplificate de la un alt Lutra nippon de la Kochi (specimen umplut de cel puțin 30 de ani) corespundeau secvențelor asemănătoare citocromului b și Niciun fragment de citocrom B mitocondrial nu a fost amplificat. În plus, fragmentele citocromului B de 307 bp au fost amplificate, cu un randament de 30-40% din totalul clonelor, din ADN-urile preparate din ficatul proaspăt sau firele de păr ale vidrelor Eurasiatice L. lutra (Europa și Asahikawa), dar nu din ADN-urile din L lutra (Letonia și China). Astfel, prezența secvenței de tip citocrom B nu este un artefact în timpul depozitării îndelungate a ADN-ului. Secvența asemănătoare citocromului b se găsește și în ADN-ul nuclear dintr-o rozătoare (Smith și colab., 1992). Secvențele de Lutra asemănătoare citocromului B se caracterizează prin prezența unei deleții pentru a provoca o schimbare a cadrului de citire și codoni de terminare (Fig. 1), prin prezența mai multor tipuri de secvențe cu variații minore (Fig. 1) și prin rata evolutivă aparent mai rapidă comparativ cu secvența citocromului B mitocondrial (vezi Fig. 2). Astfel, secvența asemănătoare citocromului b pare să nu funcționeze și este prezentă ca o pseudo-formă. Nu am examinat dacă secvența de Lutra asemănătoare citocromului B este prezentă în ADN-ul mitocondrial sau în ADN-ul nuclear.
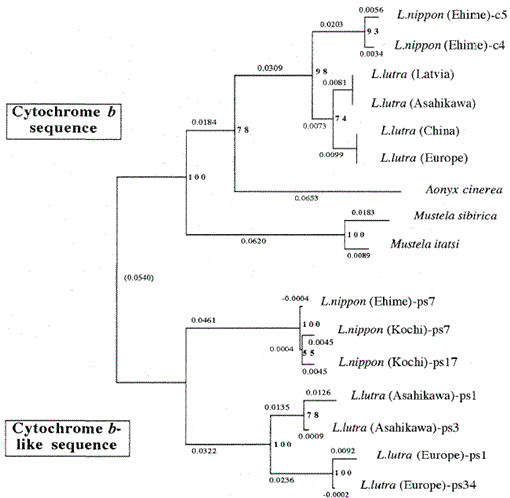
Fig. 2.
un arbore filogenetic Construit din alinierea secvenței (pe baza secvenței 1-224 din Fig. 1) din secvențele mitocondriale ale citocromului B și citocromului B cu metoda de îmbinare a vecinului. Numerele de la fiecare punct de ramificare arată valorile bootstrap (100 de replicări), ps reprezintă secvența asemănătoare citocromului B.
secvențele nucleotidice ale genelor mitocondriale ale citocromului B și citocromului B ale Lutra nippon sunt aliniate cu cele ale L. lutra (Letonia, China, Europa și Asahikawa), aonyx cinerea, Mustela sibirica și Mustela itatsi, în Fig. 1. Din secvența 1 până la 224, Lutra nippon (C4) a fost diferită de cele ale L lutra (Letonia) și L. lutra (China) în 9 și, respectiv, 7 nucleotide, toate acestea fiind situate în a treia poziție a unui codon și identificate ca diferențe de tranziție a G sau C XCT T. diferența dintre Lutra lutra a fost de 4 nucleotide. Aceste substituții nucleotidice nu determină înlocuirea aminoacizilor. Pe de altă parte, există 6 diferențe de nucleotide în cele două nevăstuici congenerice Mustela sibirica și Mustela itatsi din această regiune. Astfel, diferența de secvență dintre Lutra nippon și Lutra lutra (7-9 nucleotide: 3,6% în medie) este mai mare decât cea (6 nucleotide) dintre cele două specii Mustela. Acest lucru este, de asemenea, în concordanță cu ideea că la speciile Mustelidae, mai mult de 3,5% diferență în secvența genei citocromului b poate corespunde diferenței speciilor distincte (Masuda și Yoshida, 1994). Diferența procentuală dintre secvențele de nucleotide este prezentată în tabelul 1.
Tabelul 1.
diferența procentuală dintre secvențele de nucleotide (224 bp) prezentate în Fig. 1
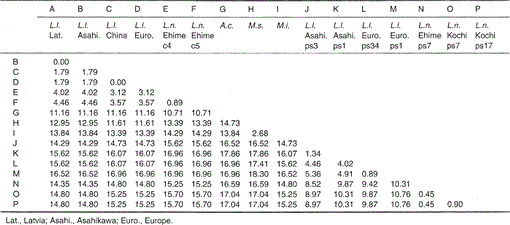
Figura 2 prezintă un arbore filogenetic Construit din datele de secvență obținute în acest studiu cu metoda de îmbinare vecin în pachetul PHYLIP versiunea 3.5 c (Felsenstein, 1993). Aceeași topologie a fost reconstruită cu metoda Fitch și Margoliash (Felsenstein, 1993). Secvențele au fost împărțite în două grupuri principale, unul este pentru genele mitocondriale ale citocromului b și celălalt pentru secvențele asemănătoare citocromului B. După cum sa menționat mai sus, secvențele de tip citocrom b au fost amplificate din ADN-ul Lutra Nippon (2 indivizi, de la Kochi și Ehime), L. lutra (Europa) și L. lutra (Asahikawa). Modelul de ramificare al secvențelor asemănătoare citocromului b ale celor două specii Lutra a fost același ca în genele citocromului B mitocondrial, dar rata evolutivă pentru secvențele asemănătoare citocromului b a fost de cel puțin două ori mai rapidă, în acord cu caracteristicile generale ale pseudo-genelor. Arborele arată, de asemenea, în mod clar că există o mare diferență genetică între Lutra nippon și Lutra lutra, iar acest rezultat este în concordanță cu studiul morfologic recent conform căruia Vidra râului japonez nu este o subspecie a vidrei Eurasiatice Lutra lutra, ci o specie distinctă, Lutra Nippon (Imaizumi și Yoshiyuki, 1989). Cu toate acestea, pentru a obține o dovadă concludentă pentru relația filogenetică dintre cele două specii, trebuie determinată o secvență ADN mai lungă de Lutra nippon. Acest lucru s-ar face atunci când a fost obținut eșantionul proaspăt al vidrei râului Japonez.