RESULTADOS Y DISCUSIÓN
Hemos amplificado el fragmento de 307 bp de los genes del citocromo b mitocondrial de las nutrias eurasiáticas Lutra lutra (Letonia), L. lutra (China), L lutra (Europa) y L. lutra (Asahikawa), la nutria asiática de garras pequeñas Aonyx cinerea y las comadrejas Mustela sibirica y Mustela itatsi, subclonaron los productos amplificados en pUC18 y determinaron sus secuencias. Las secuencias de L. lutra (Europa) y L. lutra (Asahikawa) estuvieron completamente de acuerdo con los de L lutra (China) y L lutra (Letonia), respectivamente. Las secuencias de dos individuos de Mustela itatsi fueron idénticas, pero diferentes en tres posiciones de las reportadas recientemente (Masuda y Yoshida, 1994), en las posiciones de secuencia 1-224 (ver Fig. 1).
Fig. 1.
Comparación de las secuencias de nucleótidos del citocromo b mitocondrial y secuencias similares al citocromo b de Lutra nippon (Ehime y Kochi) con las de L lutra (Letonia, China, Europa y Asahikawa), Aonyx cinerea, Mustela sibirica y M. itatsi. La longitud de la secuencia, 224 pb para L. nippon y 307 pb para los demás. Se muestran varios representantes de las secuencias similares al citocromo b (ps). Las posiciones del codón de eliminación y terminación en las secuencias similares al citocromo b se encajonan. Euro., Europe; Asahi., Asahikawa.
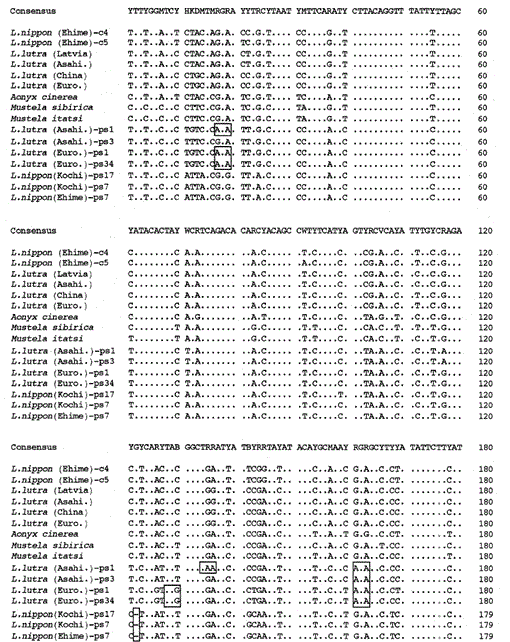
Fig. 1.
Continuación.
Por otro lado, se hicieron muchos intentos de amplificar el fragmento de ADN del gen citocromo b de Lutra nippon utilizando varias muestras de 10 a 50 años, como el hígado, el corazón y la piel conservados en formalina, el cuero curtido y los pelos de espécimen relleno, pero todos los intentos no tuvieron éxito.
Hemos logrado amplificar el fragmento de 224 pb del gen del citocromo b mitocondrial a partir de un espécimen de Lutra nippon de Ehime de 30 años de edad, similar a una momia (Fig. 1). La secuenciación directa de los productos amplificados sugirió la presencia de más de dos tipos de secuencia. Por lo tanto, los productos se subclonaron en pUC18 y se secuenciaron 30 clones. Entre ellos, nueve clones fueron identificados ambiguamente como la secuencia del citocromo b mitocondrial de Lutra nippon, ya que la secuencia era aparentemente similar a las secuencias mitocondriales de otras especies de Lutra, de las cuales ocho clones tenían la misma secuencia de nucleótidos (representada por la secuencia de L. nippon (Ehime)-c4 en la Fig. 1) pero el restante (la secuencia de L. nippon (Ehime) – c5 en la Fig. 1) era diferente en dos posiciones (21: C→T y 92: T→A). Los once clones tenían secuencias similares al citocromo b como se describe a continuación. Los 10 clones restantes, inesperadamente, tenían la secuencia casi idéntica a la secuencia del citocromo b de cerdo, lo que sugiere que se produjo una contaminación grave del ADN durante el almacenamiento del espécimen similar a una momia.
Encontramos que el ADN de un Lutra nippon (Ehime) similar a una momia contiene una secuencia similar al citocromo b. Sorprendentemente, todos los productos amplificados de otro Lutra nippon de Kochi (un espécimen relleno de al menos 30 años de edad) correspondían a secuencias similares al citocromo b, y no se amplificó ningún fragmento del citocromo b mitocondrial. Además, los fragmentos similares al citocromo b de 307 pb se amplificaron, con un rendimiento del 30-40% de los clones totales, a partir de los ADN preparados a partir del hígado fresco o pelos de las nutrias euroasiáticas L. lutra (Europa y Asahikawa), pero no de los ADN de L lutra (Letonia y China). Por lo tanto, la presencia de una secuencia similar al citocromo b no es un artefacto durante un almacenamiento prolongado de ADN. La secuencia similar al citocromo b también se encuentra en el ADN nuclear de un roedor (Smith et al., 1992). Las secuencias similares al citocromo b de Lutra se caracterizan por la presencia de una deleción que causa un desplazamiento del marco de lectura y codones de terminación (Fig. 1), por la presencia de varios tipos de secuencias con variaciones menores (Fig. 1), y por la velocidad evolutiva aparentemente más rápida en comparación con la secuencia del citocromo b mitocondrial (ver Fig. 2). Por lo tanto, la secuencia similar al citocromo b parece no funcionar y está presente como una pseudo-forma. No hemos examinado si la secuencia similar al citocromo b de Lutra está presente en el ADN mitocondrial o en el ADN nuclear.
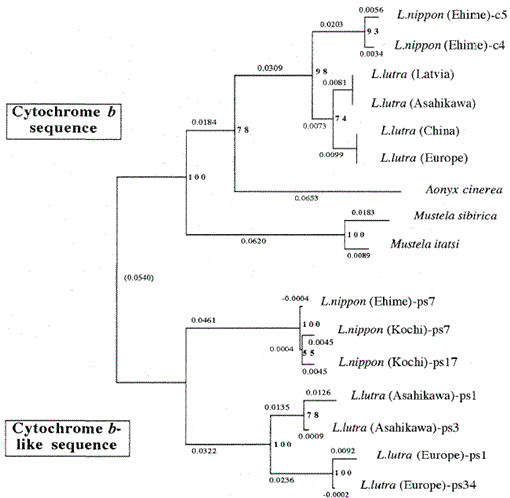
Fig. 2.
Un árbol filogenético construido a partir de la alineación de secuencias (basado en la secuencia 1-224 de la Fig. 1) de las secuencias mitocondriales del citocromo b y del citocromo b con el método de Unión de vecinos. Los números en cada punto de ramificación muestran valores de arranque (100 replicaciones), ps representa la secuencia similar al citocromo b.
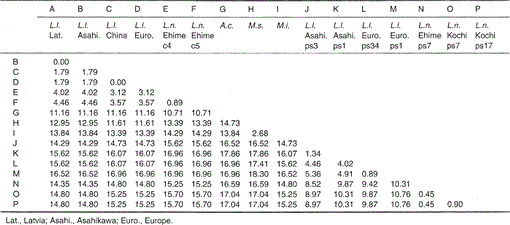
Las secuencias de nucleótidos del citocromo b mitocondrial y de genes similares al citocromo b de Lutra nippon están alineadas con las de L. lutra (Letonia, China, Europa y Asahikawa), Aonyx cinerea, Mustela sibirica y Mustela itatsi, en la Fig. 1. De la secuencia 1 a 224, Lutra nippon (C4) fue diferente de los de L lutra (Letonia) y L. lutra (China) en 9 y 7 nucleótidos, respectivamente, todos los cuales estaban ubicados en la tercera posición de un codón e identificados como diferencias de transición A↔G o C↔T. La diferencia dentro de Lutra lutra fue de 4 nucleótidos. Estas sustituciones de nucleótidos no causan reemplazo de aminoácidos. Por otro lado, hay 6 diferencias de nucleótidos en las dos comadrejas congéneres Mustela sibirica y Mustela itatsi en esta región. Por lo tanto, la diferencia de secuencia entre Lutra nippon y Lutra lutra (7-9 nucleótidos: 3,6% en promedio) es mayor que la diferencia (6 nucleótidos) entre las dos especies de Mustelas. Esto también es consistente con la idea de que en las especies de Mustélidos, una diferencia de más del 3,5% en la secuencia del gen del citocromo b puede corresponder a la diferencia de especies distintas (Masuda y Yoshida, 1994). La diferencia porcentual entre las secuencias de nucleótidos se muestra en la Tabla 1.
Cuadro 1.
Diferencia porcentual entre las secuencias de nucleótidos (224 pb) mostrada en la Fig. 1
La Figura 2 muestra un árbol filogenético construido a partir de los datos de secuencia obtenidos en este estudio con el método de Unión de vecinos en el paquete PHYLIP versión 3.5 c (Felsenstein, 1993). La misma topología fue reconstruida con el método Fitch y Margoliash (Felsenstein, 1993). Las secuencias se dividieron en dos grupos principales, uno para los genes mitocondriales del citocromo b y el otro para secuencias similares al citocromo b. Como se indicó anteriormente, las secuencias similares al citocromo b se amplificaron a partir de las ADN de Lutra nippon (2 individuos, de Kochi y Ehime), L. lutra (Europa) y L. lutra (Asahikawa). El patrón de ramificación de las secuencias similares al citocromo b de las dos especies Lutra fue el mismo que en los genes mitocondriales del citocromo b, pero la tasa evolutiva de las secuencias similares al citocromo b fue al menos dos veces más rápida, de acuerdo con las características generales de los pseudo genes. El árbol también muestra claramente que hay una gran diferencia genética entre Lutra nippon y Lutra lutra, y este resultado es consistente con el reciente estudio morfológico de que la nutria de río japonesa no es una subespecie de la nutria euroasiática Lutra Lutra, sino una especie distinta, Lutra nippon (Imaizumi y Yoshiyuki, 1989). Sin embargo, para obtener una evidencia concluyente de la relación filogenética entre las dos especies, se debe determinar una secuencia de ADN más larga de Lutra nippon. Esto se haría cuando se obtuviera la muestra fresca de nutria de río japonesa.