ERGEBNISSE UND DISKUSSION
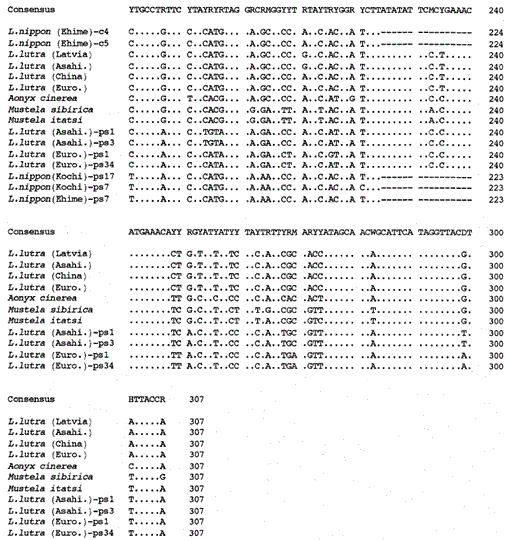
Wir haben das 307 bp Fragment der mitochondrialen Cytochrom b Gene von den eurasischen Fischottern Lutra lutra (Lettland), L. lutra (China), L lutra (Europa) und L. lutra (Asahikawa), dem asiatischen Kleinkrallenotter Aonyx cinerea und den Wieseln Mustela sibirica und Mustela itatsi amplifiziert, die amplifizierten Produkte in pUC18 und bestimmt ihre Sequenzen. Die Sequenzen von L. lutra (Europa) und L. lutra (Asahikawa) stimmten vollständig mit denen von L lutra (China) bzw. L lutra (Lettland) überein. Die Sequenzen von zwei Individuen von Mustela itatsi waren identisch, unterschieden sich jedoch in drei Positionen von denen, die kürzlich berichtet wurden (Masuda und Yoshida, 1994), in den Sequenzpositionen 1-224 (siehe Abb. 1).
Abb. 1.
Vergleich der Nukleotidsequenzen der mitochondrialen Cytochrom b und Cytochrom b-ähnlichen Sequenzen von Lutra nippon (Ehime und Kochi) mit denen von L lutra (Lettland, China, Europa und Asahikawa), Aonyx cinerea, Mustela sibirica und M. itatsi. Die Sequenzlänge, 224 bp für L. nippon und 307 bp für die anderen. Mehrere Vertreter der Cytochrom-b-ähnlichen Sequenzen (ps) sind dargestellt. Positionen des Deletions- und Terminationscodons in den Cytochrom b-ähnlichen Sequenzen sind boxed. Euro., Europa; Asahi. In : Asahikawa.
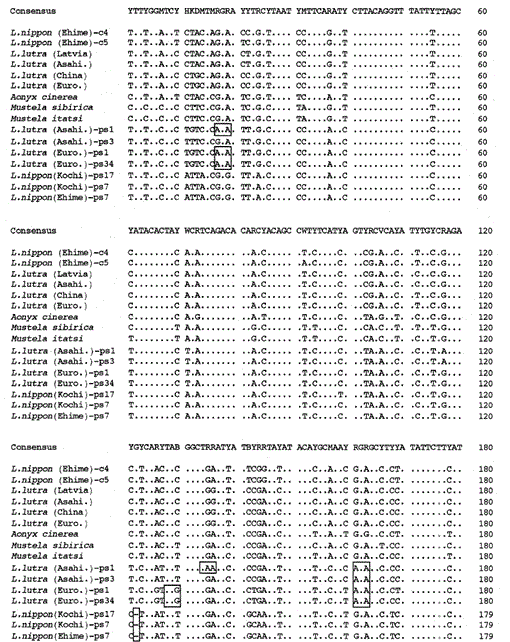
Abb. 1.
Fortsetzung.
Auf der anderen Seite wurden viele Versuche unternommen, das DNA-Fragment des Cytochrom-b-Gens von Lutra Nippon unter Verwendung verschiedener 10-50 Jahre alter Proben, wie der in Formalin konservierten Leber, des Herzens und der Haut, des gegerbten Leders und der Haare der Probe, zu amplifizieren, aber alle Versuche waren erfolglos.
Es ist uns gelungen, das 224 bp Fragment des mitochondrialen Cytochrom b Gens aus einer 30 Jahre alten mumienartigen Probe von Lutra nippon aus Ehime zu amplifizieren (Abb. 1). Die direkte Sequenzierung der amplifizierten Produkte deutete auf das Vorhandensein von mehr als zwei Sequenztypen hin. Dazu wurden die Produkte in pUC18 subkloniert und 30 Klone sequenziert. Unter ihnen wurden neun Klone mehrdeutig als die mitochondriale Cytochrom-b-Sequenz von Lutra nippon identifiziert, da die Sequenz anscheinend den mitochondrialen Sequenzen anderer Lutra-Spezies ähnlich war, von denen acht Klone die gleiche Nukleotidsequenz aufwiesen (dargestellt durch die Sequenz von L. nippon (Ehime) -c4 in Abb. 1), aber die verbleibende (die Sequenz von L. nippon (Ehime)-c5 in Abb. 1) war in zwei Positionen unterschiedlich (21: C→T und 92: T→A). Die elf Klone wiesen die Cytochrom-b-ähnlichen Sequenzen auf, wie unten beschrieben. Der Rest 10 Klone, unerwartet, hatte die fast identische Sequenz mit der Cytochrom-b-Sequenz des Schweins, Dies deutet darauf hin, dass während der Lagerung der mumienartigen Probe eine ernsthafte DNA-Kontamination auftrat.
Wir fanden heraus, dass die DNA eines mumienähnlichen Lutra Nippon (Ehime) eine Cytochrom b-ähnliche Sequenz enthält. Überraschenderweise entsprachen alle amplifizierten Produkte aus einem anderen Lutra nippon aus Kochi (mindestens 30 Jahre altes ausgestopftes Exemplar) den Cytochrom b-ähnlichen Sequenzen, und es wurde kein mitochondriales Cytochrom b-Fragment amplifiziert. Zusätzlich wurden die Cytochrom b-ähnlichen Fragmente von 307 bp mit einer Ausbeute von 30-40% der Gesamtklone aus den DNAs amplifiziert, die aus der frischen Leber oder Haaren der eurasischen Otter L. lutra (Europa und Asahikawa) hergestellt wurden, nicht jedoch aus den DNAs von L lutra (Lettland und China). Somit ist das Vorhandensein einer Cytochrom-b-ähnlichen Sequenz kein Artefakt während einer langen Lagerung von DNA. Die Cytochrom-b-ähnliche Sequenz findet sich auch in der Kern-DNA eines Nagetiers (Smith et al., 1992). Die Cytochrom-b-ähnlichen Sequenzen von Lutra sind durch das Vorhandensein einer Deletion gekennzeichnet, um eine Leserahmenverschiebung und Terminierungscodons zu verursachen (Abb. 1), durch das Vorhandensein mehrerer Arten von Sequenzen mit geringfügigen Variationen (Abb. 1) und durch die offenbar schnellere Evolutionsrate im Vergleich zur mitochondrialen Cytochrom-b-Sequenz (siehe Fig. 2). Somit scheint die Cytochrom-b-ähnliche Sequenz nicht zu funktionieren und liegt als Pseudoform vor. Wir haben nicht untersucht, ob die Cytochrom-b-ähnliche Sequenz von Lutra in der mitochondrialen DNA oder in der Kern-DNA vorhanden ist.
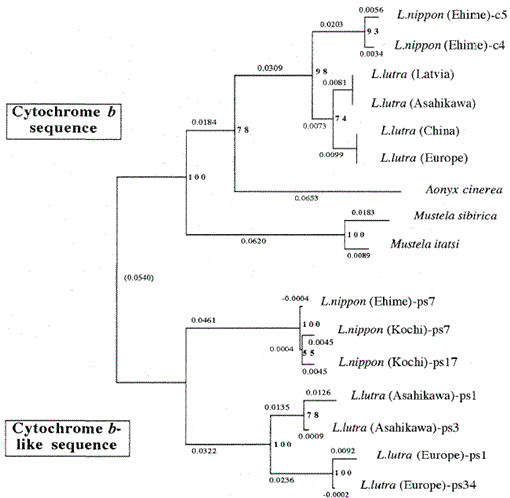
Abb. 2.
Ein phylogenetischer Baum, der aus der Sequenzausrichtung (basierend auf Sequenz 1-224 in Abb. 1) der mitochondrialen Cytochrom b- und Cytochrom b-ähnlichen Sequenzen mit dem Neighbor-Joining-Verfahren. Die Zahlen an jedem Verzweigungspunkt zeigen Bootstrap-Werte (100 Replikationen), die die Cytochrom-b-ähnliche Sequenz darstellen.
Die Nukleotidsequenzen der mitochondrialen Cytochrom-b- und Cytochrom-b-ähnlichen Gene von Lutra nippon sind mit denen von L. lutra (Lettland, China, Europa und Asahikawa), Aonyx cinerea, Mustela sibirica und Mustela itatsi in Abb. 1. Von der Sequenz 1 bis 224 unterschied sich Lutra nippon (C4) von denen von L lutra (Lettland) und L. lutra (China) in 9 bzw. 7 Nukleotiden, die sich alle an der dritten Position eines Codons befanden und als Übergangsdifferenzen A↔ G oder C↔ T identifiziert wurden. Der Unterschied innerhalb von Lutra lutra betrug 4 Nukleotide. Diese Nukleotidsubstitutionen verursachen keinen Aminosäureersatz. Auf der anderen Seite gibt es 6 Nukleotide Unterschied in den beiden kongeneren Wieseln Mustela sibirica und Mustela itatsi in dieser Region. Somit ist der Sequenzunterschied zwischen Lutra nippon und Lutra lutra (7-9 Nukleotide: durchschnittlich 3,6%) größer als der (6 Nukleotide) zwischen den beiden Mustela-Spezies. Dies steht auch im Einklang mit der Idee, dass bei den Mustelidae-Arten ein Unterschied von mehr als 3,5% in der Sequenz des Cytochrom-b-Gens dem Unterschied verschiedener Arten entsprechen kann (Masuda und Yoshida, 1994). Der prozentuale Unterschied zwischen den Nukleotidsequenzen ist in Tabelle 1 dargestellt.
Tabelle 1.
Prozentuale Differenz zwischen den Nukleotidsequenzen (224 bp) gemäß Fig. 1
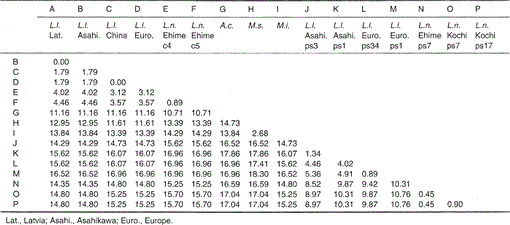
Abbildung 2 zeigt einen phylogenetischen Baum, der aus den in dieser Studie erhaltenen Sequenzdaten mit der Neighbor-Joining-Methode im PHYLIP-Paket Version 3.5c (Felsenstein, 1993) erstellt wurde. Die gleiche Topologie wurde mit der Fitch- und Margoliash-Methode rekonstruiert (Felsenstein, 1993). Die Sequenzen wurden in zwei Hauptcluster unterteilt, einer für die mitochondrialen Cytochrom-b-Gene und der andere für Cytochrom-b-ähnliche Sequenzen. Wie oben erwähnt, wurden die Cytochrom-b-ähnlichen Sequenzen aus den DNAs von Lutra nippon (2 Individuen, aus Kochi und Ehime), L. lutra (Europa) und L. lutra (Asahikawa) amplifiziert. Das Verzweigungsmuster der Cytochrom-b-ähnlichen Sequenzen der beiden Lutra-Spezies war das gleiche wie in den mitochondrialen Cytochrom-b-Genen, aber die Evolutionsrate für die Cytochrom-b-ähnlichen Sequenzen war mindestens zweimal schneller, in Übereinstimmung mit den allgemeinen Eigenschaften von Pseudogenen. Der Baum zeigt auch deutlich, dass es einen großen genetischen Unterschied zwischen Lutra nippon und Lutra lutra gibt, und dieses Ergebnis steht im Einklang mit der jüngsten morphologischen Studie, dass der japanische Flussotter keine Unterart des eurasischen Fischotters Lutra lutra ist, sondern eine eigenständige Art, Lutra nippon (Imaizumi und Yoshiyuki, 1989). Um jedoch einen schlüssigen Beweis für die phylogenetische Beziehung zwischen den beiden Arten zu erhalten, muss die DNA-Sequenz von Lutra nippon bestimmt werden. Dies würde geschehen, wenn die frische Probe des japanischen Flussotters erhalten würde.