RÉSULTATS ET DISCUSSION
Nous avons amplifié le fragment de 307 pb des gènes du cytochrome b mitochondrial des loutres eurasiennes Lutra lutra (Lettonie), L. lutra (Chine), L lutra (Europe) et L. lutra (Asahikawa), de la loutre asiatique à petites griffes Aonyx cinerea et des belettes Mustela sibirica et Mustela itatsi, sous-clonant les produits amplifiés dans pUC18 et a déterminé leurs séquences. Les séquences de L. lutra (Europe) et L. lutra (Asahikawa) étaient entièrement d’accord avec celles de L lutra (Chine) et L lutra (Lettonie), respectivement. Les séquences de deux individus de Mustela itatsi étaient identiques, mais différentes en trois positions de celles rapportées récemment (Masuda et Yoshida, 1994), dans les positions de séquence 1-224 (voir fig. 1).
Fig. 1.
Comparaison des séquences nucléotidiques des séquences mitochondriales de type cytochrome b et cytochrome b de Lutra nippon (Ehime et Kochi) avec celles de L lutra (Lettonie, Chine, Europe et Asahikawa), Aonyx cinerea, Mustela sibirica et M. itatsi. La longueur de la séquence, 224 pb pour L. nippon et 307 pb pour les autres. Plusieurs représentants des séquences de type cytochrome b (ps) sont représentés. Les positions du codon de délétion et de terminaison dans les séquences de type cytochrome b sont mises en boîte. Euro., Europe; Asahi., Asahikawa.
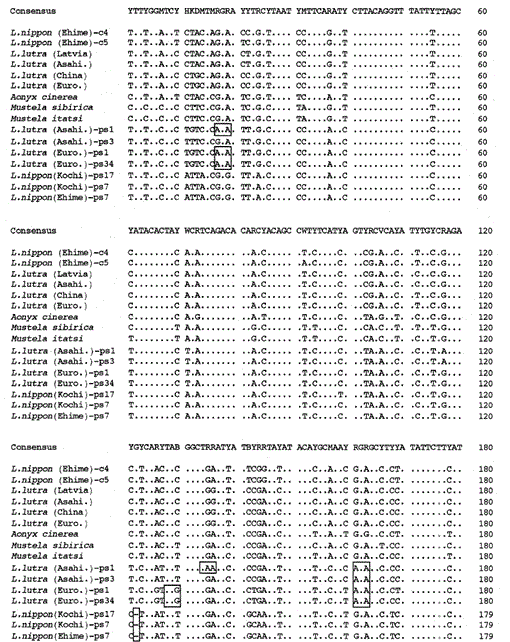
Fig. 1.
Suite.
D’autre part, de nombreuses tentatives ont été faites pour amplifier le fragment d’ADN du gène du cytochrome b de Lutra nippon en utilisant divers échantillons âgés de 10 à 50 ans, tels que le foie, le cœur et la peau conservés dans du formol, le cuir tanné et les poils du spécimen empaillé, mais toutes les tentatives ont été infructueuses.
Nous avons réussi à amplifier le fragment de 224 pb du gène du cytochrome b mitochondrial à partir d’un spécimen de Lutra nippon âgé de 30 ans provenant d’Ehime (Fig. 1). Le séquençage direct des produits amplifiés suggère la présence de plus de deux types de séquences. Les produits ont donc été sous-clonés dans pUC18 et 30 clones ont été séquencés. Parmi eux, neuf clones ont été identifiés de manière ambiguë comme étant la séquence mitochondriale du cytochrome b de Lutra nippon car la séquence était apparemment similaire aux séquences mitochondriales d’autres espèces de Lutra, dont huit clones avaient la même séquence nucléotidique (représentée par la séquence de L. nippon (Ehime)-c4 sur la Fig. 1) mais le reste (la séquence de L. nippon (Ehime)-c5 de la Fig. 1) était différent dans deux positions (21: C→T et 92: T→A). Les onze clones avaient les séquences de type cytochrome b décrites ci-dessous. Les 10 clones restants, de façon inattendue, avaient la séquence presque identique à la séquence du cytochrome b du porc, ce qui suggère qu’une contamination grave de l’ADN s’est produite pendant le stockage du spécimen ressemblant à une momie.
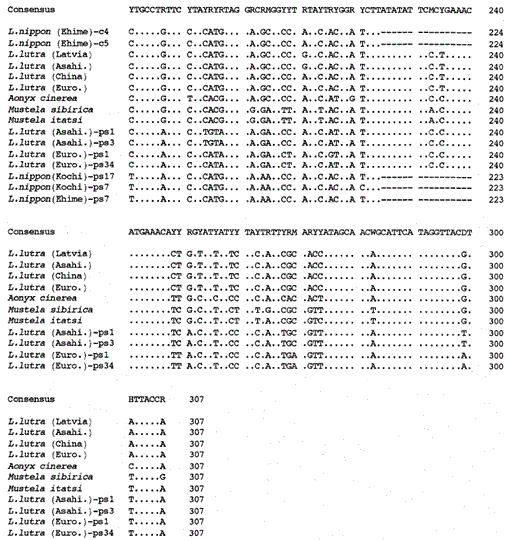
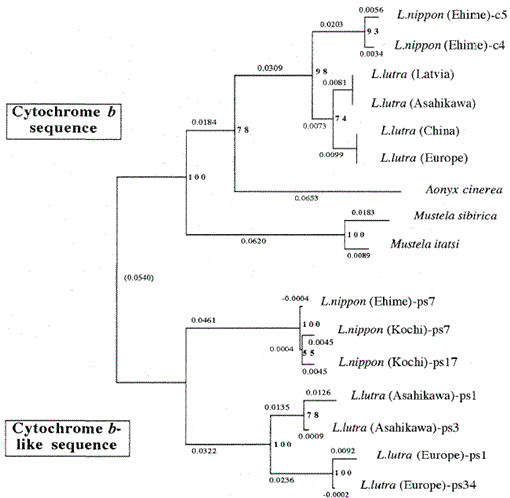
Nous avons constaté que l’ADN d’une momie de Lutra nippon (Ehime) contient une séquence de type cytochrome b. Étonnamment, tous les produits amplifiés d’un autre Lutra nippon de Kochi (spécimen empaillé d’au moins 30 ans) correspondaient aux séquences de type cytochrome b, et aucun fragment de cytochrome b mitochondrial n’a été amplifié. De plus, les fragments de type cytochrome b de 307 pb ont été amplifiés, avec un rendement de 30 à 40% du total des clones, à partir des ADN préparés à partir du foie frais ou des poils des loutres eurasiennes L. lutra (Europe et Asahikawa), mais pas à partir des ADN de L lutra (Lettonie et Chine). Ainsi, la présence d’une séquence de type cytochrome b n’est pas un artefact lors d’un stockage prolongé de l’ADN. La séquence de type cytochrome b se trouve également dans l’ADN nucléaire d’un rongeur (Smith et al., 1992). Les séquences de type cytochrome b de Lutra sont caractérisées par la présence d’une délétion pour provoquer un décalage de trame de lecture et des codons de terminaison (Fig. 1), par la présence de plusieurs types de séquences avec des variations mineures (Fig. 1), et par le taux d’évolution apparemment plus rapide par rapport à la séquence du cytochrome b mitochondrial (voir Fig. 2). Ainsi, la séquence de type cytochrome b semble ne pas fonctionner et se présente sous forme de pseudo-forme. Nous n’avons pas examiné si la séquence de type cytochrome b de Lutra est présente dans l’ADN mitochondrial ou dans l’ADN nucléaire.
Fig. 2.
Un arbre phylogénétique construit à partir de l’alignement des séquences (basé sur la séquence 1-224 de la Fig. 1) des séquences mitochondriales de type cytochrome b et cytochrome b avec la méthode de jointure voisine. Les nombres à chaque point de ramification montrent des valeurs d’amorçage (100 réplications), ps représente la séquence de type cytochrome b.
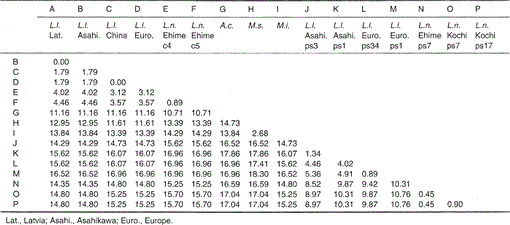
Les séquences nucléotidiques des gènes mitochondriaux de type cytochrome b et cytochrome b de Lutra nippon sont alignées avec celles de L. lutra (Lettonie, Chine, Europe et Asahikawa), Aonyx cinerea, Mustela sibirica et Mustela itatsi, sur la Fig. 1. De la séquence 1 à 224, Lutra nippon (C4) était différent de ceux de L lutra (Lettonie) et L. lutra (Chine) dans 9 et 7 nucléotides, respectivement, tous situés à la troisième position d’un codon et identifiés comme des différences de transition A↔G ou C↔T. La différence au sein de Lutra lutra était de 4 nucléotides. Ces substitutions nucléotidiques ne provoquent aucun remplacement d’acides aminés. D’autre part, il y a une différence de 6 nucléotides chez les deux belettes congénères Mustela sibirica et Mustela itatsi dans cette région. Ainsi la différence de séquence entre Lutra nippon et Lutra lutra (7-9 nucléotides : 3,6% en moyenne) est plus grande que celle (6 nucléotides) entre les deux espèces de Mustela. Ceci est également cohérent avec l’idée que chez les espèces de Mustélidés, une différence de plus de 3,5% dans la séquence du gène du cytochrome b peut correspondre à la différence d’espèces distinctes (Masuda et Yoshida, 1994). La différence en pourcentage entre les séquences nucléotidiques est indiquée dans le tableau 1.
Tableau 1.
Différence en pourcentage entre les séquences nucléotidiques (224 pb) représentées à la Fig. 1
La figure 2 montre un arbre phylogénétique construit à partir des données de séquence obtenues dans cette étude avec la méthode de jointure voisine dans le paquet PHYLIP version 3.5c (Felsenstein, 1993). La même topologie a été reconstruite avec la méthode de Fitch et Margoliash (Felsenstein, 1993). Les séquences ont été divisées en deux groupes principaux, l’un pour les gènes mitochondriaux du cytochrome b et l’autre pour les séquences de type cytochrome b. Comme indiqué ci-dessus, les séquences de type cytochrome b ont été amplifiées à partir des ADN de Lutra nippon (2 individus, de Kochi et Ehime), de L. lutra (Europe) et de L. lutra (Asahikawa). Le schéma de ramification des séquences de type cytochrome b des deux espèces de Lutra était le même que dans les gènes mitochondriaux du cytochrome b, mais le taux d’évolution des séquences de type cytochrome b était au moins deux fois plus rapide, en accord avec les caractéristiques générales des pseudo-gènes. L’arbre montre également clairement qu’il existe une grande différence génétique entre Lutra nippon et Lutra lutra, et ce résultat est cohérent avec l’étude morphologique récente selon laquelle la loutre de rivière japonaise n’est pas une sous-espèce de la loutre d’Eurasie Lutra lutra, mais une espèce distincte, Lutra nippon (Imaizumi et Yoshiyuki, 1989). Cependant, pour obtenir une preuve concluante de la relation phylogénétique entre les deux espèces, une séquence d’ADN plus longue de Lutra nippon doit être déterminée. Cela serait fait lorsque l’échantillon frais de la loutre de rivière japonaise aurait été obtenu.