結果と議論
ユーラシアカワウソLutra lutra(ラトビア)、L.lutra(中国)、L.lutra(ヨーロッパ)、L.lutra(旭川)、アジアの小爪カワウソAonyx cinereaおよびイタチMustela sibiricaおよびmustela itatsiからのミトコンドリアシトクロムb遺伝子の307bp断片を増幅し、puc18で増幅された産物をサブクローニングし、決定した。彼らのシーケンス。 Lの配列。 lutra(ヨーロッパ)とL.lutra(旭川)は,それぞれLultra(中国)とLultra(ラトビア)と完全に一致した。 Mustela itatsiの二人の個体の配列は同一であったが、最近報告されたものとは三つの位置で異なっていた(Masuda and Yoshida、1994)、配列位置1-224(図参照)。 1).
1.
lutra nippon(愛媛、高知)のミトコンドリアのシトクロムbおよびシトクロムb様配列のヌクレオチド配列とL lutra(ラトビア、中国、ヨーロッパ、旭川)、Aonyx cinerea、Mustela sibiricaおよびM.itatsiのヌクレオチド配列の比較。 配列の長さは、L.nipponの場合は224bp、他の場合は307bpである。 シトクロムb様配列(p s)のいくつかの代表を示した。 シトクロムb様配列における欠失および終止コドンの位置はボックス化されている。 ユーロ、ヨーロッパ、朝日。 旭川市
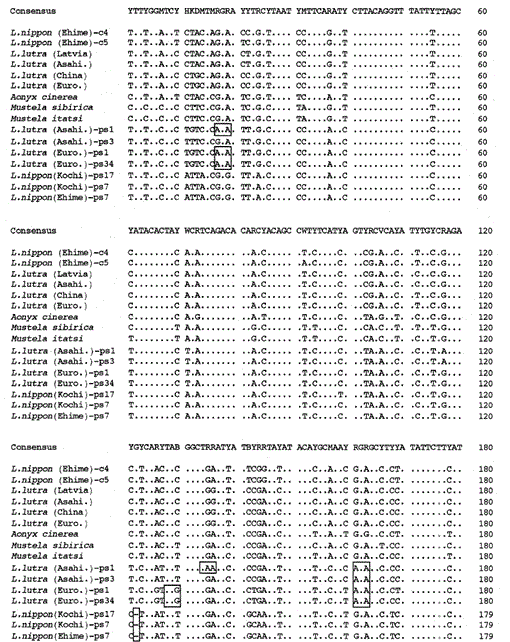
図1.1.1. 1.
続きを読む
一方、ホルマリンに保存された肝臓、心臓、皮膚、日焼けした革、ぬいぐるみの毛など、10-50歳の様々なサンプルを用いて、Lutra nipponのシトクロムb遺伝子のDNA断片を増幅する試みが多く行われたが、すべての試みは失敗した。
愛媛県産のLutra nipponの30歳のミイラ様標本から、ミトコンドリアのシトクロムb遺伝子の224bp断片を増幅することに成功しました(図。 1). 増幅された生成物の直接配列決定は、二つ以上のタイプの配列の存在を示唆した。 したがって、製品はpuc18でサブクローニングされ、30クローンが配列決定された。 その中で、9つのクローンは、他のLutra種のミトコンドリア配列と明らかに類似しているため、lutra nipponのミトコンドリアシトクロムb配列であることがあいまいに同定されました。このうち、8つのクローンは同じ塩基配列を持っていました(図中のL.nippon(Ehime)-c4の配列で表されます)。 1)しかし、残りのもの(図中のL.nippon(愛媛)-c5のシーケンス。 1)は二つの位置(21:C→Tと92:T→A)で異なっていた。 1 1個のクローンは下記のようなシトクロムb様配列を有していた。 残りの10個のクローンは、予想外に、ブタのシトクロムb配列とほぼ同一の配列を有しており、ミイラ様の標本の保存中に深刻なDNA汚染が起こったことを示唆している。
ミイラのようなルトラニッポン(愛媛)のDNAにはシトクロムb様配列が含まれていることがわかりました。 驚くべきことに、高知からの別のLutra nipponからの増幅された産物(少なくとも30歳のぬいぐるみ標本)は、シトクロムb様配列に対応し、ミトコンドリアのシトクロムb断片は増幅されなかった。 さらに、307bpのシトクロムb様断片は、ユーラシアカワウソL.lutra(ヨーロッパと旭川)の新鮮な肝臓または毛から調製したDnaから、全クローンの30-40%の収率で増幅されたが、L lutra(ラトビアと中国)からのDnaからではなかった。 したがって、シトクロムb様配列の存在は、DNAの長期保存中のアーティファクトではない。 シトクロムb様配列は、げっ歯類の核DNA中にも見出される(Smith e t a l., 1992). Lutraのシトクロムb様配列は、読み取りフレームシフトと終了コドンを引き起こす欠失の存在によって特徴付けられる(図。 1)、マイナーなバリエーションを持ついくつかのタイプの配列の存在によって(Fig. 1)、およびミトコンドリアのシトクロムb配列と比較して明らかに速い進化速度によって(図を参照してください。 2). したがって、シトクロムb様配列は機能しないように見え、擬似型として存在する。 Lutraのシトクロムb様配列がミトコンドリアDNA中に存在するか核DNA中に存在するかは調べていない。
2.
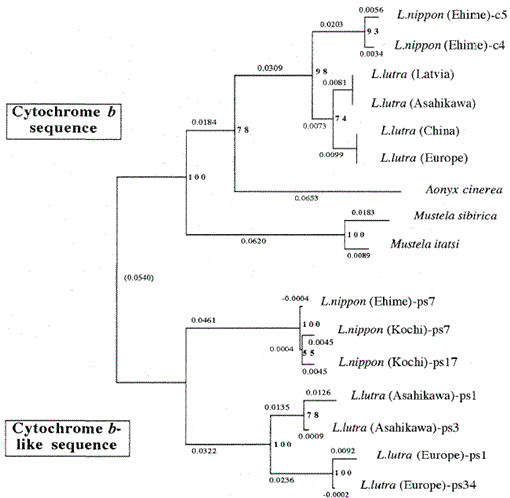
配列アライメントから構築された系統樹(図1-224の配列に基づく。 1)ミトコンドリアのシトクロムbおよびシトクロムb様配列のネイバージョイニング法を用いた。 各分岐点の数字はブートストラップ値(100回の複製)を示し、psはシトクロムb様配列を表す。
lutra nipponのミトコンドリアのシトクロムbおよびシトクロムb様遺伝子の塩基配列は、L.lutra(ラトビア、中国、ヨーロッパ、旭川)、Aonyx cinerea、Mustela sibiricaおよびmustela itatsiのものと整列している。 1. 配列1から224のうち、Lutra nippon(C4)はL lutra(Latvia)およびL.lutra(China)のものとはそれぞれ9および7ヌクレオチドで異なっており、それらはすべてコドンの第三位に位置し、過渡的な違いA≤GまたはC≤Tとして同定された。Lutra lutra内の差は4ヌクレオチドであった。 これらのヌクレオチド置換はアミノ酸置換を引き起こさない。 一方、この地域の二つの同族イタチMustela sibiricaとMustela itatsiには6つのヌクレオチド差がある。 したがって、Lutra nipponとLutra lutraの配列差(7-9ヌクレオチド:平均で3.6%)は、二つのMustela種の間のそれ(6ヌクレオチド)よりも大きい。 これはまた、Mustelidae種では、シトクロムb遺伝子の配列の3.5%以上の違いが異なる種の違いに対応する可能性があるという考えと一致している(Masuda and Yoshida、1994)。 塩基配列間の割合差を表1に示す。
表1.
図に示す塩基配列間の割合差(224bp)。 1
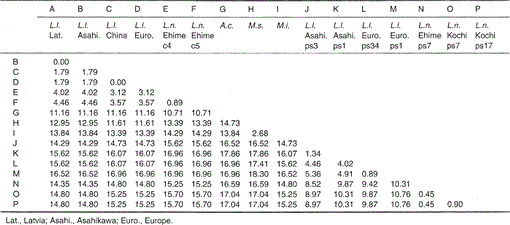
図2は、PHYLIPパッケージバージョン3.5c(Felsenstein,1993)のNeighbor-Joining法を用いて本研究で得られた配列データから構築された系統樹を示しています。 同じトポロジーは、Fitch and Margoliash法(Felsenstein、1993)で再構成されました。 配列はミトコンドリアのシトクロムb遺伝子とシトクロムb様配列の二つの主要なクラスターに分けられた。 上記のように、シトクロムb様配列は、Lutra nippon(高知と愛媛の2個体)、L.lutra(ヨーロッパ)およびL.lutra(旭川)のDnaから増幅された。 二つのLutra種のシトクロムb様配列の分岐パターンはミトコンドリアのシトクロムb遺伝子と同じであったが,シトクロムb様配列の進化速度は擬似遺伝子の一般的な特性と一致して少なくとも二倍速であった。 この木はまた、Lutra nipponとLutra lutraの間に大きな遺伝的違いがあることを明らかに示しており、この結果は、日本のカワウソがユーラシアカワウソLutra lutraの亜種ではなく、異 しかし,両種間の系統発生関係の決定的な証拠を得るためには,Lutranipponのより長いDNA配列を決定する必要がある。 これは、日本の川カワウソの新鮮なサンプルが得られたときに行われます。