RISULTATI E DISCUSSIONE
Abbiamo amplificato la 307 bp frammento di mitocondriale citocromo b geni da la lontra Eurasiatica (Lutra Lutra) (Lettonia), L. lutra (Cina), L lutra (Europa) e L. lutra (Asahikawa), l’Asiatica dalle piccole unghie otter Aonyx cinerea e la donnola Mustela Mustela sibirica e itatsi, subcloned i prodotti amplificati in pUC18 e determinato le loro sequenze. Le sequenze di L. lutra (Europa) e L. lutra (Asahikawa) concordano pienamente con quelli di L lutra (Cina) e L lutra (Lettonia), rispettivamente. Le sequenze di due individui di Mustela itatsi erano identiche, ma diverse in tre posizioni da quelle riportate di recente (Masuda e Yoshida, 1994), nelle posizioni di sequenza 1-224 (vedi Fig. 1).
Fig. 1.
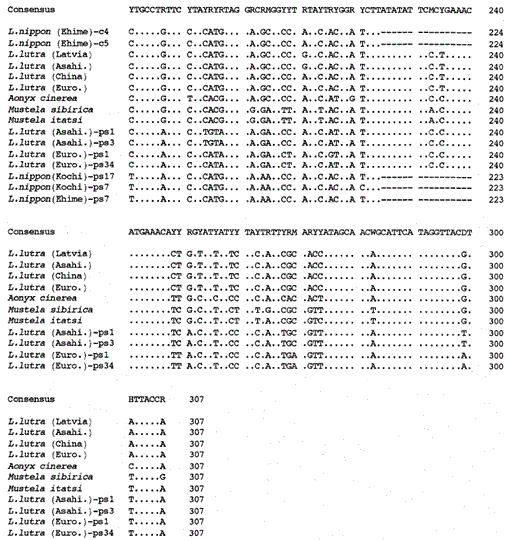
Confronto delle sequenze nucleotidiche del citocromo b mitocondriale e delle sequenze simili al citocromo b di Lutra nippon (Ehime e Kochi) con quelle di L lutra (Lettonia, Cina, Europa e Asahikawa), Aonyx cinerea, Mustela sibirica e M. itatsi. La lunghezza della sequenza, 224 bp per L. nippon e 307 bp per gli altri. Vengono mostrati diversi rappresentanti delle sequenze simili al citocromo b (ps). Le posizioni del codone di eliminazione e terminazione nelle sequenze simili al citocromo b sono inscatolate. Euro., Europa; Asahi., Asahikawa.
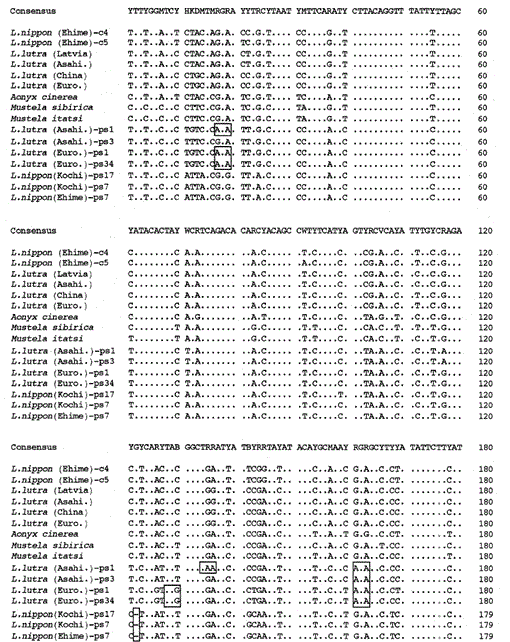
Fig. 1.
Seguito.
d’altra parte, molti tentativi sono stati fatti per amplificare il frammento di DNA del gene citocromo b di Lutra nippon utilizzando vari 10-50-anno-vecchi campioni, come il fegato, il cuore e la pelle conservati in formalina, la pelle conciata e i peli del ripieno campione, ma tutti i tentativi sono falliti.
Siamo riusciti ad amplificare il frammento di 224 bp del gene del citocromo b mitocondriale da un esemplare di Lutra nippon di 30 anni di Ehime (Fig. 1). Il sequenziamento diretto dei prodotti amplificati ha suggerito la presenza di più di due tipi di sequenza. Pertanto i prodotti sono stati succlonati in pUC18 e 30 cloni sono stati sequenziati. Tra questi, nove cloni sono stati identificati ambiguamente come la sequenza mitocondriale del citocromo b di Lutra nippon poiché la sequenza era apparentemente simile alle sequenze mitocondriali di altre specie di Lutra, di cui otto cloni avevano la stessa sequenza nucleotidica (rappresentata dalla sequenza di L. nippon (Ehime)-c4 in Fig. 1) ma il restante (la sequenza di L. nippon (Ehime)-c5 in Fig. 1) era diverso in due posizioni (21: C→T e 92: T→A). Gli undici cloni avevano le sequenze simili al citocromo b come descritto di seguito. I restanti 10 cloni, inaspettatamente, avevano la sequenza quasi identica alla sequenza del citocromo b del maiale, suggerendo una grave contaminazione del DNA avvenuta durante la conservazione del campione simile alla mummia.
Abbiamo scoperto che il DNA di un Lutra nippon simile alla mummia (Ehime) contiene una sequenza simile al citocromo B. Sorprendentemente, tutti i prodotti amplificati da un altro Lutra nippon di Kochi (almeno 30-year-old campione farcito) corrispondevano alle sequenze simili al citocromo b, e nessun frammento mitocondriale del citocromo b è stato amplificato. Inoltre, i frammenti simili al citocromo b di 307 bp sono stati amplificati, con una resa del 30-40% dei cloni totali, dai DNA preparati dal fegato fresco o dai peli delle lontre eurasiatiche L. lutra (Europa e Asahikawa), ma non dai DNA di L lutra (Lettonia e Cina). Quindi la presenza di sequenza simile al citocromo b non è un artefatto durante una lunga conservazione del DNA. La sequenza simile al citocromo b si trova anche nel DNA nucleare in un roditore (Smith et al., 1992). Le sequenze simili al citocromo b di Lutra sono caratterizzate dalla presenza di una delezione per causare uno spostamento del frame di lettura e codoni di terminazione (Fig. 1), dalla presenza di diversi tipi di sequenze con variazioni minori (Fig. 1), e dal tasso evolutivo apparentemente più veloce rispetto alla sequenza mitocondriale del citocromo b (vedi Fig. 2). Quindi la sequenza simile al citocromo b sembra non funzionare ed è presente come una pseudo-forma. Non abbiamo esaminato se la sequenza di Lutra simile al citocromo b sia presente nel DNA mitocondriale o nel DNA nucleare.
Fig. 2.
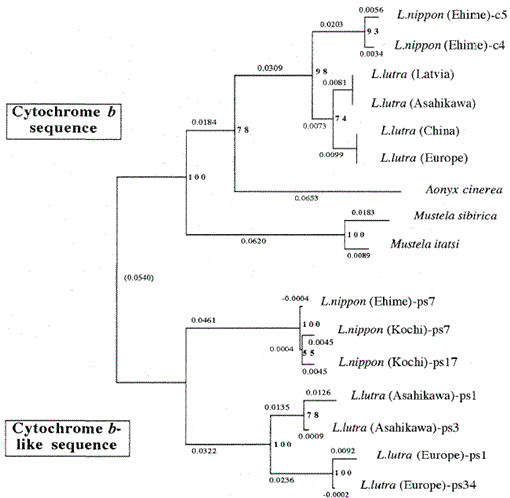
Un albero filogenetico costruito dall’allineamento della sequenza (basato sulla sequenza 1-224 in Fig. 1) delle sequenze mitocondriali del citocromo b e del citocromo b-like con il metodo Neighbor-Joining. I numeri in ogni punto di ramificazione mostrano valori di bootstrap (100 repliche), ps rappresenta la sequenza simile al citocromo B.
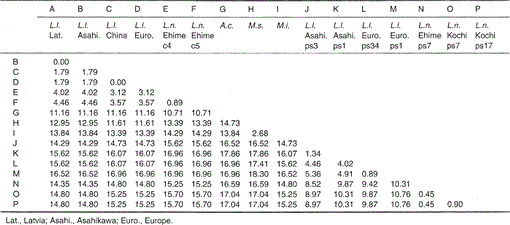
Le sequenze nucleotidiche dei geni mitocondriali del citocromo b e del citocromo b-like di Lutra nippon sono allineate con quelle di L. lutra (Lettonia, Cina, Europa e Asahikawa), Aonyx cinerea, Mustela sibirica e Mustela itatsi, in Fig. 1. Della sequenza da 1 a 224, Lutra nippon (C4) era diverso da quelli di L lutra (Lettonia) e L. lutra (Cina) in 9 e 7 nucleotidi, rispettivamente, tutti situati nella terza posizione di un codone e identificati come differenze di transizione A↔G o C↔T. La differenza all’interno di Lutra lutra era di 4 nucleotidi. Queste sostituzioni nucleotidiche non causano alcuna sostituzione di aminoacidi. D’altra parte, ci sono 6 nucleotidi differenza nelle due donnole congeneriche Mustela sibirica e Mustela itatsi in questa regione. Quindi la differenza di sequenza tra Lutra nippon e Lutra lutra (7-9 nucleotidi: 3,6% in media) è maggiore di quella (6 nucleotidi) tra le due specie di Mustela. Questo è anche coerente con l’idea che nelle specie Mustelidae, più del 3,5% di differenza nella sequenza del gene del citocromo b può corrispondere alla differenza di specie distinte (Masuda e Yoshida, 1994). La differenza percentuale tra le sequenze nucleotidiche è riportata nella Tabella 1.
Tabella 1.
Differenza percentuale tra le sequenze nucleotidiche (224 bp) mostrato in Fig. 1
La figura 2 mostra un albero filogenetico costruito dai dati di sequenza ottenuti in questo studio con il metodo di unione vicina nel pacchetto PHYLIP versione 3.5 c (Felsenstein, 1993). La stessa topologia è stata ricostruita con il metodo Fitch e Margoliash (Felsenstein, 1993). Le sequenze sono state divise in due cluster principali, uno per i geni mitocondriali del citocromo b e l’altro per le sequenze simili al citocromo B. Come detto sopra, le sequenze simili al citocromo b sono state amplificate dai DNA di Lutra nippon (2 individui, da Kochi e Ehime), L. lutra (Europa) e L. lutra (Asahikawa). Il modello di ramificazione delle sequenze simili al citocromo b delle due specie di Lutra era lo stesso dei geni mitocondriali del citocromo b, ma il tasso evolutivo per le sequenze simili al citocromo b era almeno due volte più veloce, in accordo con le caratteristiche generali degli pseudo-geni. L’albero, inoltre, mostra chiaramente che c’è una grande differenza genetica tra Lutra nippon e Lutra lutra), e questo risultato è coerente con il recente studio morfologico che i Giapponesi fiume lontra non è una sottospecie di lontra Eurasiatica (Lutra Lutra), ma di una specie distinta, Lutra nippon (Imaizumi e Yoshiyuki, 1989). Tuttavia, per ottenere una prova conclusiva della relazione filogenetica tra le due specie, è necessario determinare una sequenza di DNA più lunga del Lutra nippon. Questo sarebbe stato fatto quando è stato ottenuto il campione fresco della lontra di fiume giapponese.