wyniki i dyskusja
wzmocniliśmy fragment mitochondrialnego genu cytochromu b o długości 307 bp od wydr Lutra lutra (Łotwa), L. lutra (Chiny), l lutra (Europa) I L. lutra (Asahikawa), Wydry azjatyckiej aonyx cinerea oraz łasic Mustela sibirica i Mustela itatsi, subklonując produkty amplifikowane w puc18 i określili ich sekwencje. Sekwencje L. lutra (Europa) I L. lutra (Asahikawa) w pełni zgodziły się z L lutra (Chiny) i L lutra (Łotwa), odpowiednio. Sekwencje dwóch osobników Mustela itatsi były identyczne, ale różniły się w trzech pozycjach od Ostatnio zgłoszonych (Masuda and Yoshida, 1994), w pozycjach sekwencji 1-224 (patrz ryc. 1).
1.
porównanie sekwencji nukleotydowych mitochondrialnego cytochromu b i podobnych do cytochromu B sekwencji Lutra nippon (Ehime i Kochi) z sekwencjami L lutra (Łotwa, Chiny, Europa i Asahikawa), aonyx cinerea, Mustela sibirica i M. itatsi. Długość sekwencji, 224 bp dla L. nippon I 307 bp dla pozostałych. Przedstawiono kilku przedstawicieli sekwencji podobnych do cytochromu b (ps). Pozycje kodonu delecji i zakończenia w sekwencjach podobnych do cytochromu b są blokowane. Euro., Europa; Asahi., Asahikawa.
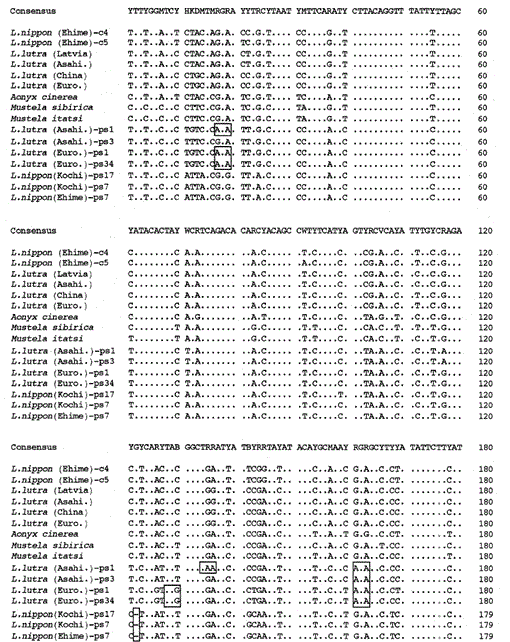
Fig. 1.
ciąg dalszy.
z drugiej strony podjęto wiele prób amplifikacji fragmentu DNA genu cytochromu B Lutry nippon przy użyciu różnych próbek sprzed 10-50 lat, takich jak wątroba, serce i skóra zachowane w formalinie, garbowana skóra i włosy wypchanych osobników, ale wszystkie próby zakończyły się niepowodzeniem.
udało nam się amplifikować 224 bp fragment mitochondrialnego genu cytochromu b z 30-letniego okazu przypominającego mumię Lutra nippon z Ehime (Fig. 1). Bezpośrednie sekwencjonowanie amplifikowanych produktów sugerowało obecność więcej niż dwóch rodzajów sekwencji. Dlatego produkty zostały sklonowane w pUC18 i zsekwencjonowano 30 klonów. Wśród nich dziewięć klonów zostało niejednoznacznie zidentyfikowanych jako mitochondrialna Sekwencja cytochromu B Lutra nippon, ponieważ sekwencja ta była najwyraźniej podobna do sekwencji mitochondrialnych innych gatunków Lutra, z których osiem klonów miało tę samą sekwencję nukleotydową (reprezentowaną przez sekwencję L. nippon (Ehime)-C4 na Fig. 1), ale pozostała (Sekwencja L. nippon (Ehime)-c5 na Fig. 1) różnił się w dwóch pozycjach (21: C→T i 92: T→A). Jedenaście klonów miało sekwencje podobne do cytochromu b, jak opisano poniżej. Pozostałe 10 klonów miało nieoczekiwanie prawie identyczną sekwencję z sekwencją świńskiego cytochromu b, co sugeruje poważne zanieczyszczenie DNA podczas przechowywania mumii.
odkryliśmy, że DNA z mumiowatej Lutry nippon (Ehime) zawiera sekwencję podobną do cytochromu B. Co zaskakujące, wszystkie amplifikowane produkty z innej Lutry nippon z Kochi (co najmniej 30-letni wypchany okaz) odpowiadały sekwencjom podobnym do cytochromu b i żaden mitochondrialny fragment cytochromu b nie został amplifikowany. Ponadto fragmenty cytochromu b-podobne do 307 bp zostały amplifikowane, z wydajnością 30-40% wszystkich klonów, z DNAs przygotowanych ze świeżej wątroby lub włosów wydr euroazjatyckich L. lutra (Europa i Asahikawa), ale nie z DNAs z L lutra (Łotwa i Chiny). Tak więc obecność sekwencji podobnej do cytochromu b nie jest artefaktem podczas długiego przechowywania DNA. Sekwencja podobna do cytochromu b znajduje się również w jądrowym DNA Gryzonia (Smith i wsp ., 1992). Sekwencje Lutra podobne do cytochromu b charakteryzują się obecnością delecji powodującej przesunięcie ramki odczytu i kodony zakończenia (rys. 1), poprzez obecność kilku rodzajów sekwencji z niewielkimi zmianami (rys. 1), oraz przez pozornie szybszą ewolucję w porównaniu z mitochondrialną sekwencją cytochromu b (patrz ryc. 2). Tak więc Sekwencja podobna do cytochromu b wydaje się nie funkcjonować i występuje jako pseudo-forma. Nie zbadano, czy sekwencja Lutra podobna do cytochromu b jest obecna w mitochondrialnym DNA, czy w DNA jądrowym.
2.
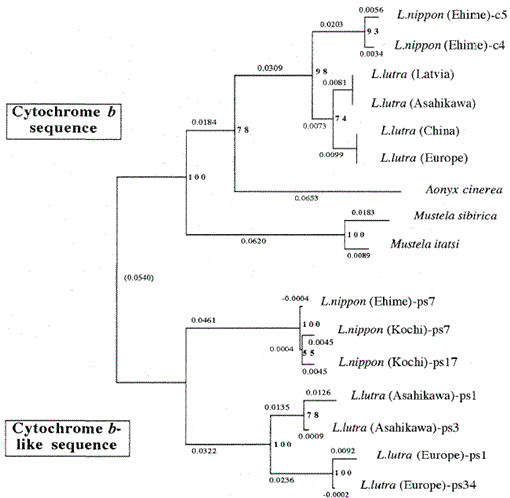
drzewo filogenetyczne zbudowane z wyrównania sekwencji (na podstawie sekwencji 1-224 na Fig. 1) mitochondrialnych sekwencji cytochromu b i cytochromu B metodą łączenia z sąsiadami. Liczby w każdym punkcie rozgałęzienia pokazują wartości bootstrap (100 replikacji), ps reprezentuje sekwencję podobną do cytochromu B.
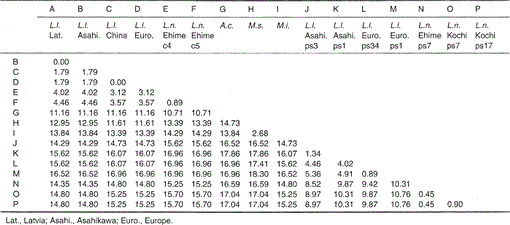
sekwencje nukleotydowe mitochondrialnych genów cytochromu B i genów podobnych do cytochromu B Lutra nippon są zgodne z sekwencjami genów L. lutra (Łotwa, Chiny, Europa i Asahikawa), aonyx cinerea, Mustela sibirica i Mustela itatsi, na Fig. 1. Z sekwencji od 1 do 224, Lutra nippon (C4) różniła się od sekwencji l lutra (Łotwa) i L. lutra (Chiny) odpowiednio w 9 i 7 nukleotydach, z których wszystkie znajdowały się w trzeciej pozycji kodonu i zidentyfikowano jako różnice przejściowe A↔G lub C↔T. różnica w obrębie Lutra lutra wynosiła 4 nukleotydy. Te podstawienia nukleotydów nie powodują wymiany aminokwasów. Z drugiej strony, Istnieje 6 różnic nukleotydów w dwóch podobnych łasicach Mustela sibirica i Mustela itatsi w tym regionie. Tak więc różnica sekwencji pomiędzy Lutra nippon i Lutra lutra (7-9 nukleotydów: średnio 3,6%) jest większa niż ta (6 nukleotydów) pomiędzy dwoma gatunkami Mustela. Jest to również zgodne z tezą, że u gatunków Mustelidae ponad 3,5% różnicy w sekwencji genu cytochromu b może odpowiadać różnicy odrębnych gatunków (Masuda and Yoshida, 1994). Procentową różnicę między sekwencjami nukleotydowymi przedstawiono w tabeli 1.
Tabela 1.
procentowa różnica pomiędzy sekwencjami nukleotydowymi (224 bp) pokazana na Fig. 1
Rysunek 2 przedstawia drzewo filogenetyczne zbudowane na podstawie danych o sekwencji uzyskanych w tym badaniu za pomocą metody łączenia sąsiadów w pakiecie PHYLIP w wersji 3.5 c (Felsenstein, 1993). Tę samą topologię zrekonstruowano metodą Fitcha i Margoliasha (Felsenstein, 1993). Sekwencje zostały podzielone na dwa główne klastry, jeden dla mitochondrialnych genów cytochromu b, a drugi dla sekwencji podobnych do cytochromu B. Jak stwierdzono powyżej, sekwencje podobne do cytochromu b zostały amplifikowane z dna Lutra nippon (2 osobniki z Kochi i Ehime), L. lutra (Europa) I L. lutra (Asahikawa). Wzór rozgałęzień sekwencji cytochromu b u dwóch gatunków Lutra był taki sam jak w mitochondrialnych genach cytochromu b, ale tempo ewolucji sekwencji cytochromu b było co najmniej dwa razy szybsze, zgodnie z ogólną charakterystyką pseudogen. Drzewo wyraźnie pokazuje również, że istnieje duża różnica genetyczna między Lutra nippon I Lutra lutra, a wynik ten jest zgodny z niedawnymi badaniami morfologicznymi, że japońska Wydra rzeczna nie jest podgatunkiem Euroazjatyckiej wydry Lutra lutra, ale odrębnym gatunkiem, Lutra nippon (Imaizumi i Yoshiyuki, 1989). Jednakże, aby uzyskać rozstrzygający dowód na filogenetyczne pokrewieństwo między dwoma gatunkami, należy określić dłuższą sekwencję DNA Lutra nippon. Miało to miejsce po pozyskaniu świeżej próbki japońskiej wydry rzecznej.