eredmények és megbeszélés
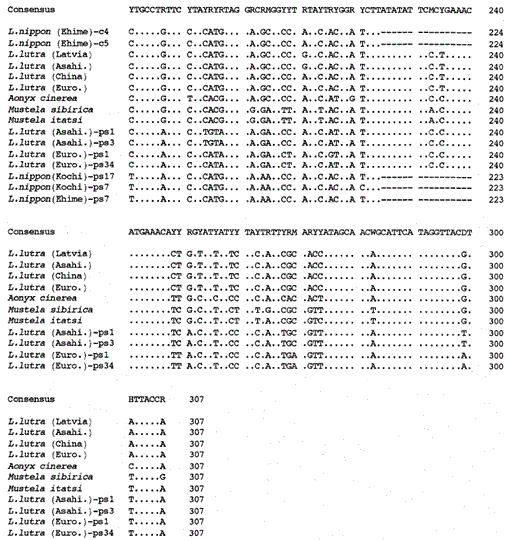
amplifikáltuk az Eurázsiai vidra Lutra lutra (Lettország), L. lutra (Kína), l lutra (Európa) és L. lutra (Asahikawa), az ázsiai kiskarmos vidra Aonyx cinerea és a menyét Mustela sibirica és Mustela itatsi mitokondriális citokróm B génjeinek 307 bp-s fragmentumát, az amplifikált termékeket alklónoztuk az Aonyx cinerea puc18 és meghatározta azok szekvenciáit. Az L szekvenciái. lutra (Európa) és L. lutra (Asahikawa) teljesen egyetértett a L lutra (Kína) és L lutra (Lettország). A Mustela itatsi két egyedének szekvenciái azonosak voltak, de három pozícióban különböztek a közelmúltban jelentettektől (Masuda and Yoshida, 1994), az 1-224.szekvencia pozíciókban (Lásd az ábrát. 1).
ábra. 1.
a Lutra nippon (Ehime és Kochi) mitokondriális citokróm b és citokróm b-szerű szekvenciáinak nukleotidszekvenciáinak összehasonlítása az L lutra (Lettország, Kína, Európa és Asahikawa), az Aonyx cinerea, Mustela sibirica és M. itatsi nukleotidszekvenciáival. A szekvencia hossza, L. nippon esetében 224 bp, a többieknél 307 bp. A citokróm b-szerű szekvenciák (ps) több képviselője látható. A deléciós és terminációs kodon helyzete a citokróm b-szerű szekvenciákban dobozos. Euró., Európa; Asahi., Asahikawa.
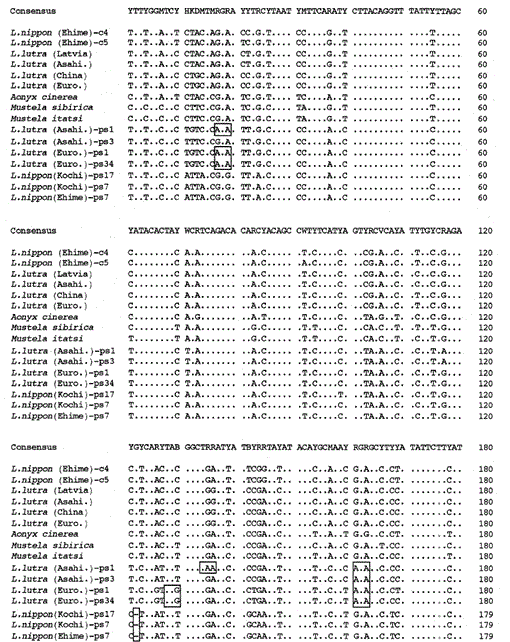
Fig. 1.
Folytatás.
másrészt számos kísérlet történt a Lutra nippon citokróm B génjének DNS-fragmensének amplifikálására különféle 10-50 éves minták felhasználásával, mint például a formalinban megőrzött máj, szív és bőr, a cserzett bőr és a töltött minta szőrszálai, de minden kísérlet sikertelen volt.
sikerült amplifikálni a mitokondriális citokróm B gén 224 bp fragmensét egy 30 éves múmiaszerű mintából Lutra nippon tól től Ehime (ábra. 1). Az amplifikált termékek közvetlen szekvenálása több mint kétféle szekvencia jelenlétére utalt. Ezért a termékeket pUC18-ban szubklónoztuk, és 30 klónt szekvenáltunk. Közülük kilenc klónt egyértelműen azonosítottak a Lutra nippon mitokondriális citokróm B szekvenciájának, mivel a szekvencia nyilvánvalóan hasonló volt más Lutra Fajok mitokondriális szekvenciáihoz, amelyek közül nyolc klónnak ugyanaz volt a nukleotidszekvenciája (amelyet a L. nippon (Ehime)-C4 ábrán. 1) de a fennmaradó (L. nippon (Ehime)-C5 sorozata az ábrán. 1) két pozícióban különbözött (21: C, t és 92: t, a). A tizenegy klónnak citokróm b – szerű szekvenciái voltak az alábbiakban leírtak szerint. A maradék 10 klónok, váratlanul, szinte azonos szekvenciával rendelkezett a sertés citokróm b szekvenciájával, ami arra utal, hogy a múmia-szerű minta tárolása során súlyos DNS-szennyeződés történt.
azt találtuk, hogy a múmia-szerű Lutra nippon (Ehime) DNS-e citokróm b-szerű szekvenciát tartalmaz. Meglepő módon egy másik Lutra nippon Kochi (legalább 30 éves töltött minta) összes amplifikált terméke megfelelt a citokróm b-szerű szekvenciáknak, és nem volt mitokondriális citokróm b fragmens amplifikálva. Ezenkívül a 307 bp citokróm b-szerű fragmenseket az összes klón 30-40% – os hozamával amplifikálták az Eurázsiai vidra L. lutra (Európa és Asahikawa) friss májából vagy szőréből készített DNS-ekből, de nem az L lutra (Lettország és Kína) DNS-éből. Így a citokróm b-szerű szekvencia jelenléte nem műtárgy a DNS hosszú tárolása során. A citokróm b-szerű szekvencia a nukleáris DNS-ben is megtalálható egy rágcsálóban (Smith et al., 1992). A Lutra citokróm b-szerű szekvenciáit a deléció jelenléte jellemzi, amely leolvasási keret eltolódást és terminációs kodonokat okoz (ábra. 1), a jelenléte többféle szekvenciák kisebb eltérések (ábra. 1), valamint a látszólag gyorsabb evolúciós sebességgel a mitokondriális citokróm B szekvenciához képest (lásd az ábrát. 2). Így a citokróm b-szerű szekvencia úgy tűnik, hogy nem működik, és pszeudo-formában van jelen. Nem vizsgáltuk, hogy a Lutra citokróm b-szerű szekvenciája jelen van-e a mitokondriális DNS-ben vagy a nukleáris DNS-ben.
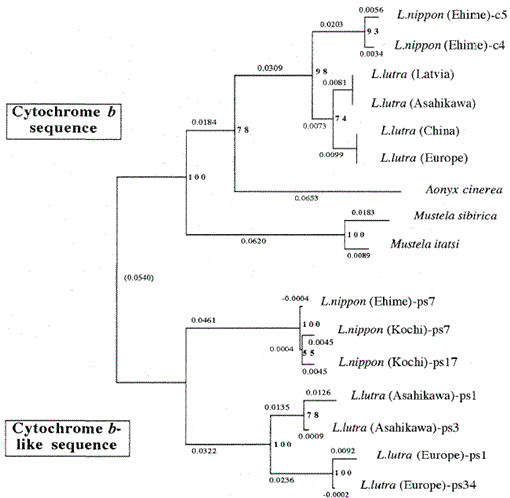
ábra. 2.
a filogenetikai fa épített a szekvencia igazítás (alapján szekvencia 1-224 ábrán. 1) a mitokondriális citokróm B és citokróm b-szerű szekvenciák a szomszéd-csatlakozási módszerrel. Az egyes elágazási pontok számai bootstrap értékeket mutatnak (100 replikáció), a PS a citokróm b-szerű szekvenciát képviseli.
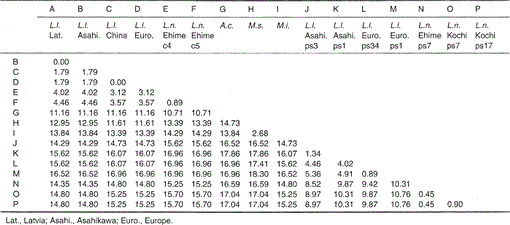
a Lutra nippon mitokondriális citokróm b és citokróm b-szerű génjeinek nukleotidszekvenciái összhangban vannak az L. lutra (Lettország, Kína, Európa és Asahikawa), az Aonyx cinerea, a Mustela sibirica és a Mustela itatsi nukleotidszekvenciáival. 1. Az 1-224.szekvencia közül a Lutra Nippon (C4) 9, illetve 7 nukleotidban különbözött az L lutra (Lettország), illetve az L. lutra (Kína) nukleotidjaitól, amelyek mindegyike a kodon harmadik helyén helyezkedett el, és átmeneti különbségként azonosították A. G. vagy C. T. a Lutra lutra-n belül a különbség 4 nukleotid volt. Ezek a nukleotid szubsztitúciók nem okoznak aminosavpótlást. Másrészt, van 6 nukleotid különbség a két congeneric menyét Mustela sibirica és Mustela itatsi ebben a régióban. Így a Lutra nippon és a Lutra lutra (7-9 nukleotid: átlagosan 3,6%) közötti szekvenciakülönbség nagyobb, mint a két Mustela faj közötti (6 nukleotid). Ez összhangban van azzal az elképzeléssel is, hogy a Mustelidae fajokban a citokróm B gén szekvenciájának több mint 3,5% – os különbsége megfelelhet a különböző fajok különbségének (Masuda and Yoshida, 1994). A nukleotidszekvenciák közötti százalékos különbséget az 1.táblázat mutatja.
1.táblázat.
százalékos különbség a nukleotidszekvenciák (224 bp) ábrán látható. 1
a 2. ábra egy filogenetikai fát mutat be, amelyet az ebben a tanulmányban a szomszéd-csatlakozási módszerrel kapott szekvenciaadatokból állítottak össze a PHYLIP csomag 3.5 c verziójában (Felsenstein, 1993). Ugyanezt a topológiát rekonstruálták a Fitch és Margoliash módszerrel (Felsenstein, 1993). A szekvenciákat két fő klaszterre osztottuk, az egyik a mitokondriális citokróm B génekre, a másik a citokróm b-szerű szekvenciákra vonatkozik. Mint fentebb említettük, a citokróm b-szerű szekvenciákat a Lutra nippon (2 egyed, Kochi és Ehime), a L. lutra (Európa) és a L. lutra (Asahikawa) DNS-éből amplifikáltuk. A két Lutra faj citokróm b-szerű szekvenciáinak elágazási mintája megegyezett a mitokondriális citokróm B génekkel, de a citokróm b-szerű szekvenciák evolúciós sebessége legalább kétszer gyorsabb volt, összhangban az álgének általános jellemzőivel. A fa azt is világosan mutatja, hogy nagy genetikai különbség van a Lutra nippon és a Lutra lutra között, és ez az eredmény összhangban van azzal a közelmúltbeli morfológiai vizsgálattal, amely szerint a japán folyami vidra nem az Eurázsiai vidra Lutra lutra alfaja, hanem egy különálló faj, a Lutra Nippon (Imaizumi and Yoshiyuki, 1989). Ahhoz azonban, hogy meggyőző bizonyítékot kapjunk a két faj filogenetikai kapcsolatáról, meg kell határozni a Lutra nippon hosszabb DNS-szekvenciáját. Erre akkor kerül sor, amikor a japán folyami vidra friss mintáját megszerezték.