RESULTADOS E DISCUSSÃO
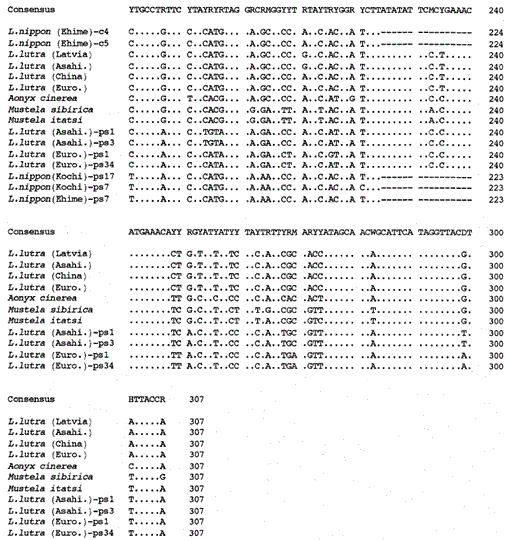
temos ampliado o 307 bp fragmento de mitocondrial citocromo b genes da Eurásia lontras (Lutra lutra (Letónia), L. lutra (China), L lutra (Europa) e L. lutra (Asahikawa), a lontra Asiática pequenas garras Ônix cinerea e as doninhas Mustela sibirica e Mustela itatsi, subcloned produtos amplificados em pUC18 e determinou suas sequências. As sequências de L. lutra (Europa) e L. lutra (Asahikawa) concordaram completamente com os de l lutra (China) e l lutra (Letônia), respectivamente. As sequências de dois indivíduos de Mustela itatsi eram idênticas, mas diferentes em três posições das relatadas recentemente (Masuda e Yoshida, 1994), nas posições de sequência 1-224 (ver Fig. 1).
Fig. 1.
a Comparação das sequências nucleotídicas do mitocondrial citocromo b e citocromo b-como sequências de Lutra nippon (Ehime e Kochi) com os de L lutra (Letónia, a China, a Europa e Asahikawa), Ônix cinerea, Mustela sibirica e M. itatsi. O comprimento da sequência, 224 bp para L. nippon e 307 bp para os outros. Vários representantes das sequências semelhantes ao citocromo b (ps) são mostrados. As posições do códon de deleção e terminação nas sequências semelhantes ao citocromo b são encaixotadas. Euro., Europa; Asahi., Asahikawa.
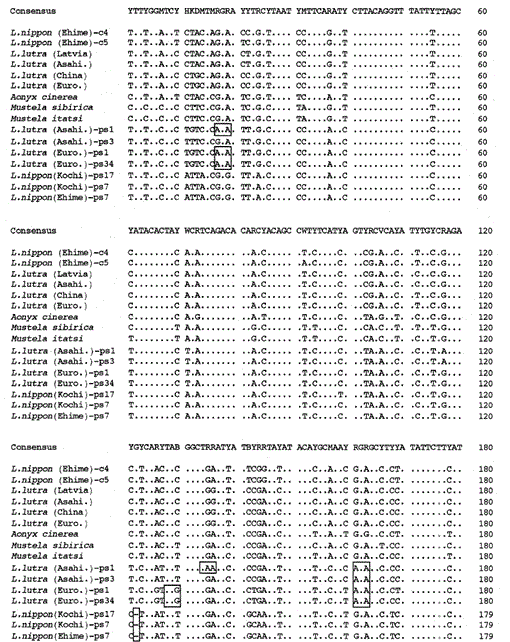
Fig. 1.
continuação.
por outro lado, foram feitas muitas tentativas para amplificar o fragmento de DNA do gene citocromo b de Lutra nippon usando vários 10-50 anos, amostras, tais como o fígado, coração e pele preservados em formol, o couro curtido e os cabelos da recheado de amostra, mas todas as tentativas foram infrutíferas.
conseguimos amplificar o fragmento de 224 bp do gene do citocromo B mitocondrial de um espécime semelhante a uma múmia de 30 anos de Lutra nippon de Ehime (Fig. 1). O sequenciamento direto dos produtos amplificados sugeriu a presença de mais de dois tipos de sequência. Portanto, os produtos foram subclonados na pUC18 e 30 clones foram sequenciados. Entre eles, nove clones foram ambiguamente identificado para ser mitocondrial citocromo b sequência de Lutra nippon uma vez que a seqüência foi aparentemente semelhantes para mitocondrial sequências de Lutra outras espécies, das quais oito clones tinham a mesma sequência dos nucleotídeos (representada pela sequência de L. nippon (Ehime)-c4 na Fig. 1) mas o restante (a sequência de L. nippon (Ehime)-c5 na Fig. 1) foi diferente em duas posições (21: C→T e 92: T→A). Os onze clones tinham as sequências semelhantes ao citocromo b, conforme descrito abaixo. Os 10 clones restantes, inesperadamente, tinham a sequência quase idêntica à sequência do citocromo B do porco, sugerindo que alguma contaminação séria do DNA ocorreu durante o armazenamento da amostra semelhante à múmia.
descobrimos que o DNA de um Lutra Nippon semelhante a uma múmia (Ehime) contém uma sequência semelhante ao citocromo B. Surpreendentemente, todos os produtos amplificados de outro Lutra nippon de Kochi (pelo menos espécime recheado de 30 anos) correspondiam às sequências semelhantes ao citocromo b, e nenhum fragmento do citocromo B mitocondrial foi amplificado. Além disso, o citocromo b-como fragmentos de 307 bp foram amplificados, com um rendimento de 30% a 40% do total de clones de Dna preparado de fresco fígado ou os cabelos da Eurásia lontras (lutra L. (Europa e Asahikawa), mas não a partir do Dna de L lutra (Letónia e China). Assim, a presença de sequência semelhante ao citocromo b não é um artefato durante um longo armazenamento de DNA. A sequência semelhante ao citocromo b também é encontrada no DNA nuclear em um roedor (Smith et al., 1992). As sequências semelhantes ao citocromo b de Lutra são caracterizadas pela presença de uma deleção para causar uma mudança de quadro de leitura e códons de terminação (Fig. 1), pela presença de vários tipos de sequências com pequenas variações (Fig. 1), e pela taxa evolutiva aparentemente mais rápida em comparação com a sequência do citocromo B mitocondrial (ver Fig. 2). Assim, a sequência semelhante ao citocromo b parece não funcionar e está presente como uma pseudo-forma. Não examinamos se a sequência semelhante ao citocromo b de Lutra está presente no DNA mitocondrial ou no DNA nuclear.
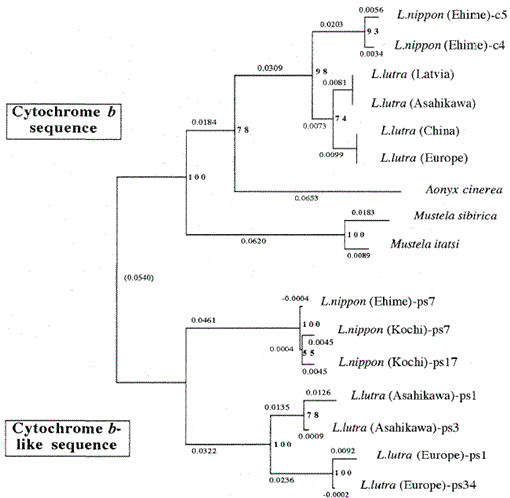
Fig. 2.
uma árvore filogenética Construída a partir do alinhamento da sequência (com base na sequência 1-224 na Fig. 1) das sequências mitocondriais do citocromo b e do citocromo b com o método de junção do vizinho. Os números em cada ponto de ramificação mostram valores de bootstrap (100 repetições), ps representa a sequência semelhante ao citocromo B.
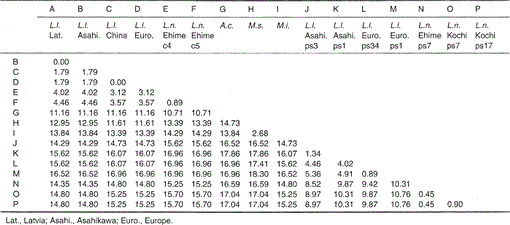
As sequências nucleotídicas de mitocondrial citocromo b e citocromo b-como os genes de Lutra nippon estão alinhados com os de L. lutra (Letónia, a China, a Europa e Asahikawa), Ônix cinerea, Mustela sibirica e Mustela itatsi, na Fig. 1. Da sequência 1 224, Lutra nippon (C4) foi diferente da dos L lutra (Letónia) e L. lutra (China), em 9 e 7 de nucleotídeos, respectivamente, os quais foram localizados na terceira posição de um codão e identificados como de transição as diferenças de Uma↔G ou C↔T. A diferença dentro de Lutra lutra foi de 4 nucleotídeos. Essas substituições de nucleotídeos não causam substituição de aminoácidos. Por outro lado, existem 6 nucleotídeos diferença nas duas doninhas congêneres Mustela sibirica e Mustela itatsi nesta região. Assim, a diferença de sequência entre Lutra nippon e Lutra lutra (7-9 nucleotídeos: 3,6% em média) é maior do que (6 nucleotídeos) entre as duas espécies de Mustela. Isso também é consistente com a ideia de que, nas espécies Mustelidae, mais de 3,5% de diferença na sequência do gene do citocromo b pode corresponder à diferença de espécies distintas (Masuda e Yoshida, 1994). A diferença percentual entre as sequências de nucleotídeos é mostrada na Tabela 1.
Quadro 1.
diferença percentual entre as sequências de nucleotídeos (224 PB) mostradas na Fig. 1
A Figura 2 mostra uma árvore filogenética Construída a partir dos dados de sequência obtidos neste estudo com o método de união de vizinhos no Pacote PHYLIP versão 3.5 c (Felsenstein, 1993). A mesma topologia foi reconstruída com o método Fitch e Margoliash (Felsenstein, 1993). As sequências foram divididas em dois grupos principais, um para os genes do citocromo B mitocondrial e outro para sequências semelhantes ao citocromo B. Como dito acima, as sequências semelhantes ao citocromo b foram amplificadas a partir dos DNAs de Lutra nippon (2 indivíduos, de Kochi e Ehime), l. lutra (Europa) e L. lutra (Asahikawa). O padrão ramificação do citocromo b-como sequências de dois Lutra espécies foi o mesmo mitocondrial citocromo b genes, mas a taxa evolutiva para o citocromo b-como sequências foi pelo menos duas vezes mais rápido, de acordo com as características gerais de pseudo-genes. A árvore também mostra claramente que há uma grande diferença genética entre Lutra nippon e Lutra lutra, e este resultado está de acordo com o recente estudo morfológico que os Japoneses do rio lontra não é uma subespécie da Eurásia lontra Lutra lutra, mas uma espécie distinta, Lutra nippon (Imaizumi e Yoshiyuki, 1989). No entanto, para obter uma evidência conclusiva para a relação filogenética entre as duas espécies, uma sequência de DNA mais longa de Lutra nippon deve ser determinada. Isso seria feito quando a amostra fresca da lontra do Rio Japonês fosse obtida.