resultat och diskussion
vi har förstärkt 307 bp-fragmentet av mitokondriella cytokrom B-gener från de eurasiska uttrarna Lutra lutra (Lettland), L. lutra (Kina), L lutra (Europa) och L. lutra (Asahikawa), den asiatiska småklövade Uttern Aonyx cinerea och vesslorna Mustela sibirica och Mustela itatsi, subklonerade de förstärkta produkterna i puc18 och bestämde deras sekvenser. Sekvenserna av L. lutra (Europa) och L. lutra (Asahikawa) kom helt överens med L lutra (Kina) respektive l lutra (Lettland). Sekvenserna av två individer av Mustela itatsi var identiska, men olika i tre positioner från det som rapporterades nyligen (Masuda och Yoshida, 1994), i sekvenspositionerna 1-224 (se Fig. 1).
Fig. 1.
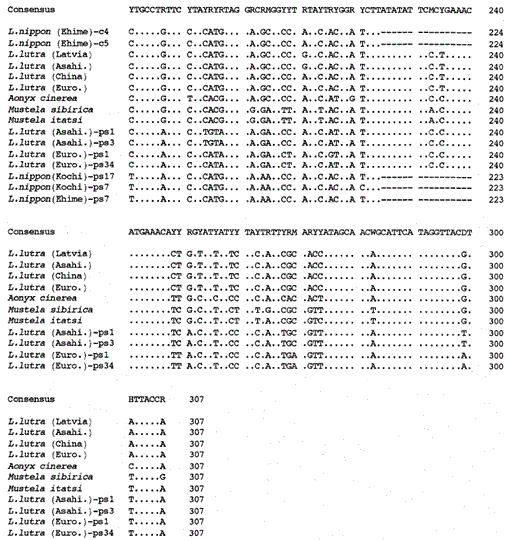
jämförelse av nukleotidsekvenserna av mitokondriell cytokrom b och cytokrom b-liknande sekvenser av Lutra nippon (Ehime och Kochi) med de av L lutra (Lettland, Kina, Europa och Asahikawa), Aonyx cinerea, Mustela sibirica och M. itatsi. Sekvenslängden, 224 bp för L. nippon och 307 bp för de andra. Flera representanter för cytokrom b-liknande sekvenser (ps) visas. Positioner för radering och avslutningskodon i cytokrom b-liknande sekvenser är boxade. Euroområdet., Europa; Asahi., Asahikawa.
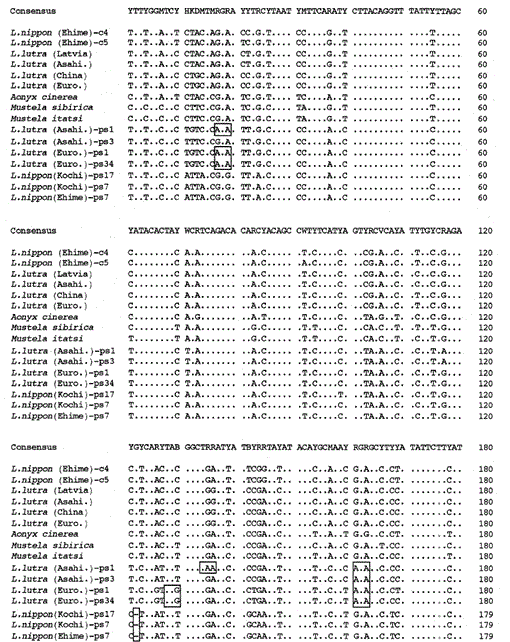
Fig. 1.
fortsättning.
å andra sidan gjordes många försök att förstärka DNA-fragmentet av cytokrom b-genen av Lutra Nippon med olika 10-50 år gamla prover, såsom lever, hjärta och hud bevarad i formalin, det garvade läderet och håren i fyllda exemplar, men alla försök misslyckades.
vi har lyckats förstärka 224 bp-fragmentet av mitokondriell cytokrom b-gen från ett 30-årigt mumialiknande prov av Lutra nippon från Ehime (Fig. 1). Direkt sekvensering av de förstärkta produkterna föreslog närvaron av mer än två typer av sekvens. Därför subklonerades produkterna i pUC18 och 30 kloner sekvenserades. Bland dem identifierades nio kloner tvetydigt för att vara mitokondriell cytokrom b-sekvens av Lutra nippon eftersom sekvensen tydligen liknade mitokondriella sekvenser från andra Lutra-arter, varav åtta kloner hade samma nukleotidsekvens (representerad av sekvensen av L. nippon (Ehime) – c4 i Fig. 1) men den återstående (sekvensen av L. nippon (Ehime)-c5 i Fig. 1) var olika i två lägen(21: C och 92: T och 92: t. De elva klonerna hade cytokrom b-liknande sekvenser som beskrivs nedan. De återstående 10 klonerna hade oväntat den nästan identiska sekvensen med griscytokrom b-sekvensen, vilket tyder på att någon allvarlig DNA-kontaminering inträffade under lagring av det mummiliknande provet.
vi fann att DNA från en mumialiknande Lutra Nippon (Ehime) innehåller en cytokrom b-liknande sekvens. Överraskande motsvarade alla de förstärkta produkterna från en annan Lutra nippon från Kochi (minst 30 år gammal fylld prov) de cytokrom b-liknande sekvenserna, och inget mitokondriellt cytokrom B-fragment förstärktes. Dessutom förstärktes de cytokrom B-liknande fragmenten av 307 bp, med ett utbyte av 30-40% av de totala klonerna, från DNA: erna framställda från den färska levern eller håren från de eurasiska uttrarna L. lutra (Europa och Asahikawa), men inte från DNA: erna från L lutra (Lettland och Kina). Således är närvaron av cytokrom b-liknande sekvens inte en artefakt under en lång lagring av DNA. Den cytokrom b-liknande sekvensen finns också i kärn-DNA i en gnagare (Smith et al., 1992). De cytokrom b-liknande sekvenserna av Lutra kännetecknas av närvaron av en deletion för att orsaka en läsramförskjutning och termineringskodoner (Fig. 1), genom närvaron av flera typer av sekvenser med mindre variationer (Fig. 1), och med den uppenbarligen snabbare evolutionära hastigheten jämfört med mitokondriell cytokrom b-sekvens (se Fig. 2). Således verkar cytokrom b – liknande sekvens inte fungera och är närvarande som en pseudoform. Vi har inte undersökt om cytokrom b-liknande sekvens av Lutra är närvarande i mitokondriellt DNA eller i kärn-DNA.
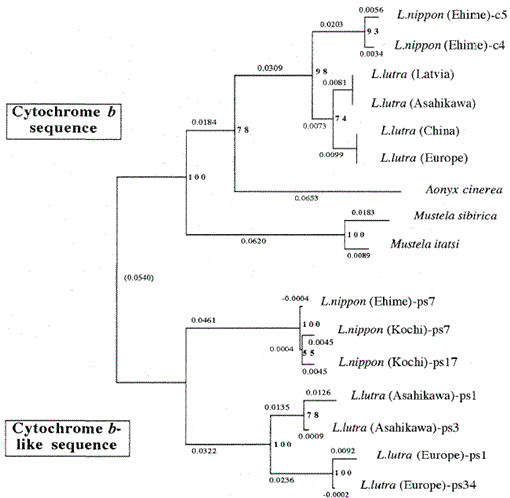
Fig. 2.
ett fylogenetiskt träd konstruerat från sekvensinriktningen (baserat på sekvens 1-224 i Fig. 1) av mitokondriell cytokrom b och cytokrom b-liknande sekvenser med Grannanslutningsmetoden. Siffrorna vid varje förgreningspunkt visar bootstrap-värden (100 replikationer), ps representerar cytokrom b-liknande sekvens.
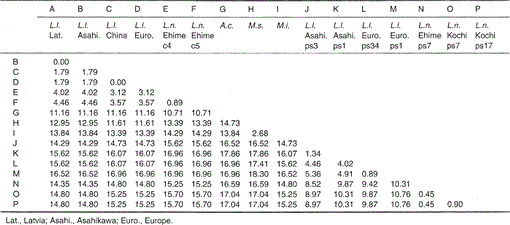
nukleotidsekvenserna av mitokondriell cytokrom b och cytokrom b-liknande gener av Lutra nippon är i linje med L. lutra (Lettland, Kina, Europa och Asahikawa), Aonyx cinerea, Mustela sibirica och Mustela itatsi, i Fig. 1. Av sekvensen 1 till 224 skilde sig Lutra nippon (C4) Från L lutra (Lettland) och L. lutra (Kina) i 9 respektive 7 nukleotider, vilka alla var belägna vid det tredje läget för ett kodon och identifierades som övergångsskillnader a Bisexuell G eller C Bisexuell T. skillnaden inom Lutra lutra var 4 nukleotider. Dessa nukleotidsubstitutioner orsakar ingen aminosyraersättning. Å andra sidan finns det 6 nukleotider skillnad i de två kongeneriska väv Mustela sibirica och Mustela itatsi i denna region. Således är sekvensskillnaden mellan Lutra nippon och Lutra lutra (7-9 nukleotider: 3,6% i genomsnitt) större än den (6 nukleotider) mellan de två Mustela-arterna. Detta överensstämmer också med tanken att i Mustelidae-arten kan mer än 3,5% skillnad i sekvens av cytokrom b-gen motsvara skillnaden mellan olika arter (Masuda och Yoshida, 1994). Den procentuella skillnaden mellan nukleotidsekvenserna visas i Tabell 1.
Tabell 1.
procentuell skillnad mellan nukleotidsekvenserna (224 bp) som visas i Fig. 1
Figur 2 visar ett fylogenetiskt träd konstruerat från sekvensdata som erhållits i denna studie med Grannanslutningsmetoden i PHYLIP-paketversionen 3.5 c (Felsenstein, 1993). Samma topologi rekonstruerades med Fitch och Margoliash-metoden (Felsenstein, 1993). Sekvenserna delades in i två huvudkluster, en är för mitokondriella cytokrom B-gener och den andra för cytokrom b-liknande sekvenser. Som nämnts ovan förstärktes de cytokrom b-liknande sekvenserna från DNAs av Lutra nippon (2 individer, från Kochi och Ehime), L. lutra (Europa) och L. lutra (Asahikawa). Förgreningsmönstret för cytokrom b-liknande sekvenser av de två Lutra-arterna var detsamma som i mitokondriella cytokrom B-gener, men evolutionshastigheten för cytokrom b-liknande sekvenser var minst två gånger snabbare, i överensstämmelse med de allmänna egenskaperna hos pseudogener. Trädet visar också tydligt att det finns en stor genetisk skillnad mellan Lutra nippon och Lutra lutra, och detta resultat överensstämmer med den senaste morfologiska studien att den japanska floduttern inte är en underart av den eurasiska Uttern Lutra lutra, utan en distinkt art, Lutra nippon (Imaizumi och Yoshiyuki, 1989). För att få ett avgörande bevis för det fylogenetiska förhållandet mellan de två arterna måste dock längre DNA-sekvens av Lutra nippon bestämmas. Detta skulle göras när det färska provet av den japanska floduttern erhölls.