- Úvod
- materiály a metody
- příprava vzorků
- gradientní ředění a amplifikace s vícenásobným posunem
- konstrukce a sekvenování knihovny
- Analýza dat
- kontrola kvality sekvence a filtrování
- taxonomické přiřazení a diverzita
- čtení sestavení, predikce genů a anotace
- přístupová čísla nukleotidových sekvencí
- výsledky
- experimentální návrh a sekvenování
- taxonomická anotace
- Metagenomická sestava, predikce genů a funkční anotace
- závěr
- autorské příspěvky
- Prohlášení o střetu zájmů
- potvrzení
- doplňkový materiál
Úvod
Koumiss, také pojmenovaný chige, chigo, arrag nebo airag, v mongolském jazyce, je typ tradičního fermentovaného mléčného výrobku. To bylo populární jídlo v Mongolsku a vnitřní Mongolsko Číny po staletí (Zhang a Zhang, 2011). Lidé v těchto regionech konzumovali koumiss během velkých slavností a obětních obětí (Zhang a Zhang, 2011). Nejstarší záznam výroby koumiss lze vysledovat zpět do dynastie Han (BC202-AD202). Tento produkt dosáhl široké popularity během dynastie Yuan (AD1271-AD1368) (Zhang et al., 2010b). V současné době je koumiss běžným jídlem pro místní obyvatele Mongolska a Vnitřního Mongolska, i když jen v několika z těchto oblastí se vyrábí v průmyslovém měřítku. Koumiss neposkytuje pouze bohaté živiny, včetně vysokého obsahu esenciálních aminokyselin a vitamínů,ale také se předpokládá, že zmírňuje širokou škálu zdravotních stavů a je prospěšný pro pooperační péči (Jagielski, 1877; Thompson, 1879).
tradičně se koumiss běžně vyrábí v dřevěných sudech, nádobách ze zvířecí kůže nebo urnách. K fermentaci dochází přirozeně při okolní teplotě po přidání filtrovaného klisního mléka do nádoby se starým koumissem, který slouží jako startovací kultura (Zhang a Zhang, 2011). Koumiss je dobrým zdrojem nových bakterií biotechnologického potenciálu (Zhang et al., 2010a; Pan a kol., 2011). Proto je velmi zajímavé prozkoumat a zachovat co nejvíce bakterií koumiss spojených s fermentací. Během posledních desetiletí byla provedena řada studií k prozkoumání bakteriální komunity koumiss (Wu et al ., 2009; Hao et al., 2010), studoval hlavně metodami založenými na kultuře, molekulární biologii a pyrosekvencích (Sun et al., 2010). Mezi těmito různými přístupy poskytla metoda založená na pyrosekvencích nejkomplexnější mikrobiotový profil koumiss nezávislý na fenotypových vlastnostech a problémech kultivability jednotlivých mikrobů. Spektrum funkčních genů kódovaných bakteriemi koumiss a jejich fermentační kapacity však zůstávají špatně charakterizovány, zejména pro vzácné mikrobiální populace.
tato studie použila techniku genomiky jedné buňky k analýze bakteriálních metagenomů 10 vzorků koumiss odebraných z Mongolska a Vnitřního Mongolska v Číně. Současná práce použila nejmodernější technologie při zkoumání bakteriální rozmanitosti mléčných výrobků. Naše práce prokázala proveditelnost objevování nízko hojných taxonů použitím metagenomického přístupu s jednou buňkou. Povzbudivé výsledky by podpořily vývoj a uplatňování nových přístupů při řešení problémů v tradiční oblasti výzkumu.
materiály a metody
příprava vzorků
celkem bylo odebráno 10 vzorků koumiss z Mongolska (MG14, MG15, MG16, MG17 a MG18) a Vnitřního Mongolska Číny (NM17, NM18, NM19, NM20 a NM21) pro studii metagenomiky. Vzorky byly odebrány asepticky a byly přepravovány v suchém ledu.
jeden mililitr každého vzorku byl předem ošetřen podle metodiky popsané v Ward et al. (2013) s některými úpravami. Krátce byly vzorky rozmrazeny v ledové lázni po dobu 3-5 minut. Poté, co se vzorky roztavily, byly podrobeny odstředění s nízkou rychlostí, aby se odstranily nečistoty a shluky eukaryotických buněk. Prokaryotické buňky byly poté peletovány z mléčného séra centrifugací při 13 000 × g po dobu 15 minut. Pelety byly znovu suspendovány ve 2 mL fyziologického roztoku pufrovaného fosfátem (PBS) s 1% Tritonem X-100 a inkubovány po dobu 2 h při 37°C, aby se lyzovaly všechny zbývající eukaryotické buňky. Následně byly bakterie peletovány centrifugací při 13 000 × g po dobu 15 minut a pelety byly znovu suspendovány v 500 µL PBS. Nakonec se centrifugační krok opakoval ještě jednou, aby se bakteriální buňky promyly.
gradientní ředění a amplifikace s vícenásobným posunem
pro detekci nízko hojných bakterií byla bakteriální suspenze odvozená z každého koumissova vzorku sériově zředěna pro následnou amplifikační reakci. Počet buněk v každém vzorku byl zhruba odhadnut pod mikroskopem (Nikon, Tokio, Japonsko) pomocí komory pro počítání buněk (Qiujing, Šanghaj, Čína). Krok ředění pokračoval, dokud počet buněk v každé bakteriální suspenzi nedosáhl přibližně 100. Vícenásobná amplifikace zředěných buněk byla provedena pomocí sady REPLI-g Single Cell Kit (Qiagen, Germantown, MD, USA) podle pokynů výrobce.
konstrukce a sekvenování knihovny
amplifikovaná DNA byla náhodně stříhána a byly vybrány fragmenty přibližně 500 bp. Po výstavbě knihovny byly pro kontrolu kvality knihovny použity PerkinElmer LabChip® Gx Touch a StepOnePlusTM Real-Time PCR systém. Nakonec byly na platformě Illumina HiSeq 2500 sekvenovány 125bp spárované koncové čtení podle pokynů výrobce.
Analýza dat
kontrola kvality sekvence a filtrování
surové čtení generované sekvencerem může obsahovat umělé čtení kontaminace adaptéru během konstrukce knihovny. Proto byly provedeny tři kroky k získání vysoce kvalitní datové sady pro čisté čtení: (1) eliminace čtení způsobených kontaminací adaptérem; (2) Odstranění čtení s průměrným skóre pod phred skóre Q30, které bylo považováno za nejnižší mezní hodnotu pro vysoce kvalitní základnu; (3)odstranění čtení s významným přebytkem “ N “ (≥5% čtení). Následná analýza byla založena na čistých datech. Kromě toho byla vypočtena statistická základní kvalita založená na Q30 a obsahu GC.
byly provedeny zarovnání proti hostitelskému genomu, aby se odstranily hostitelské kontaminující sekvence. Jakékoli čtení původem z hostitele byly vyřazeny před dalším porovnáním s referenčními sekvencemi genomu bakterií (nebo virů). Pro získání přesnějších výsledků byl v zarovnání použit model mem Burrows–Wheeler aligner (BWA) (verze 0.97 a) (Li a Durbin, 2009).
taxonomické přiřazení a diverzita
webový software Metaphlan byl použit pro taxonomické přiřazení k úrovni rodu a druhu (Segata et al., 2012). Porovnat rozmanitost druhů uvnitř a mezi vzorky, analyzovali jsme alfa-a beta-rozmanitost balíčkem souvisejícím s R.
čtení sestavení, predikce genů a anotace
abychom získali komplexnější informace, sestavili jsme stříhané fragmenty do genomu (contigs). Vzhledem k přítomnosti více druhů, které jsou běžné v metagenomických vzorcích, jsme však vylepšili metodu sestavení genomu bioinformatiky běžně používanou pro analýzu jednotlivých druhů integrací SPAdes (verze 3.6.2), Interních skriptů a metagenomických databází (Zerbino a Birney, 2008; Nurk et al., 2013).
software MetaGeneMark byl použit pro predikci genů sestavených spojů (Noguchi et al ., 2006). Redundantní geny byly odstraněny pomocí CD-HIT s pokrytím 90 a 95% identity (Li a Godzik, 2006; Fu et al., 2012). Relativní hojnost genů byla stanovena sladěním vysoce kvalitních sekvenačních čtení do katalogu genů stejným postupem. Následná discrepantní analýza byla založena na relativním množství genů. Genová anotace byla provedena sladěním vysoce kvalitních sekvencí s několika veřejnými databázemi (jmenovitě NCBI non-redundantní databáze, NR; shluky Ortologních skupin proteinů, COGs; Kyoto encyklopedie genů a genomů, KEGG) pomocí BLAST (Altschul et al., 1997). Vyhledávání domén bylo provedeno pomocí Interproscan (Mulder a Apweiler, 2008).
přístupová čísla nukleotidových sekvencí
sekvenční data hlášená v této studii byla uložena v databázi SRA (přístupové číslo.SRP083102).
výsledky
k detekci nízko hojných bakterií v koumiss byla použita technika amplifikace jedné buňky k analýze metagenomů vzorků. Tři bakteriální suspenze, každá z přibližně 100 buněk, byly odvozeny z nezávislého koumissova vzorku sériovým ředěním. Bylo analyzováno celkem 30 zředěných suspenzí. Každý zředěný vzorek dostal jiný kód vzorku, tj. identifikační číslo vzorku následované 1, 2 nebo 3, představující tři samostatná ředění. Na základě předpokladu, že některé vzácné druhy budou přítomny v jednom z ředění, bylo provedeno vícenásobné zesílení buněk; a pro každou bakteriální suspenzi koumiss byla vygenerována data 5 Gb.
z 10 koumissových vzorků bylo vygenerováno celkem 1 040 323 864 syrových čtení (celkem 30 bakteriálních suspenzí). Průměrný počet čtení pro každou 100buněčnou suspenzi byl 34 677 462 (doplňková tabulka S1). Po ořezání a filtraci nekvalifikovaných sekvencí jsme získali 1 018 381 702 čistých čtení pro všechny vzorky (doplňková tabulka S1). Hodnoty Shannonova indexu, Simpsonova indexu, Chao1 indexu a počtu pozorovaných druhů (obrázky 1-4) ukázaly, že většina vzorků koumiss měla vysokou bakteriální biologickou rozmanitost. Shannon-Wienerovy křivky diverzity ukázaly, že hloubka sekvence byla dostatečná pro všechny vzorky (Obrázek 1).
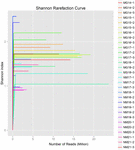
OBRÁZEK 1. Shannon rarefaction křivky, které odhadují mikrobiální rozmanitost vzorků koumiss.
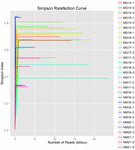
OBRÁZEK 2. Simpsonovy křivky vzácnosti, které odhadují mikrobiální rozmanitost vzorků koumiss.
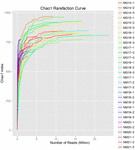
OBRÁZEK 3. Chao1 křivky vzácnosti, které odhadují mikrobiální rozmanitost vzorků koumiss.
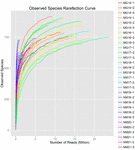
OBRÁZEK 4. Pozorovaný index vzácnosti druhů, který odhaduje mikrobiální rozmanitost vzorků koumiss.
taxonomická anotace
vysoce kvalitní sekvence byly přiřazeny k různým taxonomickým úrovním, aby umožnila hloubkovou analýzu vzorků bakteriálních společenstev. S odkazem na některé publikované studie o biodiverzitě koumiss (Wu et al ., 2009; Hao et al., 2010; Sun a kol., 2010), klasifikovali jsme známé a dříve nehlášené bakterie spojené s koumissem jako běžné a vzácné taxony.
vysoce kvalitní sekvence představovaly 13 různých rodů (obrázek 5). Tři z nich měly průměrnou relativní hojnost přes 1%, včetně Lactobacillus (L.), Lactococcus a Streptococcus. Zejména Lactobacillus a Lactococcus byly dva nejhojnější rody nalezené v koumiss. Podíl Lactobacillus ve vzorcích se pohyboval od 52,72 do 99,96%. Mezi většinou koumissových vzorků dominovali dva členové tohoto rodu, L. helveticus a L. kefirano faciens (obrázek 6). Druh L. buchneri byl přítomen převážně v mongolských vzorcích, zatímco Lactococcus lactis byl detekován ve většině vzorků bez ohledu na oblast odběru vzorků.
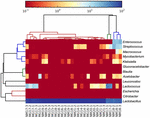
OBRÁZEK 5. Teplotní mapa ukazující relativní množství bakterií zjištěných ve vzorcích koumiss na úrovni rodu.
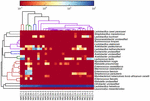
OBRÁZEK 6. Teplotní mapa ukazující relativní množství bakterií zjištěných ve vzorcích koumiss na úrovni druhů.
Metagenomická sestava, predikce genů a funkční anotace
sestavení čtení vedlo k délce sestavy 614 392 623 bp. Hodnoty N50 pro sestavy se pohybovaly od 5 596 do 35 200 bp (doplňková tabulka S2). Počet predikovaných genů ve vzorcích koumiss se pohyboval od 10 347 do 34 547, s průměrnou délkou v rozmezí od 647,82 do 985,33 bp (doplňková tabulka S3). Ačkoli empirické genové funkční analýzy byly nad rámec současné studie, anotovali jsme bakteriální mikrobiomy koumiss pomocí databází COG a KEGG, které předpovídaly genovou funkci do značné míry na základě sekvenční homologie.
celkem 545 zápasů ve výstupu anotace vykazovalo vysokou homologii s geny využití laktózy (COGs Kategorie sacharidů a metabolismu, G) a některé z nich byly umístěny ve stejném contig (Tabulka 1).
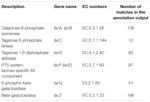
TABULKA 1. Geny související s metabolismem laktózy anotované v bakteriálním metagenomu koumiss.
některé další sekvence mohou kódovat domnělé geny v kategorii COGs transportu a metabolismu aminokyselin (E), včetně sekvencí, které odpovídaly kasein-degradujícím proteinázám, systému Opp pro absorpci oligopeptidů 4-18 reziduí a aminopeptidázám (např. leucylaminopeptidáza, peptidyl-dipeptidáza A, aminopeptidáza N, prolin iminopeptidáza a endopeptidáza). Ačkoli některé sekvence sdílely vysoké podobnosti s aminotransferázami specifickými pro arginin, aspartát, methionin a isoleucin, byla identifikována pouze jedna významná shoda, která odpovídala domnělé doméně třídy I/třídy II (IPR004839) obsahující aminotransferázu specifickou pro tyrosin a fenylalanin. Nakonec řada sekvencí sdílela vysokou homologii s aminokyselinovými lyázami, včetně s-ribosylhomocystein lyázy, argininosukcinát lyázy, aspartát amoniak-lyázy, cystathionin gama-lyázy, histidin amoniak-lyázy a O-acetylhomoserin (thiol)-lyázy.
Koumiss je populární tradiční fermentovaný mléčný výrobek v Mongolsku a Vnitřním Mongolsku v Číně. Ačkoli byla provedena řada studií ke zkoumání bakteriální rozmanitosti v koumiss, bylo získáno jen málo informací o genetické kapacitě mikrobioty koumiss. Zde jsme použili jednobuněčnou amplifikační techniku k analýze bakteriálních metagenomů koumiss, zejména se zaměřením na nízko hojnou bakteriální populaci.
typický metagenomický přístup byl dříve použit k popisu koumissovy mikrobioty. Vzhledem k vysokým nákladům na dosažení hluboké sekvence je však vzácná populace mikrobioty ve vzorcích často nedostatečně pokryta. Fylogenní i funkční metagenomy menšinových bakterií tak zůstávají omezené. Naše jednobuněčná amplifikační metoda zahrnovala sériové ředění vzorků na 100buněčné suspenze. Vzhledem k nízkému počtu buněk přítomných ve zředěných koumissových vzorcích by bylo extrahováno pouze omezené množství DNA. Amplifikační krok zvýšil množství analyzovaných materiálů DNA, a tím usnadnil analýzu metagenomu vzorků obsahujících nepatrné množství DNA. Jednou z nevýhod této metody bylo obtížné zajistit, aby sekvence každého taxonu byly v procesu stejně zesíleny; proto zde získané výsledky mohly odrážet pouze relativní množství sekvencí, ale ne absolutní množství identifikovaných taxonů nebo funkčních genů. Dosud, To by mělo malý vliv na naši analýzu, protože tato studie se liší od ostatních publikovaných prací v zaměření vzácné bakteriální populace. Data generovaná touto prací poskytují doplňující informace nedostatečně zastoupené populaci. Věříme, že současný přístup je vhodný pro budoucí analýzu diverzity bakterií pro různé typy ekologických prostředí.
bakteriální mikrobiota koumiss se skládá hlavně z bakterií mléčného kvašení (laboratoř)a některých bakterií kyseliny octové (Zhang a Zhang, 2011). Jak se dalo očekávat, náš datový soubor obsahoval většinou sekvence představující laboratorní a octové bakterie. Ve všech vzorcích dominovaly sekvence odpovídající Lactobacillus helveticus. Kromě toho, sekvence představující druh, L.kefiranofaciens. L. buchneri. L. kefiranofaciens. Enterococcus (e.) casseliflavus. E. faecalis. E. faecium. Leuconostoc mesenteroides. Byly také nalezeny Lactococcus lactis a Acetobacter pasteurianus. Identifikace sekvencí různých taxonů nemusí stačit k prokázání životaschopnosti bakterií, přesto odráží bakteriální komunitu v určitém okamžiku fermentačního procesu. Miyamoto a kol. (2010) naznačuje, že bakteriální prevalence v konečných fermentovaných produktech souvisí s jejich tolerancí vůči kyselému stresu. Obecně mají laktobacily vyšší toleranci kyselin než laktokoky,což může vysvětlit vysokou relativní hojnost sekvencí laktobacilů přítomných v našem datovém souboru. Na druhé straně častý výskyt L. helveticus v koumiss se shodoval s pozorováním dominance sekvencí L. helveticus.
v našem datovém souboru některé sekvence odpovídaly L. otakiensis, což je vzácný druh, který nebyl nikdy hlášen v koumiss nebo jiných mléčných výklencích. Druh byl nejprve popsán a izolován z nesoleného mořícího roztoku použitého při výrobě sunki, tradičního japonského nálevu (Watanabe et al ., 2009). Byl objeven profilováním polymorfismu délky amplifikovaného fragmentu na základě genu recA. Od té doby nebyl tento druh údajně spojen s jinými výklenky souvisejícími s potravinami. Je tedy pravděpodobné, že tato bakterie patří do autochtonní flóry okurek. Nemůžeme však vyloučit možnost, že nebyla nalezena jednoduše kvůli citlivosti použité detekční metody. Lactobacillus otakiensis může produkovat aminokyseliny s rozvětveným řetězcem, jako je D-leucin, d-allo-isoleucin a d-valin (Doi et al ., 2013). Má potenciál být použit při zlepšování výrobních charakteristik některých fermentovaných potravin (Kato et al ., 2011).
naše studie také identifikovala sekvence představující druh s. macedonicus, který nikdy nebyl hlášen jako bakterie spojená s koumissem. Místo toho se jedná o startovací kulturu přítomnou v řeckých ovčích a kozích sýrech (Georgalaki et al ., 2000). Někteří členové tohoto druhu jsou schopni produkovat exopolysacharidy, bakteriociny (Vincent et al., 2001; Anastasiou et al., 2015) a kyselina gama-aminomáselná (Franciosi et al., 2015). I když je tento druh často izolován z fermentovaných potravin, původní výklenek s. macedonicus byl kontroverzní (Guarcello et al., 2016). Až donedávna, Papadimitriou et al. (2015) identifikoval získaný plasmid, pSMA198, z Lactococcus lactis. Plazmid byl pravděpodobně přenesen prostřednictvím události genetické výměny předků v prostředí mléčných výrobků, což naznačuje mléčný původ s. macedonicus (Papadimitriou et al., 2015). Podobně jako s. thermophilus. S. macedonicus úzce souvisí s komenzály a oportunními patogeny komplexu s. bovis/s. equinus.
identifikace sekvencí představujících torque druhu Ruminococcus byla neočekávaná, protože tato bakterie se obvykle nachází ve střevním prostředí. Je to normální lidský střevní mikrob, který může degradovat mucinové oligosacharidy konstitutivní produkcí sekrečních glykosidáz (Hoskins et al., 1985). Nedávné klinické důkazy ukazují, že fekální hojnost tohoto druhu se mění u dětí s poruchou autistického spektra; jeho role v poruše však zůstává nejasná (Wang et al., 2013).
překvapivě některé sekvence představovaly potenciální patogeny. Například Klebsiella pneumonie je oportunní lidský patogen, který se nachází v přibližně 40% lidských a zvířecích vnitřností. Mycobacterium orygis, dříve nazývaný Oryx bacillus, je členem komplexu Mycobacterium tuberculosis, který může způsobit lidskou tuberkulózu (Dawson et al ., 2012). Tyto dva druhy byly hlášeny v syrovém mléce, ale ne koumiss. Proto by jejich přítomnost mohla připisovat kontaminaci během konvenční výroby koumiss, zejména za podmínek ne-nebo s nízkou aseptickou manipulací.
bakteriální metabolismus hraje důležitou roli při formování vlastností a kvality koumiss. Pro syntézu aromatických a aromatických sloučenin koumiss jsou vyžadovány mikrobiální procesy, jako je lipolýza a proteolýza (Gesudu et al ., 2016). Důsledně bakteriální metagenom obsahoval sekvence, které potenciálně kódují degradaci laktózy a proteolytické systémy. Na rozdíl od relativně jednoduchých katabolických drah laktózy jsou laboratorní proteolytické systémy tvořeny různorodou řadou enzymů (Chen et al., 2014). Ve srovnání s jinými detekovanými koumiss LAB je dominantní druh L. helveticus charakterizován vysokou proteolytickou aktivitou (Zhang et al ., 2015). Většina laboratoří má pouze jednu proteinázu s buněčnou obálkou, která iniciuje hydrolýzu mléčného kaseinu, zatímco L. helveticus obsahuje alespoň dva z těchto enzymů, jmenovitě PrtH a PrtH2 (Zhao et al ., 2011). Vysoké podíly sekvencí odpovídajících silným proteolytickým druhům l. helveticus a genům souvisejícím s proteolýzou se tedy mohou vázat na relativně vysoký obsah peptidů a volných aminokyselin v koumiss.
jednou z obtíží při výrobě koumiss v průmyslovém měřítku je kontrola vnímání chuti, protože koumiss se tradičně vyrábí přirozenou fermentací. Bylo tedy obtížné definovat chuť a profil klíčových aromatických složek, zejména v přítomnosti přírodních kontaminantů. Výroba klíčových aromatických složek je výsledkem fermentační a enzymatické degradace aminokyselin,jako jsou aminokyseliny s rozvětveným řetězcem, methionin a aromatické aminokyseliny (Ardo, 2006). Příklady aromatických složek zahrnují aldehydy, organické kyseliny a estery, které jsou tvořeny transaminázou (AT)-dráhy (Helinck et al., 2004). Tato cesta je iniciována transaminázou, která katalyzuje přeměnu aminokyseliny na odpovídající α-keto kyselinu (Helinck et al ., 2004). Některé sekvence v našem datovém souboru odpovídaly domnělým aminotransferázám s rozvětveným řetězcem a aromatickými aminokyselinami. Zvláště jsme našli domnělou aromatickou aminokyselinu aminotransferázu I v contigu druhu přidruženého k koumissovi, s. macedonicus. Vzhledem k tomu, že naše současná práce pouze anotovala mikrobiom v silico, naše data nestačí k tomu, aby ukázaly, že tyto identifikované genové sekvence byly skutečně funkční pro výrobu výše uvedených koumissových aromatických sloučenin. Přítomnost těchto genů nicméně naznačuje, že jsou některými možnými kandidáty na takové fermentační aktivity; dosud, k objasnění jejich přesných funkčních rolí by bylo zapotřebí další experimentální práce.
kromě toho jsme našli sekvence odpovídající jiným cestám konverze aminokyselin, včetně aminokyselinových lyáz a threonin aldolázy. První enzym katalyzuje přeměnu methioninu na methanethiol (Irmler et al., 2008), což má za následek tvorbu dimethyldisulfidu a dimethyltrisulfidu (Fernandez et al., 2000), zatímco druhý katalyzuje přeměnu threoninu na glycin a acetaldehyd (Ott et al., 2000). Podobně pouhé umístění těchto sekvencí v mikrobiomu koumiss nemůže sloužit jako jednoznačný důkaz v jejich skutečných biologických rolích; bude vyžadováno budoucí experimentální potvrzení.
závěr
tato studie použila modifikovanou metagenomickou metodu k analýze bakteriálního mikrobiomu vzorků koumiss odebraných z Mongolska a Vnitřního Mongolska Číny. Charakterizovali jsme fylogenní i funkční metagenomy vzácných druhů v koumiss; a náš datový soubor odráží vlastnosti, které mají biotechnologický zájem a potenciál. Naše studie poprvé prokázala proveditelnost začlenění jednobuněčných amplifikačních technik do detekce bakteriální mikrobioty koumiss, stejně jako mikrobiální kontaminanty. Zde vyvinuté techniky mohou být použity v budoucích studiích ke sledování změn v mikrobiomu koumiss během fermentačního procesu se zaměřením na menšinovou mikrobiální populaci. Kromě toho mohou být další techniky omics, jako je transkriptomika, metabolomika, spojeny se současnou metagenomickou analýzou, aby se potvrdily funkce a metabolická kapacita mikrobiomu koumiss.
technicky by dalším krokem této práce bylo optimalizovat současnou metodu. Například zvýšením ředění vzorku před amplifikací DNA může být zvýšena šance na odhalení vzácných a nových bakterií. Na druhou stranu, alternativní sekvenční technika, která může generovat dlouhé čtení, jako je Pacific Biosciences single molecule, technologie sekvenování v reálném čase, lze použít ke zlepšení procesu sestavování genomu.
autorské příspěvky
WZ a HZ navrhly studii. WZ, GY, JY, a L-YK napsal rukopis. QH, WH, WL, BM a TS provedly experimenty. WZ a QH analyzovala data. Všichni autoři přezkoumali rukopis.
Prohlášení o střetu zájmů
autoři prohlašují, že výzkum byl proveden bez jakýchkoli obchodních nebo finančních vztahů, které by mohly být vykládány jako potenciální střet zájmů.
potvrzení
tento výzkum byl podpořen Národní přírodovědnou nadací Číny (Grant č. 31571815).
doplňkový materiál
doplňkový materiál k tomuto článku lze nalézt online na adrese: https://www.frontiersin.org/article/10.3389/fmicb.2017.00165/full#supplementary-material
Altschul, s. F., Madden, T. L., Schaffer, a. a., Zhang, J., Zhang, z., Miller, W., a kol. (1997). Gapped BLAST a PSI-BLAST: nová generace programů pro vyhledávání v databázi proteinů. Nukleové Kyseliny Rez.25, 3389-3402. doi: 10.1093/nar / 25.17.3389
PubMed Abstrakt / CrossRef Plný Text / Google Scholar
Anastasiou, R., Driessche, G. v., Boutou, e., Kazou, m., Alexandraki, v., Vorgias, C. E., et al. (2015). Upravené kmeny Streptococcus macedonicus směrem k osmotickému stresově rezistentnímu fenotypu si zachovávají svou schopnost produkovat bakteriocin macedocin za hyperosmotických podmínek. J. Biotechnol. 212, 125–133. doi: 10.1016 / j. jbiotec.2015.08.018
PubMed Abstrakt / CrossRef Plný Text / Google Scholar
Ardo, Y. (2006). Tvorba chuti katabolismem aminokyselin. Biotechnol. ADV. 24, 238-242. doi: 10.1016 / j. biotechadv.2005.11.005
PubMed Abstrakt / CrossRef Plný Text / Google Scholar
Chen, y. f., Zhao, W. J., Wu, R. N., Sun, z. H., Zhang, W. Y., Wang, J. C., et al. (2014). Proteomová analýza Lactobacillus helveticus H9 během růstu odstředěného mléka. J.Dairy Sci. 97, 7413–7425. doi: 10.3168 / jds.2014-8520
PubMed Abstrakt / CrossRef Plný Text / Google Scholar
Dawson, K. L., Bell, a., Kawakami, R. P., Coley, k., Yates, G., and Collins, D. M. (2012). Přenos Mycobacterium orygis (m. tuberculosis complex species) z pacienta s tuberkulózou na dojnici na Novém Zélandu. J. Clin. Mikrobiol. 50, 3136–3138. doi: 10.1128 / JCM.01652-12
PubMed Abstrakt / CrossRef Plný Text / Google Scholar
doi, k., Mori, k., Mutaguchi, y., Tashiro, k., Fujino, y., Ohmori, T., et al. (2013). Návrh genomové sekvence producenta aminokyselin s rozvětveným řetězcem Lactobacillus otakiensis JCM 15040T, izolovaného z tradiční japonské okurky. Genome Announc. 1, e546-e513. doi: 10.1128 / genomeA.00546-13
PubMed Abstrakt / CrossRef Plný Text / Google Scholar
Fernandez, M., van Doesburg, W., Rutten, g. a., Marugg, J. D., Alting, A. C., van Kranenburg, R., et al. (2000). Molekulární a funkční analýzy metC genu Lactococcus lactis, kódující cystathionin beta-lyázu. Appl. Environ. Mikrobiol. 66, 42–48. doi: 10.1128/AEM.66.1.42-48.2000
PubMed Abstrakt / CrossRef Plný Text / Google Scholar
Franciosi, e., Carafa, i., Nardin, T., Schiavon, s., Poznanski, e., Cavazza, A., et al. (2015). Biodiverzita a produkce kyseliny gama-aminomáselné bakteriemi mléčného kvašení izolovanými z tradičních alpských sýrů ze syrového kravského mléka. Biomed Res. Int. 2015, 625740. doi: 10.1155/2015/625740
PubMed Abstrakt / CrossRef Plný Text / Google Scholar
Fu, L., Niu, B., Zhu, z., Wu, s., and Li, w. (2012). CD-HIT: zrychlené pro shlukování sekvenčních dat nové generace. Bioinformatics 28, 3150–3152. doi: 10.1093/bioinformatics/bts565
PubMed Abstract | CrossRef Full Text | Google Scholar
Georgalaki, M. D., Sarantinopoulos, P., Ferreira, E. S., De Vuyst, L., Kalantzopoulos, G., and Tsakalidou, E. (2000). Biochemical properties of Streptococcus macedonicus strains isolated from Greek Kasseri cheese. J. Appl. Microbiol. 88, 817–825. doi: 10.1046/j.1365-2672.2000.01055.x
PubMed Abstract | CrossRef Full Text | Google Scholar
Gesudu, Q. M., Zheng, Y., Xi, X. X., Hou, Q. C., Xie, H. Y., Huang, W. Q., et al. (2016). Zkoumání struktury a dynamiky bakteriální populace v tradičním koumiss z Vnitřního Mongolska pomocí sekvenování jedné molekuly v reálném čase. J.Dairy Sci. 99, 7852–7863. doi: 10.3168 / jds.2016-11167
PubMed Abstrakt / CrossRef Plný Text / Google Scholar
Guarcello, R., Carpino, s., Gaglio, R., Pino, a., Rapisarda, T., Caggia, C., et al. (2016). Rozsáhlá tovární aplikace vybraných autochtonních bakterií mléčného kvašení pro výrobu sýrů Chop Pecorino Siciliano. Potravinový Mikrobiol. 59, 66–75. doi: 10.1016 / j. fm.2016.05.011
PubMed Abstrakt / CrossRef Plný Text / Google Scholar
Hao, y., Zhao, L., Zhang, h., Zhai, z., Huang, y., Liu, X. a kol. (2010). Identifikace bakteriální biodiverzity v koumiss pomocí denaturační gradientní gelové elektroforézy a druhově specifické polymerázové řetězové reakce. J.Dairy Sci. 93, 1926–1933. doi: 10.3168 / jds.2009-2822
PubMed Abstrakt / CrossRef Plný Text / Google Scholar
Helinck, s., Le Bars, D., Moreau, D., and Yvon, m. (2004). Schopnost termofilních bakterií mléčného kvašení produkovat aromatické sloučeniny z aminokyselin. Appl. Environ. Mikrobiol. 70, 3855–3861. doi: 10.1128/AEM.70.7.3855-3861.2004
PubMed Abstrakt / CrossRef Plný Text / Google Scholar
Hoskins, L. C., Agustines, m., McKee, W.B., Boulding, E.T., Kriaris, M., and Niedermeyer, G. (1985). Degradace mucinu v ekosystémech lidského tlustého střeva. Izolace a vlastnosti fekálních kmenů, které degradují antigeny krevní skupiny ABH a oligosacharidy z mucinových glykoproteinů. J. Clin. Investovat. 75, 944–953. doi: 10.1172 / JCI111795
PubMed Abstrakt / CrossRef Plný Text / Google Scholar
Irmler, s., Raboud, s., Beisert, B., Rauhut, D., and Berthoud, h. (2008). Klonování a charakterizace dvou genů Lactobacillus casei kódujících cystathionin lyázu. Appl. Environ. Mikrobiol. 74, 99–106. doi: 10.1128/AEM.00745-07
PubMed Abstrakt / CrossRef Plný Text / Google Scholar
Jagielski, V. (1877). Hodnota koumiss při léčbě nevolnosti, zvracení a neschopnosti udržet jiné potraviny na žaludku. Br. Med. J. 2, 919-921. doi: 10.1136 / bmj.2.887.919
PubMed Abstrakt / CrossRef Plný Text / Google Scholar
Kato, s., Ishihara, T., Hemmi, h., Kobayashi, H., and Yoshimura, T. (2011). Změny koncentrací D-aminokyselin a mikrobiálních komunitních struktur během fermentace červených a bílých vín. J. Biosci. Bioeng. 111, 104–108. doi: 10.1016 / j. jbiosc.2010.08.019
PubMed Abstrakt / CrossRef Plný Text / Google Scholar
Li, H., and Durbin, R. (2009). Rychlé a přesné zarovnání krátkého čtení s transformací Burrows-Wheeler. Bioinformatika 25, 1754-1760. doi: 10.1093 / bioinformatika / btp324
PubMed Abstrakt / CrossRef Plný Text / Google Scholar
Li, W., and Godzik, a. (2006). Cd-hit: rychlý program pro shlukování a porovnávání velkých sad proteinových nebo nukleotidových sekvencí. Bioinformatika 22, 1658-1659. doi: 10.1093 / bioinformatika / btl158
PubMed Abstrakt / CrossRef Plný Text / Google Scholar
Miyamoto, m., Seto, T., Nakajima, h., Burenjargal, s., Gombojav, a., Demberei, S., et al. (2010). Denaturační gradientní gelová elektroforéza analýza bakterií mléčného kvašení a kvasinek v tradičním mongolském fermentovaném mléce. Jídlo Sci. Technol. Rez.16, 319-326. doi: 10.3136 / fstr.16.319
CrossRef Plný Text / Google Scholar
Mulder, NJ, and Apweiler, R. (2008). Databáze InterPro a nástroje pro analýzu proteinových domén. Curre. Protokol. Bioinformatika Kapitola 2, Jednotka2. 7. doi: 10.1002 / 0471250953. bi0207s21
PubMed Abstrakt / CrossRef Plný Text / Google Scholar
noguchi, H., Park, J., and Takagi, T. (2006). Metagen: hledání prokaryotických genů ze sekvencí brokovnice environmentálního genomu. Nukleové Kyseliny Rez.34, 5623-5630. doi: 10.1093/nar / gkl723
PubMed Abstrakt / CrossRef Plný Text / Google Scholar
Nurk, s., Bankevich, a., Antipov, D., Gurevič, A. a., Korobeynikov, a., Lapidus, A., et al. (2013). Sestavení jednobuněčných genomů a mini-metagenomů z chimérických produktů MDA. J. Biol. 20, 714–737. doi: 10.1089 / cmb.2013.0084
PubMed Abstrakt / CrossRef Plný Text / Google Scholar
Ott, a., Germond, J. E., and Chaintreau, a. (2000). Původ acetaldehydu během fermentace mléka za použití (13)prekurzorů značených C. J. Agric. Jídlo Chem. 48, 1512–1517. doi: 10.1021 / jf9904867
PubMed Abstrakt / CrossRef Plný Text / Google Scholar
Pan, D. D., Zeng, X. Q., and Yan, Y. T. (2011). Charakterizace Lactobacillus fermentum SM-7 izolované z koumiss, potenciální probiotické bakterie s účinky snižujícími hladinu cholesterolu. J.Sci. Potravina. Agricu. 91, 512–518. doi: 10.1002 / jsfa.4214
PubMed Abstrakt / CrossRef Plný Text / Google Scholar
Papadimitriou, K., Anastasiou, R., Maistrou, e., Plakas, T., Papandreou, N. C., Hamodrakas, S. J., et al. (2015). Akvizice horizontálním genovým přenosem plazmidu pSMA198 Streptococcus macedonicus ACA-DC 198 ukazuje na mléčný původ druhu. PLoS ONE 10: e0116337. doi: 10.1371 / deník.pone.0116337
PubMed Abstrakt / CrossRef Plný Text / Google Scholar
Segata, N., Waldron, L., Ballarini, a., Narasimhan, v., Jousson, O., and Huttenhower, C. (2012). Profilování metagenomické mikrobiální komunity pomocí jedinečných markerových genů specifických pro clade. Adresa. Metody 9, 811-814. doi: 10.1038 / nmeth.2066
PubMed Abstrakt / CrossRef Plný Text / Google Scholar
Sun, z., Liu, W., Zhang, J., Yu, J., Zhang, W., Cai, C., et al. (2010). Identifikace a charakterizace dominantních laktobacilů izolovaných z koumiss v Číně. J. Gen.Appl. Mikrobiol. 56, 257–265. doi: 10.2323 / jgam.56.257
PubMed Abstrakt / CrossRef Plný Text / Google Scholar
Thompson, J. (1879). Hodnota koumiss při plýtvání nemocemi. Br. Med. J. 1, 270. doi: 10.1136 / bmj.1.1466.270-c
PubMed Abstrakt / CrossRef Plný Text / Google Scholar
Vincent, S. J., Faber, E. J., Neeser, J. R., Stingele, F., and Kamerling, J. P. (2001). Struktura a vlastnosti exopolysacharidu produkovaného Streptococcus macedonicus Sc136. Glykobiologie 11, 131-139. doi: 10.1093 / glycob / 11.2.131
PubMed Abstrakt / CrossRef Plný Text / Google Scholar
Wang, L., Christophersen, C. T., Sorich, M. J., Gerber, J. P., Angley, M. T., and Conlon, m. a. (2013). Zvýšená hojnost Sutterella spp. a ruminokokové momenty ve stolici dětí s poruchou autistického spektra. Molo. Autismus 4, 42. doi: 10.1186/2040-2392-4-42
PubMed Abstrakt / CrossRef Plný Text / Google Scholar
Ward, T. L., Hosid, s., Ioshikhes, I., and Altosaar, i. (2013). Metagenom lidského mléka: analýza funkční kapacity. BMC Mikrobiol. 13:116. doi: 10.1186/1471-2180-13-116
PubMed Abstrakt / CrossRef Plný Text / Google Scholar
Watanabe, k., Fujimoto, J., Tomii, y., Sasamoto, m., Makino, h., Kudo, y., et al. (2009). Lactobacillus kisonensis sp. Novum., Lactobacillus otakiensis sp. Novum., Lactobacillus rapi sp. Novum. a Lactobacillus sunkii sp. Novum., heterofermentativní druhy izolované ze sunki, tradiční japonské okurky. Int. J. Syst. Evol. Mikrobiol. 59 (bod 4), 754-760. doi: 10.1099/ijs.0.004689-0
PubMed Abstrakt / CrossRef Plný Text / Google Scholar
Wu, R., Wang, L., Wang, J., Li, h., Menghe, B., Wu, J., et al. (2009). Izolace a předběžný probiotický výběr laktobacilů z koumiss ve Vnitřním Mongolsku. J. Základní Mikrobiol. 49, 318–326. doi: 10.1002 / jobm.200800047
PubMed Abstrakt / CrossRef Plný Text / Google Scholar
Zerbino, D. R., and Birney, e. (2008). Velvet: algoritmy pro sestavení krátkého čtení de novo pomocí grafů de Bruijn. Genome Res.18, 821-829. doi: 10.1101 / gr.074492.107
PubMed Abstrakt / CrossRef Plný Text / Google Scholar
Zhang, W., Sun, z., Sun, T., and Zhang, h. (2010a). PCR screening a sekvenční analýza klastrů iol v kmenech Lactobacillus casei izolovaných z koumiss. Folia Microbiol. (Praha) 55, 603-606. doi: 10.1007/s12223-010-0097-3
PubMed Abstrakt / CrossRef Plný Text / Google Scholar
Zhang, W., Yu, D., Sun, z., Wu, R., Chen, X., Chen, W., et al. (2010b). Kompletní genomová sekvence Lactobacillus casei Zhang, nového probiotického kmene izolovaného z tradičního domácího koumiss ve Vnitřním Mongolsku v Číně. J. Bacteriol. 192, 5268–5269. doi: 10.1128 / JB.00802-10
PubMed Abstrakt / CrossRef Plný Text / Google Scholar
Zhang, W., and Zhang, h. (2011). „Fermentace a koumiss,“ v příručce technologie fermentace potravin a nápojů, 2. Edn, ed. Y. h. Hui (Boca Raton, FL: CRC Press), 165-172.
Google Scholar
Zhang, W. Y., Chen, y. f., Zhao, W. J., Kwok, L. Y., and Zhang, H. P. (2015). Genová exprese proteolytického systému Lactobacillus helveticus H9 během fermentace mléka. Anna. Mikrobiol. 65, 1171–1175. doi: 10.3168 / jds.2014-8520
PubMed Abstrakt / CrossRef Plný Text / Google Scholar
Zhao, W., Chen, y., Sun, z., Wang, J., Zhou, z., Sun, T., et al. (2011). Kompletní genomová sekvence Lactobacillus helveticus H10. J. Bacteriol. 193, 2666–2667. doi: 10.1128 / JB.00166-11
PubMed Abstrakt / CrossRef Plný Text / Google Scholar