- introduktion
- material och metoder
- beredning av prover
- Gradientutspädning och multipel Förskjutningsförstärkning
- Bibliotekskonstruktion och sekvensering
- dataanalys
- Sekvenskvalitetskontroll och filtrering
- taxonomisk tilldelning och mångfald
- Läs montering, Genförutsägelse och anteckning
- Nukleotidsekvensanslutningsnummer
- resultat
- experimentell Design och sekvensering
- taxonomisk anteckning
- Metagenomic Assembly, Gen Prediction och funktionell Annotation
- diskussion
- slutsats
- Författarbidrag
- intressekonflikt uttalande
- bekräftelse
- tilläggsmaterial
introduktion
Koumiss, även benämnd chige, chigo, arrag eller airag, på mongoliska språket, är en typ av traditionell fermenterad mejeriprodukt. Det har varit en populär mat i Mongoliet och Inre Mongoliet i Kina i århundraden (Zhang och Zhang, 2011). Människor i dessa regioner brukade konsumera koumiss under stora festligheter och offeroffer (Zhang och Zhang, 2011). Den tidigaste registreringen av koumiss-produktionen kan spåras tillbaka till Han-dynastin (BC202-AD202). Denna produkt hade uppnått utbredd popularitet under Yuan-dynastin (AD1271-AD1368) (Zhang et al., 2010b). Numera är koumiss en vanlig mat för lokalbefolkningen i Mongoliet och Inre Mongoliet, men endast i få av dessa områden tillverkas den i industriell skala. Koumiss ger inte bara rika näringsämnen, inklusive högt innehåll av essentiella aminosyror och vitaminer, men tros också lindra ett brett spektrum av medicinska tillstånd och är fördelaktigt för postoperativ vård (Jagielski, 1877; Thompson, 1879).
traditionellt produceras koumiss vanligtvis i träfat, behållare gjorda av djurhud eller urnor. Fermentering sker naturligt vid omgivningstemperatur efter tillsats av filtrerad sto mjölk i behållaren med gammal koumiss, som fungerar som startkulturen (Zhang och Zhang, 2011). Koumiss är en bra källa till nya bakterier av bioteknikpotential (Zhang et al., 2010a; Pan et al., 2011). Därför är det av intensivt intresse att utforska och bevara så många fermentationsassocierade koumiss-bakterier som möjligt. Under de senaste decennierna utfördes ett antal studier för att undersöka koumiss bakteriesamhälle (Wu et al., 2009; Hao et al., 2010), främst studerat av kultur -, molekylärbiologi – och pyrosequencing-baserade metoder (Sun et al., 2010). Bland dessa olika tillvägagångssätt har den pyrosequencing-baserade metoden tillhandahållit den mest omfattande mikrobiotaprofilen för koumiss oberoende av fenotypiska egenskaper och problem med odling av de enskilda mikroberna. Spektrumet av funktionella gener kodade av koumiss-bakterierna och deras fermentativa kapacitet förblir emellertid dåligt karakteriserade, särskilt för de sällsynta mikrobiella populationerna.
den föreliggande studien använde enkelcellsgenomiktekniken för att analysera bakteriella metagenomer av 10 koumiss-prover som samlats in från Mongoliet och Inre Mongoliet i Kina. Det nuvarande arbetet har tillämpat toppmodern teknik för att undersöka bakteriediversiteten i mejeriprodukter. Vårt arbete har visat möjligheten att upptäcka låga rikliga taxa genom att tillämpa encellsmetagenomics-metoden. De uppmuntrande resultaten skulle främja utvecklingen och tillämpningen av nya metoder för att hantera problem inom ett traditionellt forskningsområde.
material och metoder
beredning av prover
totalt 10 koumiss-prover samlades in från Mongoliet (MG14, MG15, MG16, MG17 och MG18) och Inre Mongoliet i Kina (NM17, NM18, NM19, NM20 och NM21) för metagenomicsstudien. Prover samlades aseptiskt och transporterades i torris.
en milliliter av varje prov förbehandlades enligt den metod som beskrivs i Ward et al. (2013) med några ändringar. I korthet tinades proverna i ett isbad i 3-5 minuter. Efter att proverna smälte utsattes de för centrifugering med låg hastighet för att avlägsna föroreningar och eukaryota cellklumpar. Prokaryota celler pelleterades sedan från mjölkserum genom centrifugering vid 13 000 kg i 15 minuter. Pelletsen suspenderades på nytt i 2 mL fosfatbuffrad saltlösning (PBS) med 1% Triton X-100 och inkuberades i 2 timmar vid 37 kcal C för att lysera eventuella återstående eukaryota celler. Därefter pelleterades bakterier genom centrifugering vid 13,000 xnumx kg för 15 min och pelletsna suspenderades på nytt i 500 xnumx kg. Slutligen upprepades centrifugeringssteget en gång till för att tvätta bakteriecellerna.
Gradientutspädning och multipel Förskjutningsförstärkning
för att detektera de låga rikliga bakterierna späddes bakteriesuspensionen härledd från varje koumiss-prov seriellt för efterföljande amplifieringsreaktion. Cellnumret i varje prov uppskattades grovt under ett mikroskop (Nikon, Tokyo, Japan) med hjälp av en cellräkningskammare (Qiujing, Shanghai, Kina). Utspädningssteget fortsatte tills cellnumret i varje bakteriell suspension nådde cirka 100. Multipelförskjutningsförstärkning av de utspädda cellerna utfördes med användning av REPLI – g Single Cell Kit (Qiagen, Germantown, MD, USA) enligt tillverkarens instruktioner.
Bibliotekskonstruktion och sekvensering
Amplifierat DNA klipptes slumpmässigt och fragmenten på cirka 500 bp valdes. Efter bibliotekskonstruktionen användes PerkinElmer LabChip bisexual GX Touch och StepOnePlusTM realtids-PCR-System för bibliotekets kvalitetskontroll. Slutligen sekvenserades 125-bp-Parade slutläsningar på Illumina HiSeq 2500-plattformen enligt tillverkarens instruktioner.
dataanalys
Sekvenskvalitetskontroll och filtrering
Raw-läsningar som genereras av sequencer kan innehålla artificiella läsningar av adapterförorening under bibliotekets konstruktion. Därför utfördes tre steg för att få en högkvalitativ renläst dataset: (1) eliminering av läsningar orsakade av adapterförorening; (2) avlägsnande av läsningar med en genomsnittlig poäng under en phred-poäng på Q30, som ansågs vara den lägsta avstängningen för en högkvalitativ BAS; (3) avlägsnande av läsningar med ett betydande överskott av ”N” (5% av läsningen i 5% av läsningen). Nedströmsanalysen baserades på rena data. Dessutom beräknades den statistiska baskvaliteten, baserad på Q30 och GC-innehållet.
inriktningar mot värdgenomet utfördes för att avlägsna de värdstörda föroreningssekvenserna. Alla värd-ursprung läser kasserades innan ytterligare jämförelse med bakterier (eller virus) genomreferenssekvenser. För att få mer exakta resultat användes Burrows–Wheeler aligner (BWA) (Version 0.97 a) mem-modellen i inriktningarna (Li och Durbin, 2009).
taxonomisk tilldelning och mångfald
webbprogramvaran Metaphlan användes för taxonomisk tilldelning till släkt-och artnivåer (Segata et al., 2012). För att jämföra mångfalden av arter inom och mellan prover analyserade vi alfa – och beta-mångfalden av det R-relaterade paketet.
Läs montering, Genförutsägelse och anteckning
för att få mer omfattande information monterade vi de klippta fragmenten i genom (contigs). På grund av närvaron av flera arter, vilket är vanligt i metagenomiska prover, förbättrade vi emellertid bioinformatikgenommonteringsmetoden som normalt används för analys av enskilda arter genom att integrera spader (Version 3.6.2), interna skript och metagenomiska databaser (Zerbino och Birney, 2008; Nurk et al., 2013).
MetaGeneMark-programvaran användes för genförutsägelse av de sammansatta contigs (Noguchi et al., 2006). Överflödiga gener avlägsnades med hjälp av CD-HIT med täckningen av 90 och 95% identitet (Li och Godzik, 2006; Fu et al., 2012). Relativa överflöd av generna bestämdes genom att anpassa högkvalitativa sekvenseringsläsningar till genkatalogen med samma procedur. Den nedströms avvikande analysen baserades på genens relativa överflöd. Genanteckning utfördes genom att anpassa högkvalitativa sekvenser mot flera offentliga databaser (nämligen NCBI non-redundant database, NR; kluster av Ortologa grupper av proteiner, Kuggar; Kyoto Encyclopedia of gener och genomer, KEGG) med användning BLAST (Altschul et al., 1997). En domänsökning utfördes med hjälp av Interproscan (Mulder and Apweiler, 2008).
Nukleotidsekvensanslutningsnummer
sekvensdata som rapporterats i denna studie har deponerats i sra-databasen (anslutning nr.SRP083102).
resultat
experimentell Design och sekvensering
för att detektera de låga rikliga bakterierna i koumiss användes singelcellförstärkningstekniken för att analysera metagenomerna i proverna. Tre bakteriella suspensioner, var och en av cirka 100 celler, härleddes från ett oberoende koumiss-prov genom seriell utspädning. Totalt analyserades 30 utspädda suspensioner. Varje utspätt prov gavs en annan provkod, dvs provets identitetsnummer följt av 1, 2 eller 3, vilket representerar de tre separata utspädningarna. Baserat på en förutsättning att vissa sällsynta arter kommer att finnas i en av utspädningarna utfördes multipel förskjutningsförstärkning av cellerna; och cirka 5 Gb data genererades för varje koumiss bakteriell suspension.
totalt 1 040 323 864 råa läsningar genererades från de 10 koumiss-proverna (totalt 30 bakteriesuspensioner). Det genomsnittliga antalet läsningar för varje 100-cellsuspension var 34 677 462 (Tilläggstabell S1). Efter trimning och filtrering av de okvalificerade sekvenserna fick vi 1,018,381,702 rena läsningar för alla prover (kompletterande tabell S1). Värdena för Shannon-index, Simpson-index, Chao1-index och antalet observerade arter (figurerna 1-4) visade att de flesta koumiss-prover hade en hög bakteriell biologisk mångfald. Shannon-Wiener-mångfaldskurvorna visade att sekvensdjupet var tillräckligt för alla prover (Figur 1).
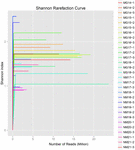
FIGUR 1. Shannon sällsynthet kurvor som uppskattar den mikrobiella mångfalden av koumiss prover.
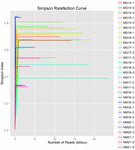
FIGUR 2. Simpson rarefaction kurvor som uppskattar den mikrobiella mångfalden av koumiss prover.
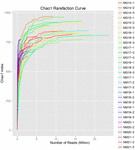
FIGUR 3. Chao1 sällsynthetskurvor som uppskattar den mikrobiella mångfalden av koumiss-prover.
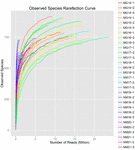
FIGUR 4. Observerade arter sällsynthet index som uppskattar den mikrobiella mångfalden av koumiss prover.
taxonomisk anteckning
de högkvalitativa sekvenserna tilldelades olika taxonomiska nivåer för att möjliggöra en djupgående analys av provbakteriesamhällena. Med hänvisning till några publicerade studier om koumiss biologisk mångfald (Wu et al., 2009; Hao et al., 2010; Sun et al., 2010) klassificerade vi de kända och tidigare inte rapporterade koumiss-associerade bakterierna som vanliga respektive sällsynta taxa.
de högkvalitativa sekvenserna representerade 13 olika släkten (Figur 5). Tre av dem hade en genomsnittlig relativ överflöd på över 1%, inklusive Lactobacillus (L.), Lactococcus och Streptococcus. Särskilt Lactobacillus och Lactococcus var de två vanligaste släktena som hittades i koumiss. Andelen laktobacillus i proverna varierade från 52,72 till 99,96%. Två medlemmar av detta släkte, L. helveticus och L. kefiranofaciens, dominerades bland de flesta koumiss prover (Figur 6). Arten L. buchneri var mest närvarande i de mongoliska proverna, medan Lactococcus lactis detekterades i de flesta prover oavsett provtagningsregion.
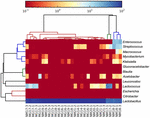
FIGUR 5. Värmekarta som visar det relativa överflödet av bakterier som upptäckts i koumiss-proverna på genusnivå.
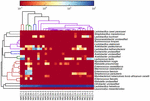
FIGUR 6. Värmekarta som visar det relativa överflödet av bakterier som upptäckts i koumiss-proverna på artnivå.
Metagenomic Assembly, Gen Prediction och funktionell Annotation
montering av läsningar resulterade i en monteringslängd av 614,392,623 bp. N50-värdena för enheterna varierade från 5 596 till 35 200 bp (Tilläggstabell S2). Antalet förutsagda gener i koumiss-proverna varierade från 10 347 till 34 547, med en genomsnittlig längd från 647,82 till 985,33 bp (kompletterande tabell S3). Även om empiriska genfunktionella analyser var utanför ramen för den aktuella studien, kommenterade Vi koumiss bakteriella mikrobiomer med hjälp av COG-och KEGG-databaserna, som förutspådde genfunktion till stor del baserad på sekvenshomologi.
totalt 545 matchningar i annoteringsutgången visade hög homologi mot laktosutnyttjandegenerna (Kuggar kategori av kolhydrat och metabolism, G), och några av dem var belägna inom samma contig (Tabell 1).
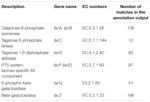
TABELL 1. Laktosmetabolismrelaterade gener kommenterade i koumiss bakteriella metagenom.
vissa andra sekvenser kan koda för förmodade gener inom COGs-kategorin aminosyratransport och metabolism (E), inklusive sekvenser som motsvarade kaseinnedbrytande proteinaser, opp-systemet för att ta upp oligopeptider med 4-18 rester och aminopeptidaser (t.ex. leucylaminopeptidas, peptidyldipeptidas A, aminopeptidas N, prolin iminopeptidas och endopeptidas). Även om vissa sekvenser delade höga likheter med aminotransferaser specifika för arginin, aspartat, metionin och isoleucin identifierades endast en signifikant matchning, vilket motsvarade en förmodad S. macedonicus-ursprung klass i/klass II-domän (IPR004839) innehållande aminotransferas specifikt för tyrosin och fenylalanin. Slutligen delade ett antal sekvenser hög homologi med aminosyralyaserna, inklusive S-ribosylhomocysteinlyas, argininosuccinatlyas, aspartat ammoniak-lyas, cystathionin gamma-lyas, histidin ammoniak-lyas och O-acetylhomoserin (tiol)-lyas.
diskussion
Koumiss är en populär traditionell fermenterad mejeriprodukt i Mongoliet och Inre Mongoliet i Kina. Även om ett antal studier har utförts för att undersöka bakteriemångfalden i koumiss, har liten information erhållits angående koumiss mikrobiotas genetiska kapacitet. Här tillämpade vi singelcellförstärkningstekniken för att analysera koumiss bakteriella metagenomer, särskilt med fokus på den låga rikliga bakteriepopulationen.
den typiska metagenomikmetoden har tidigare tillämpats för att beskriva koumiss mikrobiota. På grund av den höga kostnaden för att uppnå en djup sekvens täcks emellertid den sällsynta mikrobiotapopulationen i proverna ofta otillräckligt. Således förblir både de fylogena och funktionella metagenomerna hos minoritetsbakterierna begränsade. Vår enkelcellsförstärkningsmetod involverade seriell utspädning av prover till 100-cellsuspensioner. På grund av det låga antalet celler som finns i de utspädda koumiss-proverna skulle endast en begränsad mängd DNA extraheras. Amplifieringssteget ökade mängden DNA-material som ska analyseras och underlättade därmed metagenomanalysen av prover som innehåller små DNA-mängder. En nackdel med denna metod var svårigheten att säkerställa att sekvenser av varje taxon skulle förstärkas lika i processen; därför kunde resultaten som erhållits här endast återspegla de relativa överflödena av sekvenser men inte absoluta mängder identifierade taxa eller funktionella gener. Ändå skulle detta ha liten effekt på vår analys eftersom den aktuella studien skiljer sig från andra publicerade verk för att fokusera den sällsynta bakteriepopulationen. Data som genereras av detta arbete ger Kompletterande information till den underrepresenterade befolkningen. Vi tror att det nuvarande tillvägagångssättet är lämpligt för framtida analys av bakteriemångfald för olika typer av ekologiska miljöer.
koumiss bakteriemikrobiota bestod huvudsakligen av mjölksyrabakterier (LAB) och vissa ättiksyrabakterier (Zhang och Zhang, 2011). Som förväntat innehöll vår dataset mestadels sekvenser som representerar LAB-och ättiksyrabakterierna. Sekvenser motsvarande Lactobacillus helveticus dominerade över alla prover. Dessutom sekvenser som representerar arten, L. kefiranofaciens. L. buchneri. L. kefiranofaciens. Enterococcus (E.) casseliflavus. E. faecalis. E. faecium. Leuconostoc mesenteroides. Lactococcus lactis och Acetobacter pasteurianus hittades också. Identifieringen av sekvenser av olika taxa kanske inte räcker för att visa bakteriens livskraft, det återspeglar ändå bakteriegemenskapen vid någon tidpunkt i jäsningsprocessen. Miyamoto et al. (2010) föreslår att bakterieprevalensen i de slutliga fermenterade produkterna är relaterad till deras syrastress tolerans. I allmänhet har laktobaciller en högre syratolerans än laktokocker, vilket kan förklara det höga relativa överflödet av laktobaciller som finns i vår dataset. Å andra sidan, den frekventa förekomsten av L. helveticus i koumiss sammanföll med observationen av dominansen av L. helveticus sekvenser.
i vårt dataset motsvarade vissa sekvenser L. otakiensis, som är en sällsynt art som aldrig har rapporterats i koumiss eller andra mejerinischer. Arten beskrevs först och isolerades från den icke-saltade betningslösningen som användes för att producera sunki, en traditionell japansk pickle (Watanabe et al., 2009). Det upptäcktes genom amplifierad fragmentlängd polymorfism profilering baserat på recA-genen. Sedan dess har denna art inte rapporterats vara associerad med andra livsmedelsrelaterade nischer. Således är det troligt att denna bakterie tillhör den autoktona floran av pickles. Vi kan emellertid inte utesluta möjligheten att det inte hittades helt enkelt på grund av känsligheten hos detekteringsmetoden som används. Lactobacillus otakiensis kan producera D-grenade aminosyror, såsom d-leucin, d-allo-isoleucin och d-valin (Doi et al., 2013). Det har potential att användas för att förbättra produktionsegenskaperna hos vissa fermenterade livsmedel (Kato et al., 2011).
vår studie identifierade också sekvenser som representerar arten S. macedonicus, som aldrig har rapporterats som en koumiss-associerad bakterie. Istället är det en startkultur som finns i grekiska får-och getostar (Georgalaki et al., 2000). Vissa medlemmar av denna art kan producera exopolysackarider, bakteriociner (Vincent et al., 2001; Anastasiou et al., 2015) och gamma-aminosmörsyra (Franciosi et al., 2015). Även om denna art ofta isoleras från fermenterade livsmedel har den ursprungliga nischen av S. macedonicus varit kontroversiell (Guarcello et al., 2016). Inte förrän nyligen, Papadimitriou et al. (2015) identifierade en förvärvad plasmid, pSMA198, från Lactococcus lactis. Plasmid överfördes sannolikt via en förfäders genetisk utbyteshändelse inom en mejeriproduktmiljö,vilket antydde S. macedonicus (Papadimitriou et al., 2015). Liknar S. thermophilus. S. macedonicus är nära besläktad med kommensaler och opportunistiska patogener i S. bovis/S. equinus-komplexet.
identifieringen av sekvenser som representerar arten Ruminococcus vridmoment var oväntad, eftersom denna bakterie vanligtvis finns i tarmmiljön. Det är en normal mänsklig tarmmikrob som kan försämra mucinoligosackarider genom konstitutiv produktion av sekretoriska glykosidaser (Hoskins et al., 1985). Nya kliniska bevis visar att fekal överflöd av denna art förändras hos barn med autismspektrumstörning; emellertid förblir dess roll i störningen oklar (Wang et al., 2013).
överraskande representerade några av sekvenserna potentiella patogener. Klebsiella lunginflammation är till exempel en opportunistisk mänsklig patogen som finns i cirka 40% av människors och djurs tarmar. Mycobacterium orygis, tidigare kallad oryx bacillus, är medlem i Mycobacterium tuberculosis-komplexet som kan orsaka mänsklig tuberkulos (Dawson et al., 2012). Dessa två arter har rapporterats i obehandlad mjölk men inte koumiss. Därför kan deras närvaro tillskriva föroreningen under konventionell koumiss – produktion, särskilt under icke-eller låg-aseptiska manipulationsbetingelser.
bakteriell metabolism spelar en viktig roll för att bilda koumiss egenskaper och kvalitet. Mikrobiella baserade processer såsom lipolys och proteolys krävs för att syntetisera koumiss aromatiska och smakföreningar (Gesudu et al., 2016). Konsekvent innehöll bakteriemetagenomen sekvenser som potentiellt kodar för laktosnedbrytning och proteolytiska system. Till skillnad från de relativt enkla laktoskataboliska vägarna består LABBPROTEOLYTISKA system av ett varierat utbud av enzymer (Chen et al., 2014). Jämfört med andra detekterade koumiss LAB kännetecknas den dominerande arten L. helveticus med en hög proteolytisk aktivitet (Zhang et al., 2015). De flesta laboratorier har bara ett cellhöljeproteinas som initierar mjölkkaseinhydrolysen, medan L. helveticus innehåller minst två av dessa enzymer, nämligen PrtH och PrtH2 (Zhao et al., 2011). Således kan de höga proportionerna av sekvenser som motsvarar de starka proteolytiska arterna av L. helveticus och proteolysrelaterade gener länka till det relativt höga innehållet av peptider och fria aminosyror i koumiss.
en svårighet i industriell skala koumiss produktion är kontrollen av smakuppfattning, eftersom koumiss traditionellt har gjorts genom naturlig jäsning. Således har det varit svårt att definiera smaken och profilen för viktiga smakkomponenter, särskilt i närvaro av de naturliga föroreningarna. Produktionen av viktiga smakkomponenter är ett resultat av fermentativ och enzymatisk nedbrytning av aminosyror, såsom grenade aminosyror, metionin och aromatiska aminosyror (Ardo, 2006). Exempel på smakkomponenter innefattar aldehyder, organiska syror och estrar, som bildas av transaminas (AT)-väg (Helinck et al., 2004). Denna väg initieras av ett transaminas som katalyserar omvandlingen av en aminosyra till dess motsvarande sackaros-keto-syra (Helinck et al., 2004). Vissa sekvenser i vår dataset motsvarade förmodade grenade kedjor och aromatiska aminosyraaminotransferaser. Särskilt fann vi ett förmodat aromatiskt aminosyraaminotransferas I i kontig av den koumiss-associerade arten, S. macedonicus. Eftersom vårt nuvarande arbete endast kommenterade mikrobiomet i silico, är våra data inte tillräckliga för att visa att dessa identifierade gensekvenser verkligen var funktionella för att producera de ovannämnda koumiss-smakföreningarna. Närvaron av dessa gener tyder ändå på att de är några möjliga kandidater för sådana fermentativa aktiviteter; ändå skulle ytterligare experimentellt arbete krävas för att belysa deras exakta funktionella roller.
dessutom fann vi sekvenser motsvarande andra aminosyraomvandlingsvägar inklusive aminosyralyaser och treoninaldolas. Det tidigare enzymet katalyserar omvandlingen av metionin till metantiol (Irmler et al., 2008), vilket resulterar i dimetyldisulfid och dimetyltrisulfidbildning (Fernandez et al., 2000), medan den senare katalyserar omvandlingen av treonin till glycin och acetaldehyd (Ott et al., 2000). På samma sätt kan bara lokalisering av dessa sekvenser inom koumiss-mikrobiomen inte fungera som bestämda bevis i deras faktiska biologiska roller; framtida experimentell bekräftelse kommer att krävas.
slutsats
föreliggande studie använde en modifierad metagenomicsmetod för att analysera bakteriell mikrobiom av koumiss-prover som samlats in från Mongoliet och Inre Mongoliet i Kina. Vi karakteriserade både fylogena och funktionella metagenomer av de sällsynta arterna i koumiss; och vårt dataset återspeglar egenskaper som är av biotekniskt intresse och potential. Vår studie har för första gången visat möjligheten att införliva encelliga amplifieringstekniker vid detektering av koumiss bakteriell mikrobiota, såväl som mikrobiella föroreningar. De tekniker som utvecklats häri kan användas i framtida studier för att övervaka förändringar i koumiss mikrobiom längs jäsningsprocessen, med fokus på minoritets mikrobiell population. Vidare kan andra omiktekniker, såsom transkriptomik, metabolomik, kopplas till den nuvarande metagenomikanalysen för att bekräfta koumiss-mikrobiomets funktioner och metaboliska kapacitet.
tekniskt sett skulle nästa steg i detta arbete vara att optimera den nuvarande metoden. Till exempel, genom att öka provutspädningen före DNA-amplifiering, kan chansen att avslöja sällsynta och nya bakterier förbättras. Å andra sidan kan en alternativ sekvenseringsteknik som kan generera långa läsningar, såsom Pacific Biosciences single molecule, realtidssekvenseringsteknik, användas för att förbättra genommonteringsprocessen.
Författarbidrag
WZ och HZ utformade studien. WZ, GY, JY och L-YK skrev manuskriptet. QH, WH, WL, BM och TS utförde experiment. WZ och QH analyserade data. Alla författare granskade manuskriptet.
intressekonflikt uttalande
författarna förklarar att forskningen genomfördes i avsaknad av kommersiella eller finansiella relationer som kan tolkas som en potentiell intressekonflikt.
bekräftelse
denna forskning stöddes av National Natural Science Foundation of China (Grant No.31571815).
tilläggsmaterial
Tilläggsmaterialet för denna artikel finns online på: https://www.frontiersin.org/article/10.3389/fmicb.2017.00165/full#supplementary-material
Altschul, S. F., Madden, T. L., Schaffer, A. A., Zhang, J., Zhang, Z., Miller, W., et al. (1997). Gapped BLAST och PSI-BLAST: en ny generation av protein databas sökprogram. Nukleinsyror Res. 25, 3389-3402. doi: 10.1093 / nar / 25.17.3389
PubMed Abstrakt / CrossRef fulltext / Google Scholar
A., A., A., A., A., A., A., A., A., A., A., A., A., A., A., A., A., A., A., A., A., A., A., A., A., A., A., A., A., A., A., A., A., A. (2015). Konstruerade stammar av Streptococcus macedonicus mot en osmotisk stressresistent fenotyp behåller sin förmåga att producera bakteriocin macedocin under hyperosmotiska förhållanden. J. Bioteknol. 212, 125–133. doi: 10.1016 / j. jbiotec.2015.08.018
PubMed Abstrakt / CrossRef Fulltext / Google Scholar
Ardo, Y. (2006). Smakbildning genom aminosyrakatabolism. Bioteknol. Adv. 24, 238-242. doi: 10.1016 / j. biotechadv.2005.11.005
PubMed Abstrakt / CrossRef fulltext / Google Scholar
Chen, Y. F., Zhao, W. J., Wu, R. N., Sun, zh, Zhang, W. Y., Wang, J. C., et al. (2014). Proteomanalys av Lactobacillus helveticus H9 under tillväxt i skummjölk. J. Mejeri Sci. 97, 7413–7425. doi: 10.3168/jds.2014-8520
PubMed Abstrakt / CrossRef fulltext / Google Scholar
Dawson, K. L., Bell, A., Kawakami, R. P., Coley, K., Yates, G. och Collins, D. M. (2012). Överföring av Mycobacterium orygis (M. tuberculosis complex species) från en tuberkulospatient till en mjölkko i Nya Zeeland. J. Clin. Mikrobiol. 50, 3136–3138. doi: 10.1128/JCM.01652-12
PubMed Abstrakt / CrossRef fulltext / Google Scholar
han är en av de mest kända och mest kända i världen. (2013). Utkast till genomsekvens av D-Grenad aminosyraproducent Lactobacillus otakiensis JCM 15040t, isolerad från en traditionell japansk pickle. Genome Announc. 1, e546-e513. doi: 10.1128 / genomeA.00546-13
PubMed Abstrakt / CrossRef fulltext / Google Scholar
han är en av de mest kända och mest kända i världen. (2000). Molekylära och funktionella analyser av metC-genen av Lactococcus lactis, som kodar för cystathionin beta-lyas. Appl. Miljö. Mikrobiol. 66, 42–48. doi: 10.1128 / AEM.66.1.42-48.2000
PubMed Abstrakt / CrossRef fulltext / Google Scholar
I., Carafa, I., Nardin, T., Schiavon, S., Poznanski, E., Cavazza, A., et al. (2015). Biologisk mångfald och gamma-aminosmörsyraproduktion av mjölksyrabakterier isolerade från traditionella alpina råmjölkostar. Biomed Res. Int. 2015, 625740. doi: 10.1155/2015/625740
PubMed Abstrakt / CrossRef fulltext / Google Scholar
Fu, L., Niu, B., Zhu, Z., Wu, S. och Li, W. (2012). CD-HIT: accelererad för att klustera nästa generations sekvenseringsdata. Bioinformatics 28, 3150–3152. doi: 10.1093/bioinformatics/bts565
PubMed Abstract | CrossRef Full Text | Google Scholar
Georgalaki, M. D., Sarantinopoulos, P., Ferreira, E. S., De Vuyst, L., Kalantzopoulos, G., and Tsakalidou, E. (2000). Biochemical properties of Streptococcus macedonicus strains isolated from Greek Kasseri cheese. J. Appl. Microbiol. 88, 817–825. doi: 10.1046/j.1365-2672.2000.01055.x
PubMed Abstract | CrossRef Full Text | Google Scholar
Gesudu, Q. M., Zheng, Y., Xi, X. X., Hou, Q. C., Xie, H. Y. Han är en av de mest kända. (2016). Undersöka bakteriell populationsstruktur och dynamik i traditionell koumiss från Inre Mongoliet med hjälp av en enda molekyl realtidssekvensering. J. Mejeri Sci. 99, 7852–7863. doi: 10.3168/jds.2016-11167
PubMed Abstrakt / CrossRef fulltext / Google Scholar
det finns många olika typer av produkter. (2016). En stor fabrik skala tillämpning av utvalda autoktona mjölksyrabakterier för PDO Pecorino Siciliano ostproduktion. Mat Mikrobiol. 59, 66–75. doi: 10.1016 / j.fm. 2016.05.011
PubMed Abstrakt / CrossRef fulltext / Google Scholar
hao, Y., Zhao, L., Zhang, H., Zhai, Z., Huang, Y., Liu, X., et al. (2010). Identifiering av den bakteriella biologiska mångfalden i koumiss genom denaturering av gradientgelelektrofores och artspecifik polymeraskedjereaktion. J. Mejeri Sci. 93, 1926–1933. doi: 10.3168/jds.2009-2822
PubMed Abstrakt / CrossRef fulltext / Google Scholar
Helinck, S., Le Bars, D., Moreau, D. och Yvon, M. (2004). Förmåga hos termofila mjölksyrabakterier att producera aromföreningar från aminosyror. Appl. Miljö. Mikrobiol. 70, 3855–3861. doi: 10.1128 / AEM.70.7.3855-3861.2004
PubMed Abstrakt / CrossRef fulltext / Google Scholar
Hoskins, L. C., Agustines, M., McKee, W. B., Boulding, E. T., Kriaris, M. och Niedermeyer, G. (1985). Mucin nedbrytning i mänskliga kolon ekosystem. Isolering och egenskaper hos fekala stammar som bryter ned ABH-blodgruppsantigener och oligosackarider från mucinglykoproteiner. J. Clin. Investera. 75, 944–953. doi: 10.1172 / JCI111795
PubMed Abstrakt / CrossRef fulltext / Google Scholar
Irmler, S., Raboud, S., Beisert, B., Rauhut, D. och Berthoud, H. (2008). Kloning och karakterisering av två Lactobacillus casei-gener som kodar för ett cystathioninlyas. Appl. Miljö. Mikrobiol. 74, 99–106. doi: 10.1128 / AEM.00745-07
PubMed Abstrakt / CrossRef Fulltext / Google Scholar
Jagielski, V. (1877). Värdet av koumiss vid behandling av illamående, kräkningar och oförmåga att behålla annan mat i magen. Br. Med. J. 2, 919-921. doi: 10.1136 / bmj.2.887.919
PubMed Abstrakt / CrossRef fulltext / Google Scholar
Kato, S., Ishihara, T., Hemmi, H., Kobayashi, H. och Yoshimura, T. (2011). Förändringar i d-aminosyrakoncentrationer och mikrobiella samhällsstrukturer under jäsning av röda och vita viner. J. Biosci. Bioeng. 111, 104–108. doi: 10.1016/j.jbiosc.2010.08.019
PubMed Abstrakt / CrossRef fulltext / Google Scholar
Li, H. och Durbin, R. (2009). Snabb och exakt kortläsning med Burrows-Wheeler transform. Bioinformatik 25, 1754-1760. doi: 10.1093 / bioinformatik / btp324
PubMed Abstrakt / CrossRef fulltext / Google Scholar
Li, W. och Godzik, A. (2006). Cd-hit: ett snabbt program för att klustera och jämföra stora uppsättningar protein-eller nukleotidsekvenser. Bioinformatik 22, 1658-1659. doi: 10.1093 / bioinformatik / btl158
PubMed Abstrakt / CrossRef fulltext / Google Scholar
Miyamoto, M., Seto, T., Nakajima, H., Burenjargal, S., Gombojav, A., Demberei, S., et al. (2010). Denaturerande gradientgelelektroforesanalys av mjölksyrabakterier och jäst i traditionell mongolisk fermenterad mjölk. Mat Sci. Technol. Res 16, 319-326. doi: 10.3136 / fstr.16.319
CrossRef fulltext / Google Scholar
Mulder, NJ och Apweiler, R. (2008). InterPro-databasen och verktyg för proteindomänanalys. Curr. Protokoll. Bioinformatik Kapitel 2, Enhet2. 7. doi: 10.1002 / 0471250953. bi0207s21
PubMed Abstrakt / CrossRef fulltext / Google Scholar
Noguchi, H., Park, J. och Takagi, T. (2006). Metagen: prokaryot genfynd från miljögenom hagelgevärssekvenser. Nukleinsyror Res. 34, 5623-5630. doi: 10.1093 / nar / gkl723
PubMed Abstrakt / CrossRef fulltext / Google Scholar
nurk, S., Bankevich, A., Antipov, D., Gurevich, A. A., Korobeynikov, A., Lapidus, A., et al. (2013). Montering av encelliga genom och mini-metagenomer från chimära mda-produkter. J. Dator. Biol. 20, 714–737. doi: 10.1089 / cmb.2013.0084
PubMed Abstrakt / CrossRef fulltext / Google Scholar
Ott, A., Germond, J. E. och Chaintreau, A. (2000). Ursprung av acetaldehyd under mjölkfermentering med användning av (13)C-märkta prekursorer. J. Agric. Mat Chem. 48, 1512–1517. doi: 10.1021 / jf9904867
PubMed Abstrakt / CrossRef fulltext / Google Scholar
Pan, dd, Zeng, XQ och Yan, yt (2011). Karakterisering av Lactobacillus fermentum SM-7 isolerad från koumiss, en potentiell probiotisk bakterie med kolesterolsänkande effekter. J. Sci. Mat. Agric. 91, 512–518. doi: 10.1002/jsfa.4214
PubMed Abstrakt / CrossRef Fulltext / Google Scholar
Papadimitriou, K. Det är en av de mest populära och mest populära. (2015). Förvärv genom horisontell genöverföring av plasmid pSMA198 av Streptococcus macedonicus ACA-DC 198 pekar mot artens mejeriursprung. PLoS EN 10: e0116337. doi: 10.1371 / tidskrift.pone.0116337
PubMed Abstrakt / CrossRef fulltext / Google Scholar
Segata, N., Waldron, L., Ballarini, A., Narasimhan, V., Jousson, O. och Huttenhower, C. (2012). Metagenomic microbial gemenskapsprofilering genom att använda unika clade-specifika markörgener. Nat. Metoder 9, 811-814. doi: 10.1038 / nmeth.2066
PubMed Abstrakt / CrossRef fulltext / Google Scholar
Sun, Z., Liu, W., Zhang, J., Yu, J., Zhang, W., Cai, C., et al. (2010). Identifiering och karakterisering av de dominerande laktobacillerna isolerade från koumiss i Kina. J. Gen.Appl. Mikrobiol. 56, 257–265. doi: 10.2323/jgam.56.257
PubMed Abstrakt / CrossRef Fulltext / Google Scholar
Thompson, J. (1879). Värdet av koumiss i slöseri med sjukdomar. Br. Med. J. 1, 270. doi: 10.1136 / bmj.1.1466.270-c
PubMed Abstrakt / CrossRef fulltext / Google Scholar
Vincent, sj, Faber, Ej, Neeser, Jr, Stingele, F. och Kamerling, J. P. (2001). Struktur och egenskaper hos exopolysackariden producerad av Streptococcus macedonicus Sc136. Glykobiologi 11, 131-139. doi: 10.1093 / glycob / 11.2.131
PubMed Abstrakt / CrossRef fulltext / Google Scholar
Wang, L., Christophersen, C. T., Sorich, M. J., Gerber, J. P., Angley, M. T. och Conlon, MA (2013). Ökad överflöd av Sutterella spp. och Ruminococcus vridmoment i avföring hos barn med autismspektrumstörning. Mol. Autism 4, 42. doi: 10.1186/2040-2392-4-42
PubMed Abstrakt / CrossRef fulltext / Google Scholar
Ward, TL, Hosid, S., Ioshikhes, I. och Altosaar, I. (2013). Human milk metagenome: en funktionell kapacitetsanalys. BMC Microbiol. 13:116. doi: 10.1186/1471-2180-13-116
PubMed Abstrakt / CrossRef fulltext / Google Scholar
han är en av de mest kända och mest kända i världen. (2009). Lactobacillus kisonensis sp. nov., Lactobacillus otakiensis sp. nov., Lactobacillus rapi sp. nov. och Lactobacillus sunkii sp. nov., heterofermentativa arter isolerade från sunki, en traditionell japansk pickle. Int. J. Syst. Evol. Mikrobiol. 59 (Pt 4), 754-760. doi: 10.1099/ijs.0.004689-0
PubMed Abstrakt / CrossRef fulltext / Google Scholar
Wu, R., Wang, L., Wang, J., Li, H., Menghe, B., Wu, J., et al. (2009). Isolering och preliminärt probiotiskt urval av laktobaciller från koumiss i Inre Mongoliet. J. Grundläggande Mikrobiol. 49, 318–326. doi: 10.1002/jobm.200800047
PubMed Abstrakt / CrossRef fulltext / Google Scholar
Zerbino, D. R. och Birney, E. (2008). Sammet: algoritmer för de novo kortläst montering med hjälp av de Bruijn grafer. Genomet Res. 18, 821-829. doi: 10.1101 / gr.074492.107
PubMed Abstrakt / CrossRef fulltext / Google Scholar
Zhang, W., Sun, Z., Sun, T. och Zhang, H. (2010a). PCR-screening och sekvensanalys av iol-kluster i Lactobacillus casei-stammar isolerade från koumiss. Folia Microbiol. (Praha) 55, 603-606. doi: 10.1007 / s12223-010-0097-3
PubMed Abstrakt / CrossRef fulltext / Google Scholar
Zhang, W., Yu, D., Sun, Z., Wu, R., Chen, X., Chen, W., et al. (2010b). Komplett genomsekvens av Lactobacillus casei Zhang, en ny probiotisk stam isolerad från traditionell hemlagad koumiss i Inre Mongoliet, Kina. J. Bakteriol. 192, 5268–5269. doi: 10.1128 / JB.00802-10
PubMed Abstrakt / CrossRef fulltext / Google Scholar
Zhang, W. och Zhang, H. (2011). ”Jäsning och koumiss,” i Handbok för mat och dryck Fermentation Technology, 2nd Edn, ed. Y. H. Hui (Boca Raton, FL: CRC Press), 165-172.
Google Scholar
Zhang, wy, Chen, YF, Zhao, WJ, Kwok, Ly och Zhang, HP (2015). Genuttryck av proteolytiskt system av Lactobacillus helveticus H9 under mjölkfermentering. Ann. Mikrobiol. 65, 1171–1175. doi: 10.3168/jds.2014-8520
PubMed Abstrakt / CrossRef fulltext / Google Scholar
Zhao, W., Chen, Y., Sun, Z., Wang, J., Zhou, Z., Sun, T., et al. (2011). Komplett genomsekvens av Lactobacillus helveticus H10. J. Bakteriol. 193, 2666–2667. doi: 10.1128 / JB.00166-11
PubMed Abstrakt / CrossRef Fulltext / Google Scholar