はじめに
コウミスマイクロバイオームは、モンゴル語でchige、chigo、arrag、airagとも呼ばれる伝統的な発酵乳製品の一種である。 それは何世紀にもわたって中国のモンゴルと内蒙古で人気のある食べ物でした(Zhang and Zhang、2011)。 これらの地域の人々は、壮大な祭りや犠牲の供物の間にkoumissを消費していました(Zhang and Zhang、2011)。 Koumissの生産の最も早い記録は漢王朝(BC202-AD202)にさかのぼることができます。 この製品は元朝(AD1271-AD1368)の間に広範な人気を得ていました(Zhang et al。,2010b). 今日では、クミスはモンゴルと内モンゴル自治区の地元の人々のための一般的な食品ですが、これらの地域のいくつかでのみ、工業規模で製造されています。 Koumissは必須アミノ酸およびビタミンの高い内容を含む豊富な栄養素を、だけでなく、提供しますが、また病状の広い範囲を取り除くと信じられ、postoperative心配(Jagielski、1877;Thompson、1879)
伝統的に、koumissは一般的に木製の樽、動物の皮や壷で作られた容器で生産されています。 発酵は、濾過された牝馬ミルクをスターター培養物として機能する古いkoumissを用いて容器に添加した後、周囲温度で自然に起こる(Zhang and Zhang、2011)。 Koumissは、バイオテクノロジーの可能性のある新規細菌の良好な供給源である(Zhang et al. ら、2 0 1 0a;Pan e t a l., 2011). したがって、できるだけ多くの発酵関連koumiss細菌を探索し、保存することは非常に興味深いものです。 過去数十年の間に、koumiss細菌群集を調査するために多くの研究が行われた(Wu et al. ら,2 0 0 9;Hao e t a l. 主に培養法、分子生物学およびパイロシーケンスに基づく方法によって研究されている(Sun et al.,2010)。, 2010). これらの異なるアプローチの中で、パイロシーケンスベースの方法は、表現型形質と個々の微生物の栽培性の問題から独立したkoumissの最も包括的な微生物相プロ しかし、koumiss細菌によってコードされた機能的遺伝子のスペクトルとその発酵能力は、特にまれな微生物集団のために、不十分な特徴を残します。
本研究では、中国のモンゴルと内モンゴルから収集した10koumissサンプルの細菌メタゲノムを分析するために、単一細胞ゲノミクス技術を使用しました。 現在の研究では、乳製品の細菌の多様性を調査する際に最先端の技術を適用しています。 我々の研究は、単一細胞メタゲノミクスアプローチを適用することにより、低豊富な分類群を発見する可能性を実証しています。 奨励された結果は、従来の研究分野における問題に取り組むための新しいアプローチの開発と適用を促進するだろう。
材料と方法
サンプルの調製
メタゲノミクス研究のためにモンゴル(MG14、MG15、MG16、MG17、およびMG18)と中国の内モンゴル(NM17、NM18、NM19、NM20、およびNM21)から合計10 試料を無菌的に収集し,ドライアイス中で輸送した。
各サンプルの1ミリリットルを、Ward et al. (2013)いくつかの変更を加えた。 簡単に言えば、試料を氷浴中で3〜5分間解凍した。 試料を溶融した後、それらを低速遠心分離に供して、不純物および真核細胞塊を除去した。 次いで、原核生物細胞を、1 3,0 0 0×gで1 5分間の遠心分離によって乳血清からペレット化した。 ペレットを1%Triton X−1 0 0で2mlのリン酸緩衝生理食塩水(PBS)中に再懸濁し、残りの真核細胞を溶解するために3 7℃で2時間インキュベートした。 続いて、細菌を1 3,0 0 0×gで1 5分間の遠心分離によってペレット化し、ペレットを5 0 0μ lのPBS中に再懸濁した。 最後に、遠心分離ステップをもう一度繰り返し、細菌細胞を洗浄した。
勾配希釈および多重変位増幅
低豊富な細菌を検出するために、各koumissサンプル由来の細菌懸濁液を、その後の増幅反応のために連続的に希釈した。 各試料中の細胞数を、細胞計数チャンバ(Qiujing、上海、中国)を用いて顕微鏡(Nikon、東京、日本)下で大まかに推定した。 希釈工程は、各細菌懸濁液中の細胞数が約1 0 0に達するまで継続した。 希釈された細胞の多重変位増幅を、REPLI−g Single Cell Kit(Qiagen,Germantown,MD,USA)を使用して、製造業者の説明書に従って行った。
ライブラリーの構築と配列決定
増幅されたDNAをランダムに剪断し、約500bpの断片を選択した。 ライブラリの構築後、PerkinElmer LabChip®GX TouchおよびStepOnePlusTM Real-Time PCR Systemをライブラリ品質検査に使用しました。 最後に、製造業者の指示に従って、125bpペアエンド読み取りをIllumina HiSeq2500プラットフォーム上で配列決定しました。
データ分析
シーケンス品質チェックとフィルタリング
シーケンサによって生成された生の読み取りには、ライブラリ構築中にアダプタ汚染の人為的な読み取りが含まれている可能性があります。 したがって、高品質のクリーンリードデータセットを得るために三つのステップを実行しました:(1)アダプタ汚染に起因する読み取りの除去; (2)高品質のベースの最低カットオフと考えられていたQ30のphredスコアを下回る平均スコアを持つ読み取りの除去;(3)”N”の有意な過剰(読み取りの≥5%)を 下流の分析は、クリーンデータに基づいていました。 さらに、Q3 0およびGC含有量に基づいて、統計的基準品質を計算した。
宿主ゲノムに対するアラインメントを行い、宿主由来の汚染配列を除去した。 任意の宿主由来の読み取りは、細菌(またはウイルス)ゲノム参照配列とのさらなる比較の前に廃棄された。 より正確な結果を得るために、Burrows–Wheeler aligner(BWA)(バージョン0.97a)MEMモデルを整列に使用した(Li and Durbin、2009)。
分類学的割り当てと多様性
ウェブソフトウェアMetaphlanは、属および種レベルへの分類学的割り当てに使用されました(Segata et al., 2012). サンプル内およびサンプル間の種の多様性を比較するために、我々は、r関連パッケージによってアルファとベータの多様性を分析しました。
Read Assembly,Gene Prediction,And Annotation
より包括的な情報を得るために、せん断された断片をゲノム(contigs)に組み立てました。 しかし、メタゲノムサンプルで一般的である複数の種の存在のために、我々はスペード(バージョン3.6.2)、社内スクリプト、およびメタゲノムデータベースを統合す, 2013).
MetaGeneMarkソフトウェアは、組み立てられたcontigsの遺伝子予測に使用されました(Noguchi et al., 2006). 冗長遺伝子を、9 0%および9 5%の同一性のカバレッジを有するCD−HITを用いて除去した(LiおよびGodzik、2 0 0 6;Fu e t a l., 2012). 遺伝子の相対的存在量は、同じ手順を使用して、高品質の配列決定読み取りを遺伝子カタログに整列させることによって決定された。 下流の不一致分析は、遺伝子の相対的存在量に基づいていた。 遺伝子注釈は、いくつかの公共のデータベース(すなわちNCBI非冗長データベース、NR;タンパク質のOrthologousグループのクラスター、COGs)に対して高品質の配列を整列させることに; KYOTO Encyclopedia o f Genes and Genomes,KEGG)using BLAST(Altschul e t a l., 1997). ドメイン検索は、Interproscan(Mulder and Apweiler,2008)を使用して実行されました。
塩基配列アクセッション番号
本研究で報告された配列データは、SRAデータベースに寄託されています(Accession No.SRP083102)。
結果
実験デザインと配列決定
koumissの低豊富な細菌を検出するために、単一細胞増幅技術を使用してサンプルのメタゲノムを分析しました。 三つの細菌懸濁液、約100細胞のそれぞれは、シリアル希釈によって独立したkoumissサンプルから誘導されました。 合計3 0個の希釈懸濁液を分析した。 各希釈サンプルには、異なるサンプルコード、すなわち、サンプル同一番号の後に1、2、または3が続き、3つの別々の希釈を表すものが与えられた。 いくつかの希少種が希釈のいずれかに存在するという前提に基づいて、細胞の複数の変位増幅が行われ、約5Gbのデータがすべてのkoumiss細菌懸濁液につ
10個のkoumissサンプル(合計30個の細菌懸濁液)から合計1,040,323,864個の生の読み取りが生成されました。 各100細胞懸濁液の読み取りの平均数は34,677,462であった(補足表S1)。 修飾されていないシーケンスのトリミングとフィルタリングの後、すべてのサンプルの1,018,381,702クリーンリードを取得しました(補足表S1)。 シャノン指数、シンプソン指数、Chao1指数、観察種数(図1-4)の値は、ほとんどのkoumissサンプルが高い細菌の生物多様性を持っていたことを示した。 Shannon-Wiener多様性曲線は、シーケンスの深さがすべてのサンプルに対して適切であることを示しました(図1)。
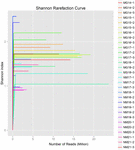
図1. Koumissのサンプルの微生物多様性を推定するShannonの希薄化のカーブ。
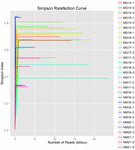
図2. Koumissのサンプルの微生物多様性を推定するSimpsonの希薄化のカーブ。
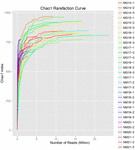
図3. Koumissサンプルの微生物多様性を推定するchao1希薄化曲線。
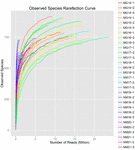
図4. Koumissサンプルの微生物多様性を推定する観察された種の希薄化指数。
分類学的注釈
高品質の配列は、サンプル細菌群集の詳細な分析を可能にするために、異なる分類学的レベルに割り当てられました。 Koumissの生物多様性に関するいくつかの出版された研究を参照して(Wu et al. ら,2 0 0 9;Hao e t a l. ら、2 0 1 0;Sun e t a l.、2010)、我々はそれぞれ、一般的かつ希少な分類群として知られており、以前に報告されていないkoumiss関連細菌を分類しました。
高品質の配列は13の異なる属を表していました(図5)。 そのうちの三つは、ラクトバチルス(L.)、ラクトコッカス、および連鎖球菌を含む1%以上の平均相対存在量を有していた。 特に,Lactobacillus属とLactococcus属はkoumissに見られる二つの最も豊富な属であった。 サンプル中の乳酸菌の割合は52.72から99.96%の範囲であった。 この属の二つのメンバー、L.helveticusとL.kefiranofaciensは、ほとんどのkoumissサンプルの間で支配されていた(図6)。 種L. buchneriは主にモンゴルの試料に存在したが,lactococcuslactisはサンプリング領域に関係なくほとんどの試料に検出された。
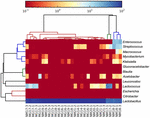
図5. 属レベルでkoumissサンプルで検出された細菌の相対的な存在量を示すヒートマップ。
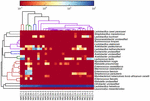
図6. 種レベルでkoumissサンプルで検出された細菌の相対的な存在量を示すヒートマップ。
メタゲノムアセンブリ、遺伝子予測、および機能的注釈
読み取りの組み立ては、614,392,623bpの組立長をもたらした。 アセンブリのN50値は5,596から35,200bpの範囲であった(補足表S2)。 Koumissサンプルにおける予測遺伝子の数は10,347から34,547の範囲であり、平均長さは647.82から985.33bp(補足表S3)であった。 経験的遺伝子機能解析は、現在の研究の範囲を超えていたが、我々は主に配列相同性に基づいて遺伝子機能を予測したCOGとKEGGデータベースを使用してkoumiss細菌
注釈出力の合計545マッチは、乳糖利用遺伝子(炭水化物および代謝のCOGsカテゴリ、G)と高い相同性を示し、そのうちのいくつかは同じコンティグ内に位置していた(表1)。
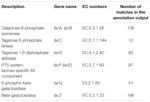
表1. ラクトース代謝関連遺伝子は、koumiss細菌メタゲノムに注釈されている。
いくつかの他の配列は、カゼイン分解プロテイナーゼに対応する配列、4-18残基のオリゴペプチドを取るためのOppシステム、およびアミノペプチダーゼ(例えば、ロイシルアミノペプチダーゼ、ペプチジル-ジペプチダーゼA、アミノペプチダーゼN、プロリンイミノペプチダーゼ、およびエンドペプチダーゼに対応する配列を含む、アミノ酸輸送および代謝(E)のCOGsカテゴリ内の推定遺伝子をコードする可能性がある。)。 いくつかの配列は、アルギニン、アスパラギン酸、メチオニン、およびイソロイシンに特異的なアミノトランスフェラーゼと高い類似性を共有していたが、唯一の重要な一致は、推定S.macedonicus由来のクラスI/クラスIIドメイン(IPR004839)に対応し、同定された-チロシンとフェニルアラニンに特異的なアミノトランスフェラーゼを含む。 最後に,s-リボシルホモシステインリアーゼ,アルギニノコハク酸リアーゼ,アスパラギン酸アンモニア-リアーゼ,シスタチオニンγ-リアーゼ,ヒスチジンアンモニア-リアーゼ,O-アセチルホモセリン(チオール)-リアーゼを含むアミノ酸リアーゼと高い相同性を共有した。
ディスカッション
Koumissは、中国のモンゴルと内モンゴルで人気のある伝統的な発酵乳製品です。 Koumissの細菌の多様性を調査するためにいくつかの調査が行われたが、ほとんど情報はkoumissの微生物叢の遺伝的能力に関する得られなかった。 ここでは、特に低豊富な細菌集団に焦点を当て、koumiss細菌メタゲノムを分析するために単一細胞増幅技術を適用しました。
典型的なメタゲノミクスアプローチは、以前にkoumiss微生物叢を記述するために適用されています。 しかし、深い配列を達成するためのコストが高いため、サンプル中のまれな微生物叢の集団はしばしば不十分に覆われている。 したがって、少数の細菌の系統発生および機能的メタゲノムの両方が限られたままである。 私たちの単一細胞増幅法は、100細胞懸濁液にサンプルのシリアル希釈を関与していました。 希釈されたkoumissサンプル中に存在する細胞の数が少ないため、限られた量のDNAのみが抽出される。 増幅ステップは分析すべきDNA材料の量を増加させ,微小DNA量を含む試料のメタゲノーム分析を容易にした。 この方法の一つの欠点は、各分類群の配列がプロセスで均等に増幅されることを保証することの難しさであった; したがって、ここで得られた結果は、配列の相対的存在量のみを反映することができ、同定された分類群または機能的遺伝子の絶対量は反映しない。 しかし、本研究はまれな細菌集団を集中させる上で他の出版された研究とは異なるので、これは我々の分析にほとんど影響を及ぼさないであろう。 この作業によって生成されたデータは、過小評価された人口に補完的な情報を提供します。 このアプローチは,異なるタイプの生態環境に対する細菌の多様性の将来の分析に適していると考えている。
koumiss細菌の微生物叢は、主に乳酸菌(LAB)といくつかの酢酸菌で構成されています(Zhang and Zhang、2011)。 予想通り、私たちのデータセットには、主に実験室と酢酸菌を表す配列が含まれていました。 Lactobacillus helveticusに対応する配列は、すべてのサンプルにわたって支配していた。 また、種を表す配列、L.kefiranofaciens。 L.buchneri。 L.kefiranofaciens。 エンテロコッカス(E.)casseliflavus。 E.faecalis。 E.faecium。 ロイコノストック-メセンテロイデス Lactococcuslactis,Acetobacterpasteurianusも発見した。 異なる分類群の配列の同定は、細菌の生存率を示すのに十分ではないかもしれないが、それにもかかわらず、発酵プロセスのある時点での細菌群集を 宮本他 (2010)は、最終発酵生成物中の細菌の有病率がそれらの酸ストレス耐性に関連していることを示唆している。 一般に、乳酸桿菌は乳酸球菌よりも高い酸耐性を有し、これは我々のデータセットに存在する乳酸桿菌配列の高い相対的存在量を説明する可能性がある。 一方、Lの頻繁な発生。 koumissにおけるhelveticusはL.helveticus配列の優位性の観察と一致した。
我々のデータセットでは、いくつかのシーケンスは、koumissまたは他の乳製品のニッチで報告されていない希少種であるL.otakiensisに対応していました。 この種を最初に記載し,日本の伝統的なピクルスであるサンキの製造に使用される非塩漬けピクルス溶液から単離した(Watanabe et al., 2009). Reca遺伝子に基づく増幅断片長多型プロファイリングにより発見された。 それ以来、この種は他の食物関連のニッチと関連していることは報告されていない。 したがって、この細菌はピクルスの自生植物に属している可能性が高い。 しかし、採用された検出方法の感度だけでは見つからなかった可能性を排除することはできません。 Lactobacillus otakiensisは、d−ロイシン、d−アロ−イソロイシンおよびd−バリンなどのd−分枝鎖アミノ酸を産生することができる(Doi e t a l., 2013). それはある特定の発酵食品の生産の特徴の改善で使用される潜在性を有します(Kato et al., 2011).
我々の研究では、koumiss関連細菌として報告されたことのない種S.macedonicusを表す配列も同定されました。 代わりに、それはギリシャの羊およびヤギのチーズに存在するスターター培養物である(Georgalaki et al., 2000). この種のいくつかのメンバーは、外多糖類、バクテリオシンを産生することができる(Vincent et al. ら,2 0 0 1;Anastasiou e t a l. ら、2 0 1 5)、およびγ−アミノ酪酸(Franciosi e t a l., 2015). この種は発酵食品から頻繁に単離されているにもかかわらず、S.macedonicusの元のニッチは議論の余地がある(Guarcello et al., 2016). 最近まで、Papadimitriou et al. ら(2 0 1 5)は、Lactococcus lactisから取得したプラスミドpsma1 9 8を同定した。 このプラスミドは、乳製品環境内で祖先の遺伝的交換事象を介して移動された可能性が高く、s.macedonicusの乳製品起源を示唆している(Papadimitriou e t a l., 2015). S.thermophilusに似ています。 S.macedonicusはs.bovis/s.equinus複合体の共生および日和見病原体と密接に関連している。
この細菌は通常腸内環境で見られるため、種Ruminococcus torquesを表す配列の同定は予期せぬものでした。 これは、分泌グリコシダーゼの構成的産生によってムチンオリゴ糖を分解することができる正常なヒト腸微生物である(Hoskins et al., 1985). 最近の臨床的証拠は、この種の糞便量が自閉症スペクトラム障害を有する小児で変化することを示しているが、障害におけるその役割は不明である(Wang et al., 2013).
驚くべきことに、いくつかの配列は潜在的な病原体を表していました。 例えば、クレブシエラ肺炎は、ヒトおよび動物の腸の約40%に存在する日和見的なヒト病原体である。 Mycobacterium orygisは、以前はoryx bacillusと呼ばれていましたが、ヒト結核を引き起こす可能性のある結核菌複合体のメンバーです(Dawson et al., 2012). これら二つの種は生乳で報告されているが、koumissでは報告されていない。 したがって、それらの存在は、特に非または低無菌操作条件下で、従来のkoumiss生産中の汚染に起因する可能性があります。
工業規模のコウミスの生産の難しさの一つは、コウミスが伝統的に自然発酵によって作られてきたように、味の知覚の制御です。 したがって、特に天然の汚染物質の存在下では、風味および主要な風味成分のプロファイルを定義することは困難であった。 主要な香味成分の生産は、分岐鎖アミノ酸、メチオニンおよび芳香族アミノ酸などのアミノ酸の発酵および酵素分解の結果である(Ardo、2006)。 香味成分の例としては、トランスアミナーゼ(A T)経路によって形成されるアルデヒド、有機酸およびエステルが挙げられる(Helinck e t a l., 2004). この経路は、アミノ酸の対応するα−ケト酸への変換を触媒するトランスアミナーゼによって開始される(Helinck e t a l., 2004). 我々のデータセットのいくつかの配列は、推定分岐鎖および芳香族アミノ酸アミノトランスフェラーゼに対応していた。 特に,koumiss関連種S.macedonicusのコンティグにおいて,推定的な芳香族アミノ酸アミノトランスフェラーゼiを見出した。 我々の現在の研究は、silicoでマイクロバイオームに注釈を付けただけなので、我々のデータは、これらの同定された遺伝子配列が実際に前述のkoumissフレーバー化合物を産生するのに機能的であったことを示すのに十分ではない。 それにもかかわらず、これらの遺伝子の存在は、彼らがそのような発酵活性のためのいくつかの可能な候補であることを示唆している;まだ、さらなる実験的研究は、それらの正確な機能的役割を解明するために必要となるであろう。
さらに、アミノ酸リアーゼやスレオニンアルドラーゼなど、他のアミノ酸変換経路に対応する配列を発見しました。 前者の酵素は、メチオニンのメタンチオールへの変換を触媒する(Irmler et al. る(Fernandez e t a l.,2 0 0 8)。 ら、2 0 0 0)、一方、後者は、スレオニンのグリシンおよびアセトアルデヒドへの変換を触媒する(Ott e t a l.,2 0 0 0)。, 2000). 同様に、これらの配列をkoumissマイクロバイオーム内に配置するだけでは、実際の生物学的役割における明確な証拠として役立つことはできません。
結論
本研究では、中国のモンゴルと内モンゴルから収集したkoumissサンプルの細菌マイクロバイオームを分析するために修正メタゲノミクス法を使用しました。 我々はkoumissの希少種の系統発生と機能メタゲノムの両方を特徴とし、我々のデータセットは、バイオテクノロジーの関心と可能性のある形質を反映しています。 我々の研究は、初めてkoumiss細菌微生物叢だけでなく、微生物汚染物質を検出する際に単一細胞増幅技術を組み込むことの実現可能性を実証しています。 ここで開発された技術は、少数の微生物集団に焦点を当てて、発酵プロセスに沿ったkoumiss微生物叢の変化を監視するために将来の研究に使用すること さらに、転写、代謝学などの他のオミックス技術は、koumissマイクロバイオームの機能と代謝能力を確認するために、現在のメタゲノミクス分析に結合すること
技術的には、この作業の次のステップは、現在の方法を最適化することです。 例えば、DNA増幅の前に試料希釈を増加させることによって、希少で新規な細菌を発見する機会を改善することができる。 一方、pacific Biosciences single molecule、real-time sequencing technologyなどの長い読み取りを生成することができる代替の配列決定技術を用いて、ゲノム組立プロセスを改善することができる。
著者の貢献
WZとHZが研究を設計しました。 WZ、GY、JY、L-YKが原稿を書いた。 QH、WH、WL、BM、およびTSは実験を行った。 WZとQHはデータを分析しました。 すべての著者が原稿をレビューしました。
利益相反声明
著者らは、この研究は利益相反の可能性と解釈される可能性のある商業的または財政的関係がない場合に行われたと宣言している。
謝辞
この研究は、中国国家自然科学財団(助成第31571815号)によって支援されました。
補足資料
この記事の補足資料はオンラインで見つけることができます: https://www.frontiersin.org/article/10.3389/fmicb.2017.00165/full#supplementary-material
Altschul,S.F.,Madden,T.L.,Schaffer,A.A.,Zhang,J.,Zhang,Z.,Miller,W.、ら。 (1997). Gapped BLASTおよびPSI-BLAST:タンパク質データベース検索プログラムの新世代。 核酸Res.25,3389-3402. doi:10.1093/nar/25.17.3389
PubMed要約/CrossRef全文/Google Scholar
Anastasiou,R.,Driessche,G.V.,Boutou,E.,Kazou,M.,Alexandraki,V.,Vorgias,C.E.,et al. (2015). 浸透圧ストレス耐性表現型に向かってストレプトコッカスmacedonicusの設計株は、高浸透圧条件下でバクテリオシンmacedocinを産生する能力を保持します。 J.Biotechnol. 212, 125–133. 土井:10.1016/j.jbiotec.2015.08.018
PubMed要約/CrossRef全文/Google Scholar
アルド,Y.(2006). アミノ酸の異化による味の形成。 バイオテクノール 24,238-242 土井:10.1016/j.biotechadv.2005.11.005
PubMed抄録/CrossRef全文/Google Scholar
Chen,Y.F.,Zhao,W.J.,Wu,R.N.,Sun,Z.H.,Zhang,W.Y.,Wang,J.C.,et al. (2014). 脱脂乳中の成長中のLactobacillus helveticus H9のプロテオーム解析。 J.Dairy Sci. 97, 7413–7425. ドイ:10.3168/jds.2014-8520
PubMed要約/CrossRef全文/Google Scholar
Dawson,K.L.,Bell,A.,Kawakami,R.P.,Coley,K.,Yates,G.,And Collins,D.M.(2012). 結核患者からニュージーランドの乳牛へのMycobacterium orygis(M.tuberculosis complex種)の伝達。 J.Clin. ミクロビオール 50, 3136–3138. 土井:10.1128/JCM.01652-12
PubMed抄録/CrossRef全文/Google Scholar
ドイ,K.,森,K.,Mutaguchi,Y.,田代,K.,藤野,Y.,大森,T.,et al. (2013). 日本の伝統的なピクルスから単離されたD分岐鎖アミノ酸生産者Lactobacillus otakiensis JCM15040Tのドラフトゲノム配列。 ゲノムアナウンサー 1、e546-e513。 土井:10.1128/genomeA.00546-13
PubMed Abstract/CrossRef Full Text|Google Scholar
Fernandez,M.,van Doesburg,W.,Rutten,G.A.,Marugg,J.D.,Alting,A.C.,van Kranenburg,r.,et al. (2000). シスタチオニンβ-リアーゼをコードするLactococcuslactisのmetc遺伝子の分子および機能解析。 Appl. エンビロン ミクロビオール 66, 42–48. ドイ:10.1128/AEM。66.1.42-48.2000
PubMed Abstract/CrossRef Full Text|Google Scholar
Franciosi,E.,Carafa,I.,Nardin,T.,Schiavon,S.,Poznanski,E.,Cavazza,A.,et al. (2015). 伝統的な高山生の牛乳チーズから単離された乳酸菌による生物多様性とγ-アミノ酪酸生産。 Biomed Res.Int. 2015, 625740. 土井: 10.1155/2015/625740
PubMed Abstract|CrossRef全文|Google Scholar
Fu,L.,Niu,B.,Zhu,Z.,Wu,S.,And Li,W.(2012). CD-HIT:次世代シーケンシング-データをクラスタリングするために加速されます。 Bioinformatics 28, 3150–3152. doi: 10.1093/bioinformatics/bts565
PubMed Abstract | CrossRef Full Text | Google Scholar
Georgalaki, M. D., Sarantinopoulos, P., Ferreira, E. S., De Vuyst, L., Kalantzopoulos, G., and Tsakalidou, E. (2000). Biochemical properties of Streptococcus macedonicus strains isolated from Greek Kasseri cheese. J. Appl. Microbiol. 88, 817–825. doi: 10.1046/j.1365-2672.2000.01055.x
PubMed Abstract | CrossRef Full Text | Google Scholar
Gesudu, Q. M., Zheng, Y., Xi, X. X., Hou, Q. C., Xie, H. Y. ら、huang,W. (2016). 単一分子リアルタイム配列を用いて、内モンゴルの伝統的なkoumissにおける細菌集団の構造とダイナミクスを調査する。 J.Dairy Sci. 99, 7852–7863. ドイ:10.3168/jds.2016-11167
PubMed要約/CrossRef全文/Google Scholar
Guarcello,R.,Carpino,S.,Gaglio,R.,Pino,A.,Rapisarda,T.,Caggia,C.,et al. (2016). PDO Pecorino Sicilianoのチーズ生産のための指定自生の乳酸菌の大きい工場スケールの適用。 食品マイクロバイオール。 59, 66–75. doi:10.1016/j.fm.2016.05.011
PubMed要約/CrossRef全文/Google Scholar
Hao,Y.,Zhao,L.,Zhang,H.,Zhai,Z.,Huang,Y.,Liu,X.,et al. (2010). 変性勾配ゲル電気泳動および種特異的ポリメラーゼ連鎖反応によるkoumissにおける細菌の生物多様性の同定。 J.Dairy Sci. 93, 1926–1933. ドイ:10.3168/jds.2009-2822
PubMed要約/CrossRef全文/Google Scholar
ることができると考えられています。 好熱性乳酸菌がアミノ酸から芳香化合物を産生する能力。 Appl. エンビロン ミクロビオール 70, 3855–3861. ドイ:10.1128/AEM。70.7.3855-3861.2004
PubMed要約|CrossRef全文|Google Scholar
とができるようにすることを目的としています。 ヒト結腸生態系におけるムチン分解。 ムチン糖タンパク質からのABH血液型抗原およびオリゴ糖を分解する糞便株の単離および特性。 J.Clin. 投資する。 75, 944–953. 土井:10.1172/JCI111795
PubMed要約/CrossRef全文/Google Scholar
Irmler,S.,Raboud,S.,Beisert,B.,Rauhut,D.,And Berthoud,H.(2008). シスタチオニンリアーゼをコードする二つの乳酸菌casei遺伝子のクローニングとキャラクタリゼーション。 Appl. エンビロン ミクロビオール 74, 99–106. ドイ:10.1128/AEM。00745-07
PubMed要約/CrossRef全文/Google Scholar
ヤギェルスキV.(1877年)。 吐き気、嘔吐、および胃の上に他の食物を保持することができないことの治療におけるkoumissの価値。 Br. メッド J.2,919-921. 土井:10.1136/2.887.919
PubMed要約/CrossRef全文/Google Scholar
2011年12月28日、加藤・石原・辺見・小林・吉村らによる研究発表が行われた。 赤および白ワインの発酵中のD-アミノ酸濃度および微生物群集構造の変化。 J.Biosci. バイオエン… 111, 104–108. 土井:10.1016/j.jbiosc.2010.08.019
Pubmed抄録/CrossRef全文/Google Scholar
ら,2 0 0 9)。 Burrows-Wheeler transformを使用した高速で正確な短読アライメント。 バイオインフォマティクス25,1754-1760. doi:10.1093/bioinformatics/btp324
PubMed要約/CrossRef全文/Google Scholar
ら(2 0 0 6)。 Cd-hit:タンパク質またはヌクレオチド配列の大規模なセットをクラスタリングし、比較するための高速プ バイオインフォマティクス22,1658-1659. doi:10.1093/バイオインフォマティクス/btl158
PubMed要約/CrossRef全文/Google Scholar
本研究では、以下の研究成果を発表しています。 (2010). モンゴルの伝統的な発酵乳中の乳酸菌および酵母の変性勾配ゲル電気泳動分析。 食品科学. テクノル Res.16,319-326. ドイ:10.3136/fstr。16.319
CrossRef全文|Google Scholar
ることを示した。 蛋白質の範囲の分析のためのInterProのデータベースそして用具。 カー プロトコ バイオインフォマティクス第2章、Unit2.7。 doi:10.1002/0471250953.bi0207s21
PubMed要約/CrossRef全文/Google Scholar
Noguchi,H.,Park,J.,およびTakagi,T.(2006). メタゲン: 環境ゲノムショットガン配列からの原核生物の遺伝子発見。 核酸Res.34,5623-5630. doi:10.1093/nar/gkl723
PubMed要約/CrossRef全文/Google Scholar
Nurk,S.,Bankevich,A.,Antipov,D.,Gurevich,A.A.,Korobeynikov,A.,Lapidus,a.,et al. (2013). キメラMDA製品から単細胞ゲノムとミニメタゲノムを組み立てる。 J.Comput. バイオル 20, 714–737. ドイ:10.1089/cmb.2013.0084
PubMed要約/CrossRef全文/Google Scholar
Ott,A.,Germond,J. (2000). (13)C標識前駆体を用いた牛乳発酵中のアセトアルデヒドの起源。 J.アグリッチ。 食べ物ケム。 48, 1512–1517. doi:10.1021/jf9904867
PubMed要約/CrossRef全文/Google Scholar
Pan,D.D.,Zeng,X.Q.,And Yan,Y.T.(2011). コレステロール低下効果を有する潜在的なプロバイオティクス細菌であるkoumissから単離されたLactobacillus fermentum SM-7の特性評価。 J.Sci. 食べ物!. アグリック 91, 512–518. ドイ:10.1002/jsfa.4214
PubMed要約/CrossRef全文/Google Scholar
パパディミトリオウK. ることができます。,Anastasiou,R.,Maistrou,E.,Plakas,T.,Papandreou,N.C.,Hamodrakas,S.J.,et al. (2015). Streptococcus macedonicus ACA-DC198によるプラスミドpsma198の水平遺伝子導入による獲得は、種の乳製品起源に向かってポイントします。 PLoS ONE10:e0116337. doi:10.1371/ジャーナル。ポネ0116337
PubMed要約/CrossRef全文/Google Scholar
とができるようになりました。 ユニークなクレード特異的マーカー遺伝子を使用してメタゲノム微生物コミュニティプロファイリング。 ナット 9,811-814. ドイ:10.1038/nmeth.2066
Pubmed要約/CrossRef全文/Google Scholar
Sun,Z.,Liu,W.,Zhang,J.,Yu,J.,Zhang,W.,Cai,c.,et al. (2010). 中国のkoumissから単離された支配的な乳酸菌の同定と特性評価。 J.Gen.Appl. ミクロビオール 56, 257–265. 土井:10.2323/jgam.56.257
PubMed要約/CrossRef全文/Google Scholar
トンプソン,J.(1879). 無駄になる病気のkoumissの価値。 Br. メッド 1,270円 ドイ:10.1136/bmj.1.1466.270-c
PubMed要約/CrossRef全文/Google Scholar
とができるようにすることを目的としています。 Streptococcus macedonicus Sc136によって産生される外多糖類の構造および特性。 グリコバイオロジー11,131-139. doi:10.1093/glycob/11.2.131
PubMed要約/CrossRef全文/Google Scholar
Wang,L.,Christophersen,C.T.,Sorich,M.J.,Gerber,J.P.,Angley,M.T.,And Conlon,M.A.(2013). Sutterella sppの豊富さの増加。 そして、自閉症スペクトラム障害の子供の糞便中の反芻動物のトルク。 モル 4.42 土井: 10.1186/2040-2392-4-42
PubMed Abstract|CrossRef全文|Google Scholar
とができるようになりました。 ヒトミルクメタゲノーム:機能的能力分析。 BMCミクロビオルスモーカー。 13:116. 土井: 10.1186/1471-2180-13-116
PubMed Abstract|CrossRef全文|Google Scholar
渡辺K.,藤本J.,富井Y.,笹本M.,牧野H.,工藤Y.,et al. (2009). Lactobacillus kisonensis sp. 【送料無料】【,Lactobacillus otakiensis sp. 【送料無料】【、ラクトバチルス-ラピsp. 【送料無料】【 およびLactobacillus sunkii sp. 【送料無料】【、日本の伝統的なピクルスであるsunkiから単離された異発酵種。 Int. J.シスト エヴォル ミクロビオール 59(Pt4),754-760. ドイ:10.1099/ijs.0.004689-0
Pubmed要約/CrossRef全文/Google Scholar
Wu,R.,Wang,L.,Wang,J.,Li,H.,Menghe,B.,Wu,J.,et al. (2009). 内モンゴルのkoumissからの乳酸菌の分離そして予備のprobiotic選択。 J.Basic Microbiol。 49, 318–326. ドイ:10.1002/jobm.200800047
PubMed要約/CrossRef全文/Google Scholar
Zerbino,D.R.,およびBirney,E.(2008). Velvet:de Bruijnグラフを使用したde novo short read assemblyのアルゴリズム。 ゲノムRes.18,821-829. 土井:10.1101/gr.074492.107
PubMed要約/CrossRef全文/Google Scholar
Zhang,W.,Sun,Z.,Sun,T.,およびZhang,H.(2010a). Koumissから単離されたLactobacillus casei株におけるIOLクラスターのPCRスクリーニングと配列解析。 フォリア-ミクロビオール (プラハ)55、603-606。 土井:10.1007/12223-010-0097-3
PubMed Abstract|CrossRef全文|Google Scholar
Zhang,W.,Yu,D.,Sun,Z.,Wu,R.,Chen,X.,Chen,W.,et al. (2010b)。 中国内モンゴル自治区の伝統的な自家製koumissから単離された新しいプロバイオティクス株であるLactobacillus casei Zhangの完全なゲノム配列。 J.Bacteriol. 192, 5268–5269. 土井:10.1128/JB.00802-10
PubMed要約/CrossRef全文/Google Scholar
張、W.、および張、H.(2011)。 “発酵koumiss、”ハンドブック食品-飲料の発酵技術、2Edn ed. Y.H.Hui(Boca Raton,FL:CRC Press),165-172.
ることができると考えられている。 牛乳発酵中のLactobacillus helveticus H9のタンパク質分解系の遺伝子発現。 アン ミクロビオール 65, 1171–1175. ドイ:10.3168/jds.2014-8520
PubMed要約/CrossRef全文/Google Scholar
Zhao,W.,Chen,Y.,Sun,Z.,Wang,J.,Zhou,Z.,Sun,T.,et a l. (2011). Lactobacillus helveticus H10の完全なゲノム配列。 J.Bacteriol. 193, 2666–2667. 土井:10.1128/JB.00166-11
PubMed Abstract/CrossRef Full Text|Google Scholar