- Introduksjon
- Materialer Og Metoder
- Fremstilling Av Prøver
- Gradientfortynning Og Multiple Fortrengningsforsterkning
- Bibliotek Konstruksjon Og Sekvensering
- Dataanalyse
- Sekvenskvalitetskontroll Og Filtrering
- Taksonomisk Tildeling Og Mangfold
- Les Assembly, Gene Prediction, And Annotation
- Nukleotidsekvenstall
- Resultater
- Eksperimentell Design Og Sekvensering
- Taksonomisk Merknad
- Metagenomisk Montering, Genprediksjon Og Funksjonell Merknad
- Diskusjon
- Konklusjon
- Forfatterbidrag
- Interessekonflikt
- Anerkjennelse
- Supplerende Materiale
Introduksjon
Koumiss, også kalt chige, chigo, arrag, eller airag, i mongolsk språk, er en type tradisjonelle gjæret meieri produkt. Det har vært en populær mat i Mongolia Og Indre Mongolia I Kina i århundrer (Zhang Og Zhang, 2011). Folk i disse regionene pleide å konsumere koumiss under store festligheter og offergaver (Zhang Og Zhang, 2011). De tidligste nedtegnelsene av koumiss-produksjonen kan spores tilbake til Han-Dynastiet (BC202-AD202). Dette produktet hadde oppnådd utbredt popularitet under Yuan-Dynastiet (AD1271-AD1368) (Zhang et al.( 2010b). I Dag er koumiss en vanlig mat for Lokalbefolkningen I Mongolia og Indre Mongolia, men bare i få av disse områdene produseres den i industriell skala. Koumiss gir ikke bare rike næringsstoffer, inkludert høyt innhold av essensielle aminosyrer og vitaminer, men antas også å lindre et bredt spekter av medisinske forhold og er gunstig for postoperativ omsorg (Jagielski, 1877; Thompson, 1879).
tradisjonelt er koumiss vanligvis produsert i trefat, beholdere laget av dyrehud eller urner. Fermentering skjer naturlig ved omgivelsestemperatur etter tilsetning av filtrert hoppemelk i beholderen med gammel koumiss, som tjener som startkultur (Zhang Og Zhang, 2011). Koumiss er en god kilde til nye bakterier av bioteknologisk potensial (Zhang et al., 2010a; P. Et al., 2011). Derfor er det av stor interesse å utforske og bevare så mange fermenteringsassosierte koumiss-bakterier som mulig. I løpet av de siste tiårene ble det utført en rekke studier for å undersøke koumiss bakterielle samfunn (Wu Et al., 2009; Hao et al., 2010), hovedsakelig studert av kultur -, molekylærbiologi-og pyrosequencing-baserte metoder (Sun et al., 2010). Blant disse forskjellige tilnærmingene har den pyrosequencing-baserte metoden gitt den mest omfattende mikrobiotaprofilen til koumiss uavhengig av fenotypiske egenskaper og problemer med dyrkbarhet av de enkelte mikrober. Spekteret av funksjonelle gener kodet av koumiss-bakteriene og deres fermentative kapasiteter forblir imidlertid dårlig karakterisert, spesielt for de sjeldne mikrobielle populasjonene.
denne studien brukte single cell genomics teknikk for å analysere bakterielle metagenomer av 10 koumiss prøver samlet fra Mongolia og Indre Mongolia I Kina. Det nåværende arbeidet har brukt state-of-art teknologier for å undersøke bakteriell mangfold i meieriprodukter. Vårt arbeid har vist muligheten for å oppdage lav-rikelig taxa ved å bruke single cell metagenomics tilnærming. De oppmuntrende resultatene vil fremme utvikling og anvendelse av nye tilnærminger i å takle problemer i et tradisjonelt forskningsfelt.
Materialer Og Metoder
Fremstilling Av Prøver
Totalt 10 koumiss-prøver ble samlet inn fra Mongolia (Mg14, MG15, MG16, MG17 og MG18) Og Indre Mongolia I Kina (NM17, NM18, NM19, NM20 og NM21) for metagenomikkstudien. Prøver ble samlet aseptisk og ble transportert i tøris.
En milliliter av hver prøve ble forbehandlet i henhold til metodikken beskrevet I Ward et al. (2013) med noen endringer. Kort sagt ble prøvene tint i et isbad i 3-5 min. Etter at prøvene smeltet, ble de utsatt for sentrifugering med lav hastighet for å fjerne urenheter og eukaryotiske celleklumper. Prokaryote celler ble deretter pelletert fra melkeseraen ved sentrifugering ved 13 000 × g i 15 minutter. Pelletsene ble re-suspendert i 2 mL fosfatbufret saltvann (PBS) med 1% Triton X-100 og inkubert i 2 timer ved 37°C for å lyse opp eventuelle gjenværende eukaryote celler. Deretter ble bakterier pelletert ved sentrifugering ved 13 000 × g i 15 min og pelletsene ble suspendert på nytt i 500 µ PBS. Til slutt ble sentrifugeringstrinnet gjentatt en gang til for å vaske bakteriecellene.
Gradientfortynning Og Multiple Fortrengningsforsterkning
for å oppdage bakterier med lite innhold ble bakteriesuspensjonen avledet fra hver koumiss-prøve fortynnet serielt for påfølgende forsterkningsreaksjon. Cellenummeret i hver prøve ble grovt estimert under et mikroskop (Nikon, Tokyo, Japan) ved hjelp av et celletellekammer (Qiujing, Shanghai, Kina). Fortynningstrinnet ble videreført til celletallet i hver bakteriesuspensjon nådde omtrent 100. Multiple fortrengningsforsterkning av de fortynnede cellene ble utført ved BRUK AV REPLIKA-g Enkeltcellesett (Qiagen, Germantown, MD, USA) i henhold til produsentens instruksjoner.
Bibliotek Konstruksjon Og Sekvensering
Forsterket DNA ble skåret tilfeldig, og fragmenter av ca 500 bp ble valgt. Etter bibliotekets konstruksjon ble PerkinElmer LabChip® GX Touch Og StepOnePlusTM Sanntids PCR-System brukt til bibliotekets kvalitetskontroll. Til slutt ble 125-bp-parret lesing sekvensert På Illumina HiSeq 2500-plattformen i henhold til produsentens instruksjoner.
Dataanalyse
Sekvenskvalitetskontroll Og Filtrering
Raw-leser generert av sequenceren kan inneholde kunstig lesing av adapterkontaminering under bibliotekkonstruksjonen. Derfor ble det utført tre trinn for å oppnå et høyverdig rent lesedatasett: (1) eliminering av leser forårsaket av adapterkontaminering; (2) fjerning av leser med en gjennomsnittlig score under en phred score På Q30, som ble ansett som den laveste cutoff for en høy kvalitet base; (3) fjerning av leser med et betydelig overskudd av » N » (≥5% av lese). Nedstrømsanalysen var basert på de rene dataene. Videre ble den statistiske basiskvaliteten, basert På Q30 og gc-innholdet, beregnet.
Justeringer mot vertsgenomet ble utført for å fjerne de vertsstammede forurensningssekvensene. Enhver vertsstammet lesing ble kassert før videre sammenligning med bakterier (eller virus) genom referansesekvenser. For å oppnå mer nøyaktige resultater ble Burrows–Wheeler aligner (Bwa) (Versjon 0.97 a) MEM-modellen brukt i justeringene (Li Og Durbin, 2009).
Taksonomisk Tildeling Og Mangfold
webprogramvaren Metaphlan ble brukt til taksonomisk tildeling til slekts-og artsnivåer (Segata et al., 2012). For å sammenligne mangfoldet av arter innenfor og mellom prøver, analyserte vi alfa – og beta-mangfoldet av Den R-relaterte pakken.
Les Assembly, Gene Prediction, And Annotation
for å få mer omfattende informasjon, vi samlet de skåret fragmenter i genomet (contigs). På grunn av tilstedeværelsen av flere arter, som er vanlig i metagenomiske prøver, forbedret vi bioinformatikkgenommonteringsmetoden som normalt brukes til enkeltartanalyse ved å integrere Spader (Versjon 3.6.2), interne skript og metagenomiske databaser (Zerbino og Birney, 2008; Nurk et al., 2013).
Den MetaGeneMark programvare ble brukt for genet prediksjon av de sammensatte contigs (Noguchi et al., 2006). Overflødige gener ble fjernet VED HJELP AV CD-HIT med dekning av 90 og 95% identitet (Li Og Godzik, 2006; Fu et al., 2012). Relative forekomster av gener ble bestemt ved å justere sekvensering av høy kvalitet til genkatalogen ved hjelp av samme prosedyre. Nedstrøms uoverensstemmelsesanalysen var basert på genets relative overflod. Genannotasjon ble utført ved å tilpasse sekvensene av høy kvalitet mot flere offentlige databaser (nemlig NCBI ikke-redundant database, NR; Klynger Av Orthologe Grupper av proteiner, Tannhjul; Kyoto Encyclopedia Av Gener Og Genomer, KEGG) ved HJELP AV BLAST (Altschul et al., 1997). Et domenesøk ble utført Ved Hjelp Av Interproscan (Mulder Og Apweiler, 2008).
Nukleotidsekvenstall
sekvensdataene rapportert i denne studien er deponert I Sra-databasen (Tiltredelse Nr.SRP083102).
Resultater
Eksperimentell Design Og Sekvensering
for å oppdage lav-rikelig bakterier i koumiss ble enkeltcelleforsterkningsteknikken brukt til å analysere metagenomene av prøvene. 100 celler, ble avledet fra en uavhengig koumiss-prøve ved seriell fortynning. Totalt 30 fortynnede suspensjoner ble analysert. Hver fortynnet prøve ble gitt en annen prøvekode, dvs. prøveidentitetsnummeret etterfulgt av 1, 2 eller 3, som representerer de tre separate fortynningene. Basert på en premiss om at noen sjeldne arter vil være tilstede i en av fortynningene, ble det utført flere forskyvningsforsterkning av cellene; og rundt 5 Gb data ble generert for hver koumiss bakteriell suspensjon.
Totalt 1.040.323.864 rålesninger ble generert fra 10 koumiss-prøvene (totalt 30 bakterielle suspensjoner). Gjennomsnittlig antall lesinger for hver 100-cellersuspensjon var 34 677 462 (Supplerende Tabell S1). Etter trimming og filtrering av de ukvalifiserte sekvensene, oppnådde vi 1,018,381,702 rene leser for alle prøver (Supplerende Tabell S1). Verdiene Av Shannon index, Simpson index, Chao1 index og antall observerte arter (Figur 1-4) viste at de fleste koumiss-prøver hadde høy bakteriell biologisk mangfold. Shannon-Wiener-mangfoldskurvene viste at sekvensdybden var tilstrekkelig for alle prøver (Figur 1).
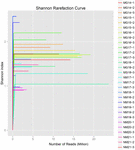
FIGUR 1. Shannon rarefaction kurver som anslår det mikrobielle mangfoldet av koumiss prøver.
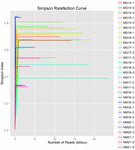
FIGUR 2. Simpson rarefaction kurver som anslår mikrobiell mangfold av koumiss prøver.
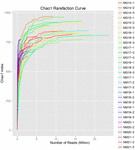
FIGUR 3. Chao1 rarefaction kurver som anslår det mikrobielle mangfoldet av koumiss prøver.
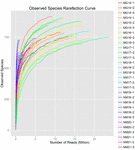
FIGUR 4. Observert arter sjeldnere indeks som anslår mikrobiell mangfold av koumiss prøver.
Taksonomisk Merknad
sekvensene av høy kvalitet ble tildelt forskjellige taksonomiske nivåer for å muliggjøre en grundig analyse av prøvebakteriesamfunnene. Med henvisning til noen publiserte studier på koumiss biologisk mangfold (Wu Et al., 2009; Hao et al., 2010; Sol et al., 2010), klassifiserte vi de kjente og tidligere ikke rapporterte koumiss-assosierte bakteriene som henholdsvis vanlige og sjeldne taxa.
sekvensene av høy kvalitet representerte 13 forskjellige slekter (Figur 5). Tre av dem hadde en gjennomsnittlig relativ overflod på over 1%, inkludert Lactobacillus (L.), Lactococcus og Streptococcus. Spesielt, Lactobacillus og Lactococcus var de to mest tallrike slekter funnet i koumiss. Andelen Laktobacillus i prøvene varierte fra 52,72 til 99,96%. To medlemmer av denne slekten, L. helveticus og l. kefiranofaciens, ble dominert blant de fleste koumiss-prøver (Figur 6). Arten L. buchneri var tilstede hovedsakelig i de mongolske prøvene, Mens Lactococcus lactis ble påvist i de fleste prøver uavhengig av prøvetakingsområdet.
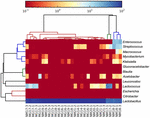
FIGUR 5. Heatmap viser den relative overflod av bakterier oppdaget i koumiss prøvene på slekten nivå.
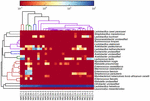
FIGUR 6. Heatmap viser den relative overflod av bakterier oppdaget i koumiss prøvene på artsnivå.
Metagenomisk Montering, Genprediksjon Og Funksjonell Merknad
Montering av lesingene resulterte i en samlingslengde på 614 392 623 bp. N50-verdiene for enhetene varierte fra 5,596 til 35,200 bp (Supplerende Tabell S2). Antall forventede gener i koumiss-prøvene varierte fra 10.347 til 34.547, med en gjennomsnittlig lengde fra 647.82 til 985.33 bp (Supplerende Tabell S3). Selv om empiriske genfunksjonelle analyser var utenfor omfanget av den nåværende studien, annoterte vi koumiss bakterielle mikrobiomer ved hjelp AV COG-og KEGG-databasene, som forutslo genfunksjonen i stor grad basert på sekvenshomologi.
totalt 545 kamper i annotasjonsutgangen viste høy homologi til laktoseutnyttelsesgenene (Tannhjulskategori karbohydrat Og metabolisme, G), og noen av dem var plassert innenfor samme contig (Tabell 1).
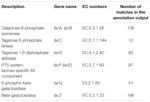
TABELL 1. Laktose metabolisme-relaterte gener annotert i koumiss bakteriell metagenome.
Noen andre sekvenser kan kode for antatte gener innenfor Tannhjulskategorien aminosyretransport og metabolisme (E), inkludert sekvenser som korresponderte med kasein-nedverdigende proteinaser, opp-systemet for opptak av oligopeptider av 4-18 rester og aminopeptidaser (f.eks. leucylaminopeptidase, peptidyl-dipeptidase A, aminopeptidase N, prolin iminopeptidase og endopeptidase). Selv om noen sekvenser delte høye likheter med aminotransferasene som var spesifikke for arginin, aspartat, metionin og isoleucin, ble bare en signifikant match identifisert, noe som tilsvarte et antatt s. macedonicus-opprinnelse klasse i/KLASSE II-domene (IPR004839) – inneholdende aminotransferase spesifikk for tyrosin og fenylalanin. Til slutt delte en rekke sekvenser høy homologi med aminosyrelyasene, inkludert S-ribosylhomocysteinlyase, argininosuccinatlyase, aspartat ammoniakk-lyase, cystathionin gamma-lyase, histidin ammoniakk-lyase og o-acetylhomoserin (tiol) – lyase.
Diskusjon
Koumiss er et populært tradisjonelt gjæret meieriprodukt i Mongolia og Indre Mongolia I Kina. Selv om en rekke studier har blitt utført for å undersøke bakteriell mangfold i koumiss, har det blitt oppnådd lite informasjon om den genetiske kapasiteten til koumiss mikrobiota. Her brukte vi enkeltcelleforsterkningsteknikken for å analysere koumiss bakterielle metagenomer, spesielt med fokus på den lave bakteriepopulasjonen.
den typiske metagenomikkmetoden har tidligere blitt brukt for å beskrive koumiss mikrobiota. På grunn av de høye kostnadene ved å oppnå en dyp sekvens, dekkes imidlertid den sjeldne mikrobiotapopulasjonen i prøvene ofte utilstrekkelig. Dermed forblir både fylogene og funksjonelle metagenomer av minoritetsbakteriene begrenset. Vår enkeltcelleforsterkningsmetode involverte seriell fortynning av prøver til 100-cellersuspensjoner. På grunn av det lave antallet celler som er tilstede i de fortynnede koumiss-prøvene, vil bare en begrenset MENGDE DNA bli ekstrahert. Amplifikasjonstrinnet økte mengden DNA-materialer som skulle analyseres og dermed lette metagenomanalysen av prøver som inneholdt SMÅ DNA-mengder. En ulempe med denne metoden var vanskeligheten med å sikre at sekvenser av hvert taxon ville bli like forsterket i prosessen; derfor kan resultatene som er oppnådd her bare gjenspeile de relative overflodene av sekvenser, men ikke absolutte mengder identifiserte taxa eller funksjonelle gener. Likevel vil dette ha liten effekt på vår analyse, da den nåværende studien skiller seg fra andre publiserte verk i å fokusere den sjeldne bakteriepopulasjonen. Data generert av dette arbeidet gir utfyllende informasjon til den underrepresenterte befolkningen. Vi mener den nåværende tilnærmingen er egnet for fremtidig analyse av bakteriemangfold for ulike typer økologiske miljøer.
koumiss bakterieflora består hovedsakelig av melkesyrebakterier (LAB) og noen eddiksyrebakterier (Zhang Og Zhang, 2011). Som forventet inneholdt datasettet for det meste sekvenser som representerer LABORATORIET og eddiksyrebakteriene. Sekvenser som tilsvarer Lactobacillus helveticus dominerte på tvers av alle prøver. Dessuten er sekvenser som representerer arten, l. kefiranofaciens. L. buchneri. L. kefiranofaciens. Enterococcus (e.) casseliflavus. E. faecalis. E. faecium. Leuconostoc mesenteroides. Lactococcus lactis og Acetobacter pasteurianus ble også funnet. Identifikasjonen av sekvenser av forskjellige taxa kan ikke være nok til å vise bakteriens levedyktighet, det reflekterer likevel bakteriesamfunnet på et tidspunkt i gjæringsprosessen. Miyamoto et al. (2010) antyder at bakteriell prevalens i de endelige fermenterte produktene er relatert til deres syrestresstoleranse. Generelt har laktobaciller en høyere syretoleranse enn laktokokker, noe som kan forklare den høye relative overflod av laktobacilli-sekvenser som er tilstede i datasettet vårt. På den annen side er den hyppige forekomsten Av L. helveticus i koumiss sammenfalt med observasjon av dominans Av l. helveticus sekvenser.
i vårt datasett korresponderte noen sekvenser Til L. otakiensis, som er en sjelden art som aldri har blitt rapportert i koumiss eller andre meieri nisjer. Arten ble først beskrevet og isolert fra den ikke-saltede pickling-løsningen som ble brukt til å produsere sunki, en tradisjonell Japansk pickle (Watanabe et al., 2009). Det ble oppdaget ved forsterket fragment lengde polymorfisme profilering basert på reca genet. Siden da har denne arten ikke blitt rapportert å være forbundet med andre matrelaterte nisjer. Således er det sannsynlig at denne bakterien tilhører den autochtone flora av pickles. Vi kan imidlertid ikke utelukke muligheten for at den ikke ble funnet bare på grunn av sensitiviteten til deteksjonsmetoden som ble brukt. Lactobacillus otakiensis kan produsere d-forgrenede aminosyrer, som d-leucin, d-allo-isoleucin og D-valin (Doi et al., 2013). Det har potensial til å bli brukt til å forbedre produksjonsegenskapene til visse fermenterte matvarer (Kato et al., 2011).
vår studie identifiserte også sekvenser som representerer arten s. macedonicus, som aldri har blitt rapportert som en koumiss-assosiert bakterie. I stedet er det en startkultur til stede i greske sauer og geitoster (Georgalaki et al., 2000). Noen medlemmer av denne arten er i stand til å produsere eksopolysakkarider, bakteriociner(Vincent et al., 2001; Anna Et al., 2015), og gamma-aminosmørsyre (Franciosi et al., 2015). Selv om denne arten ofte er isolert fra fermenterte matvarer, har Den opprinnelige nisjen Til s. macedonicus vært kontroversiell (Guarcello et al., 2016). Ikke før nylig, Papadimitriou et al. (2015) identifisert en ervervet plasmid, pSMA198, Fra Lactococcus lactis. Plasmidet ble sannsynligvis overført via en forfedre genetisk utvekslingshendelse innenfor et meieriproduktmiljø, noe som antydet melkenes opprinnelse Til s. macedonicus (Papadimitriou et al., 2015). Ligner På s. thermophilus. S. macedonicus er nært knyttet til commensals og opportunistiske patogener Av s. bovis/s. equinus-komplekset.
identifikasjonen av sekvenser som representerer arten Ruminococcus moments var uventet, da denne bakterien vanligvis finnes i tarmmiljøet. Det er en normal menneskelig tarmmikrobe som kan nedbryte mucinoligosakkarider ved konstitutiv produksjon av sekretoriske glykosidaser (Hoskins et al., 1985). Nylige kliniske bevis viser at fecal overflod av denne arten er endret hos barn med autismespektrumforstyrrelse; imidlertid er dens rolle i uorden uklart (Wang et al., 2013).
Overraskende representerte noen av sekvensene potensielle patogener. Klebsiella lungebetennelse er for eksempel et opportunistisk humant patogen som ligger i rundt 40% av mennesker og dyr. Mycobacterium orygis, tidligere kalt oryx bacillus, er medlem Av Mycobacterium tuberculosis complex som kan forårsake menneskelig tuberkulose (Dawson et al., 2012). Disse to artene har blitt rapportert i rå melk, men ikke koumiss. Derfor kan deres tilstedeværelse tilskrives forurensningen under konvensjonell koumissproduksjon, spesielt under ikke – eller lav-aseptiske manipulasjonsforhold.
Bakteriell metabolisme spiller en viktig rolle i å danne koumiss egenskaper og kvalitet. Mikrobielle-baserte prosesser som lipolyse og proteolyse er nødvendig for å syntetisere koumiss aromatiske og smaksforbindelser (Gesudu et al., 2016). Konsekvent inneholdt det bakterielle metagenomet sekvenser som potensielt koder for laktosedegradering og proteolytiske systemer. I motsetning til de relativt enkle laktosekatabolske veiene består LABORATORIEPROTEOLYTISKE systemene av et mangfoldig utvalg av enzymer (Chen et al., 2014). Sammenlignet med andre detekterte koumiss LAB, er den dominerende arten l. helveticus preget av høy proteolytisk aktivitet (Zhang et al., 2015). De fleste LAB har bare en celle-konvolutt proteinase som initierer melk kasein hydrolyse, Mens l. helveticus inneholder minst To av disse enzymene, Nemlig PrtH Og PrtH2 (Zhao et al ., 2011). Dermed kan de høye andelene av sekvenser som svarer til de sterke proteolytiske artene Av l. helveticus og proteolyserelaterte gener koble til det relativt høye innholdet av peptider og frie aminosyrer i koumiss.
en vanskelighet i industriell skala koumiss produksjon er kontroll av smak persepsjon, som koumiss har vært tradisjonelt laget av naturlig gjæring. Dermed har det vært vanskelig å definere smaken og profilen til viktige smakskomponenter, spesielt i nærvær av de naturlige forurensningene. Produksjonen av viktige smakskomponenter er et resultat av fermentativ og enzymatisk nedbrytning av aminosyrer, som forgrenede aminosyrer, metionin og aromatiske aminosyrer (Ardo, 2006). Eksempler på smakskomponenter inkluderer aldehyder, organiske syrer og estere, som dannes av transaminase (At)-vei (Helinck et al., 2004). Denne banen initieres av en transaminase som katalyserer omdannelsen av en aminosyre til dens tilsvarende α-keto syre (Helinck et al., 2004). Noen sekvenser i vårt datasett korresponderte med antatte forgrenede og aromatiske aminosyreaminotransferaser. Spesielt fant vi en antatt aromatisk aminosyre aminotransferase I i contig av koumiss-assosierte arter, S. macedonicus. Siden vårt nåværende arbeid bare annoterte mikrobiomet i silico, er våre data ikke nok til å vise at disse identifiserte gensekvensene faktisk var funksjonelle for å produsere de nevnte koumiss smakforbindelsene. Tilstedeværelsen av disse genene tyder likevel på at de er noen mulige kandidater for slike fermentative aktiviteter; likevel vil ytterligere eksperimentelt arbeid være nødvendig for å belyse deres eksakte funksjonelle roller.
Videre fant vi sekvenser som tilsvarer andre aminosyrekonverteringsveier, inkludert aminosyrelyaser og treonin aldolase. Det tidligere enzymet katalyserer omdannelsen av metionin til metanetiol (Irmler et al., 2008), noe som resulterer i dannelse av dimetyldisulfid og dimetyltrisulfid (Fernandez et al., 2000), mens den sistnevnte katalyserer omdannelsen av treonin til glycin og acetaldehyd (Ott et al., 2000). På samme måte kan bare å finne disse sekvensene i koumiss-mikrobiomet ikke tjene som klart bevis i deres faktiske biologiske roller; fremtidig eksperimentell bekreftelse vil være nødvendig.
Konklusjon
denne studien brukte en modifisert metagenomikkmetode for å analysere bakteriemikrobiomet av koumiss-prøver samlet fra Mongolia og Indre Mongolia I Kina. Vi karakteriserte både fylogene og funksjonelle metagenomer av de sjeldne artene i koumiss; og vårt datasett gjenspeiler egenskaper som er av bioteknologisk interesse og potensial. Vår studie har for første gang vist muligheten for å inkorporere enkeltcelleforsterkningsteknikker for å oppdage koumiss bakteriell mikrobiota, samt mikrobielle forurensninger. Teknikkene som er utviklet her, kan brukes i fremtidige studier for å overvåke endringer i koumiss-mikrobiomet langs gjæringsprosessen, med fokus på minoritetsmikrobialpopulasjonen. Videre kan andre omics-teknikker, som transkriptomikk, metabolomikk, kobles til den nåværende metagenomikkanalysen for å bekrefte funksjonene og metabolske kapasiteten til koumiss-mikrobiomet.
Teknisk sett vil neste trinn i dette arbeidet være å optimalisere dagens metode. For eksempel, ved å øke prøvefortynningen før DNA-forsterkning, kan sjansen for å avdekke sjeldne og nye bakterier forbedres. På den annen side kan en alternativ sekvenseringsteknikk som kan generere lange leser, for Eksempel Pacific Biosciences single molecule, sanntids sekvenseringsteknologi, brukes til å forbedre genommonteringsprosessen.
Forfatterbidrag
WZ OG HZ utformet studien. WZ, GY, JY og L-YK skrev manuskriptet. QH, WH, WL, BM og TS utførte eksperimenter. WZ og QH analyserte data. Alle forfattere har gjennomgått manuskriptet.
Interessekonflikt
forfatterne erklærer at forskningen ble utført i fravær av kommersielle eller økonomiske forhold som kan tolkes som en potensiell interessekonflikt.
Anerkjennelse
denne forskningen ble støttet Av National Natural Science Foundation Of China (Grant No. 31571815).
Supplerende Materiale
Supplerende Materiale for denne artikkelen kan bli funnet online på: https://www.frontiersin.org/article/10.3389/fmicb.2017.00165/full#supplementary-material
A. V., A. V., A. V., A. V., A. V., A. V., A. V., A. V., A. V., A. V., A. V., A. V., A. V., A. V., et al. (1997). Gapped BLAST og PSI-BLAST: en ny generasjon av protein database søk programmer. Nukleinsyrer Res. 25, 3389-3402. doi: 10.1093 / nar | 25.17.3389
PubMed Abstrakt | CrossRef Fulltekst / Google Scholar
A. V., A. V., A. V., A. V., A. V., A. V., A. V., A. V., A. V., A. V., A. v., A. v., a. v., et al. (2015). Konstruerte stammer Av Streptococcus macedonicus mot en osmotisk stressresistent fenotype beholder sin evne til å produsere bakteriocin macedocin under hyperosmotiske forhold. J. Biotechnol. 212, 125–133. doi: 10.1016 / j.jbiotec.2015.08.018
PubMed Abstract | CrossRef Full Text | Google Scholar
Ardo, Y. (2006). Smaksdannelse ved aminosyre katabolisme. Bioteknologi. Adv. 24, 238-242. doi: 10.1016 / j.biotechadv.2005.11.005
PubMed Abstract | CrossRef Full Text | Google Scholar
Chen, Y. F., Zhao, W. J., Wu, R. N., Sun, Zh, Zhang, W. Y., Wang, J. C., et al. (2014). Proteomanalyse Av Lactobacillus helveticus H9 under vekst i skummet melk. J. Meieri Sci. 97, 7413–7425. doi: 10.3168 / jds.2014-8520
PubMed Abstrakt | CrossRef Fulltekst | Google Scholar
Dawson, K. L., Bell, A., Kawakami, R. P., Coley, K., Yates, G. og Collins, D. M. (2012). Overføring Av Mycobacterium orygis (m. tuberculosis komplekse arter) fra en tuberkulose pasient til en melkeku I New Zealand. J. Clin. Mikrobiol. 50, 3136–3138. doi: 10.1128 / JCM.01652-12
PubMed Abstrakt | CrossRef Fulltekst | Google Scholar
Doi, K., Mori, K., Mutaguchi, Y., Tashiro, K., Fujino, Y., Ohmori, T., et al. (2013). Utkast til genomsekvens Av d-forgrenet aminosyreprodusent Lactobacillus otakiensis JCM 15040T, isolert fra en tradisjonell Japansk pickle. Genome Announc. 1, e546-e513. doi: 10.1128 / genomeA.00546-13
PubMed Abstrakt | CrossRef Fulltekst | Google Scholar
A., M., van Doesburg, W., Rutten, G. A., Marugg, J. D., Alting, A. C., van Kranenburg, R., et al. (2000). Molekylære og funksjonelle analyser av metc-genet Av Lactococcus lactis, som koder for cystathionin beta-lyase. Appl. Miljø. Mikrobiol. 66, 42–48. doi: 10.1128 / AEM.66.1.42-48.2000
PubMed Abstrakt | CrossRef Fulltekst | Google Scholar
I., Carafa, I., Nardin, T., Schiavon, S., Poznanski, E., Cavazza, A., Et al. (2015). Biologisk mangfold og gamma-aminosmørsyre produksjon av melkesyrebakterier isolert fra tradisjonelle alpine rå kumelk oster. Biomed Res. Int. 2015, 625740. doi: 10.1155/2015/625740
PubMed Abstrakt / Fulltekst / Google Scholar
Fu, L., Niu, B., Zhu, Z., Wu, S., Og Li, W. (2012). CD-HIT: akselerert for clustering neste generasjons sekvenseringsdata. Bioinformatics 28, 3150–3152. doi: 10.1093/bioinformatics/bts565
PubMed Abstract | CrossRef Full Text | Google Scholar
Georgalaki, M. D., Sarantinopoulos, P., Ferreira, E. S., De Vuyst, L., Kalantzopoulos, G., and Tsakalidou, E. (2000). Biochemical properties of Streptococcus macedonicus strains isolated from Greek Kasseri cheese. J. Appl. Microbiol. 88, 817–825. doi: 10.1046/j.1365-2672.2000.01055.x
PubMed Abstract | CrossRef Full Text | Google Scholar
Gesudu, Q. M., Zheng, Y., Xi, X. X., Hou, Q. C., Xie, H. Y., Huang, W. Q., et al. (2016). Undersøke bakteriell populasjon struktur og dynamikk i tradisjonell koumiss Fra Indre Mongolia ved hjelp av enkelt molekyl sanntids sekvensering. J. Meieri Sci. 99, 7852–7863. doi: 10.3168 / jds.2016-11167
PubMed Abstract | CrossRef Full Text | Google Scholar
G., G., G., G., G., G., G., G., G., G., G., G., G., G., G., G., G., G., g., et al. (2016). En stor fabrikkskala anvendelse av utvalgte autochtone melkesyrebakterier for PDO Pecorino Siciliano osteproduksjon. Mat Mikrobiol. 59, 66–75. doi: 10.1016 / j.fm. 2016.05.011
PubMed Abstrakt | CrossRef Fulltekst | Google Scholar
Hao, Y., Zhao, L., Zhang, H., Zhai, Z., Huang, Y., Liu, X., et al. (2010). Identifikasjon av bakteriell biologisk mangfold i koumiss ved denaturering gradient gel elektroforese og artsspesifikk polymerasekjedereaksjon. J. Meieri Sci. 93, 1926–1933. doi: 10.3168 / jds.2009-2822
PubMed Abstrakt | CrossRef Fulltekst | Google Scholar
Helinck, S., Le Bars, D., Moreau, D., Og Yvon, M. (2004). Evne til termofile melkesyrebakterier til å produsere aromaforbindelser fra aminosyrer. Appl. Miljø. Mikrobiol. 70, 3855–3861. doi: 10.1128 / AEM.70.7.3855-3861.2004
PubMed Abstrakt | CrossRef Fulltekst | Google Scholar
Hoskins, L. C., Agustines, M., McKee, W. B., Boulding, E. T., Kriaris, M. og Niedermeyer, G. (1985). Mucin nedbrytning i humane kolon økosystemer. Isolering og egenskaper av fekale stammer som nedbryter BLODGRUPPEANTIGENER og oligosakkarider fra mucin glykoproteiner. J. Clin. Investere. 75, 944–953. doi: 10.1172 / JCI111795
PubMed Abstrakt | CrossRef Fulltekst | Google Scholar
Irmler, S., Raboud, S., Beisert, B., Rauhut, D., Og Berthoud, H. (2008). Kloning og karakterisering av To lactobacillus casei-gener som koder for en cystathioninlyase. Appl. Miljø. Mikrobiol. 74, 99–106. doi: 10.1128 / AEM.00745-07
PubMed Abstrakt | CrossRef Fulltekst | Google Scholar
Jagielski, V. (1877). Verdien av koumiss i behandlingen av kvalme, oppkast og manglende evne til å beholde annen mat på magen. Br. Med. J. 2, 919-921. doi: 10.1136 / bmj.2.887.919
PubMed Abstrakt | CrossRef Fulltekst | Google Scholar
Kato, S., Ishihara, T., Hemmi, H., Kobayashi, H. ,Og Yoshimura, T. (2011). Endringer I D-aminosyrekonsentrasjoner og mikrobielle samfunnsstrukturer under gjæring av røde og hvite viner. J. Biosci. Bioeng. 111, 104–108. doi: 10.1016 / j.jbiosc.2010.08.019
PubMed Abstract | CrossRef Full Text | Google Scholar
Li, H., Og Durbin, R. (2009). Rask og nøyaktig kort lese justering Med Burrows-Wheeler transform. Bioinformatikk 25, 1754-1760. doi: 10.1093/bioinformatikk | btp324
PubMed Abstrakt | CrossRef Fulltekst / Google Scholar
Li, W., Og Godzik, A. (2006). Cd-hit: et raskt program for clustering og sammenligning av store sett med protein-eller nukleotidsekvenser. Bioinformatikk 22, 1658-1659. doi: 10.1093 / bioinformatikk | btl158
PubMed Abstrakt | CrossRef Fulltekst / Google Scholar
M., M., Seto, T., Nakajima, H., Burenjargal, S., Gombojav, A., Demberei, S., et al. (2010). Denaturering gradient gel elektroforese analyse av melkesyrebakterier og gjær i tradisjonell mongolsk gjæret melk. Mat Sci. Teknologi. Res. 16, 319-326. doi: 10.3136 / fstr.16.319
CrossRef Fulltekst | Google Scholar
Mulder, Nj, Og Apweiler, R. (2008). InterPro database og verktøy for protein domene analyse. Curr. Protokoll. Bioinformatikk Kapittel 2, Unit2. 7. doi: 10.1002 / 0471250953. bi0207s21
PubMed Abstrakt | Kryssref Full Tekst | Google Scholar
Noguchi, H., Park, J., Og Takagi, T. (2006). MetaGene: prokaryotisk genfunn fra miljøgenom haglgeværsekvenser. Nukleinsyrer Res. 34, 5623-5630. doi: 10.1093 / nar / gkl723
PubMed Abstrakt | CrossRef Fulltekst | Google Scholar
A. V., G. A., G. A., G. A., G. A., G. A., G. A., A., A., A., A., A., A., A., A., A., A., A., A., A., A., A., A., A., A., a., a., et al. (2013). Montering av enkeltcellegenomer og mini-metagenomer fra kimære mda-produkter. J. Comput. Biol. 20, 714–737. doi: 10.1089 / cmb.2013.0084
PubMed Abstrakt | CrossRef Fulltekst | Google Scholar
Ott, A., Germond, J. E., Og Chaintreau, A. (2000). Opprinnelse av acetaldehyd under melkgæring ved bruk av (13)C-merkede forløpere. J. Agric. Matkjem. 48, 1512–1517. doi: 10.1021 / jf9904867
PubMed Abstrakt | CrossRef Fulltekst | Google Scholar
Pan, Dd, Zeng, Xq, Og Yan, Yt (2011). Karakterisering Av Lactobacillus fermentum sm-7 isolert fra koumiss, en potensiell probiotisk bakterie med kolesterolsenkende effekter. J. Sci. Mat. Agric. 91, 512–518. doi: 10.1002 / jsfa.4214
PubMed Abstrakt | CrossRef Fulltekst | Google Scholar
Papadimitriou, K. I tillegg til dette har de også en rekke andre kilder, som for eksempel N. C., N. C., N. C., N. C., N. C., N. C., N. C., N. C., N. C., N. C., N. C., N. C., N. C., N. C., N. C., Et al. (2015). Oppkjøp gjennom horisontal genoverføring av plasmid pSMA198 Av Streptococcus macedonicus ACA-DC 198 peker mot artens meieri opprinnelse. PLoS EN 10: e0116337. doi: 10.1371 / tidsskrift.pone.0116337
PubMed Abstrakt | CrossRef Fulltekst | Google Scholar
Segata, N., Waldron, L., Ballarini, A., Narasimhan, V., Jousson, O. ,Og Huttenhower, C. (2012). Metagenomic mikrobielle samfunnet profilering ved hjelp av unike clade-spesifikke markør gener. Nat. Metoder 9, 811-814. doi: 10.1038 / nmeth.2066
PubMed Abstrakt | CrossRef Fulltekst | Google Scholar
J., Yu, J., Yu, J., Zhang, W., Cai, C., et al. (2010). Identifikasjon og karakterisering av de dominerende laktobacillene isolert fra koumiss I Kina. J. Gen. Appl. Mikrobiol. 56, 257–265. doi: 10.2323 / jgam.56.257
PubMed Abstrakt | CrossRef Fulltekst | Google Scholar
Thompson, J. (1879). Verdien av koumiss i å kaste bort sykdommer. Br. Med. J. 1, 270. doi: 10.1136 / bmj.1.1466.270-c
PubMed Abstrakt | CrossRef Fulltekst | Google Scholar
Vincent, S. J., Faber, E. J., Neeser, J. R., Stingele, F., Og Kamerling, J. P. (2001). Struktur og egenskaper av exopolysaccharide produsert Av Streptococcus macedonicus Sc136. Glykobiologi 11, 131-139. doi: 10.1093 / glykob / 11.2.131
PubMed Abstrakt | CrossRef Fulltekst | Google Scholar
Wang, L., Christophersen, C. T., Sorich, M. J., Gerber, J. P., Angley, M. T., Og Conlon, M. A. (2013). Økt overflod Av Sutterella spp. og Ruminococcus moments i avføring av barn med autisme spektrum lidelse. Mol. Autisme 4, 42. doi: 10.1186/2040-2392-4-42
PubMed Abstrakt / Fulltekst / Google Scholar
Ward, T. L., Hosid, S., Ioshikhes, I., Og Altosaar, I. (2013). Human milk metagenome: en funksjonskapasitetsanalyse. BMC Mikrobiol. 13:116. doi: 10.1186/1471-2180-13-116
PubMed Abstrakt / Fulltekst / Google Scholar
A., A., A., A., a., a., a., a., a., a., a., a., a., a., a., a., a., a., a., a., et al. (2009). Lactobacillus kisonensis sp. nov., Lactobacillus otakiensis sp. nov., Lactobacillus rapi sp. nov. Og Lactobacillus sunkii sp. nov., heterofermentative arter isolert fra sunki, en tradisjonell Japansk pickle. Int. J. Syst. Evol. Mikrobiol. 59 (Pt 4), 754-760. doi: 10.1099 / ijs.0.004689-0
PubMed Abstrakt | CrossRef Fulltekst | Google Scholar
Wu, R., Wang, L., Wang, J., Li, H., Menghe, B., Wu, J., et al. (2009). Isolering og foreløpig probiotisk utvalg av laktobaciller fra koumiss I Indre Mongolia. J. Grunnleggende Mikrobiol. 49, 318–326. doi: 10.1002 / jobm.200800047
PubMed Abstrakt | CrossRef Fulltekst | Google Scholar
Zerbino, D. R., Og Birney, E. (2008). Velvet: algoritmer for de novo kort lese montering ved Hjelp Av De Bruijn grafer. Genom Res. 18, 821-829. doi: 10.1101 / gr.074492.107
PubMed Abstrakt | CrossRef Fulltekst | Google Scholar
Zhang, W., Sun, Z., Sun, T., Og Zhang, H. (2010a). PCR-screening og sekvensanalyse av iol-klynger i Lactobacillus casei-stammer isolert fra koumiss. Folia Mikrobiol. (Praha) 55, 603-606. doi: 10.1007 / s12223-010-0097-3
PubMed Abstrakt / Fulltekst / Google Scholar
W., Yu, D., Sun, Z., Wu, R., Chen, X., Chen, W., et al. (2010b). Komplett genomsekvens Av Lactobacillus casei Zhang, en ny probiotisk stamme isolert fra tradisjonell hjemmelaget koumiss I Indre Mongolia, Kina. J. Bakteriol. 192, 5268–5269. doi: 10.1128 / JB.00802-10
PubMed Abstrakt | CrossRef Fulltekst | Google Scholar
Zhang, W., Og Zhang, H. (2011). «Fermentering og koumiss», I Håndbok For Fermenteringsteknologi For Mat Og Drikke, 2. Edn, red. Y. H. Hui (Boca Raton, FL: CRC Press), 165-172.
Google Scholar
Zhang, Wy, Chen, Yf, Zhao, Wj, Kwok, Ly, Og Zhang, Hp (2015). Genuttrykk av proteolytisk system Av Lactobacillus helveticus H9 under melkfermentering. Anne. Mikrobiol. 65, 1171–1175. doi: 10.3168 / jds.2014-8520
PubMed Abstrakt | CrossRef Fulltekst | Google Scholar
Zhao, W., Chen, Y., Sun, Z., Wang, J., Zhou, Z., Sun, T., et al. (2011). Komplett genomsekvens Av Lactobacillus helveticus H10. J. Bakteriol. 193, 2666–2667. doi: 10.1128 / JB.00166-11
PubMed Abstrakt | CrossRef Full Tekst / Google Scholar