- Introducere
- materiale și metode
- pregătirea probelor
- diluare în Gradient și amplificare cu deplasare multiplă
- construcția bibliotecii și secvențierea
- analiza datelor
- verificarea calității secvenței și filtrarea
- atribuire taxonomică și diversitate
- citiți asamblarea, Predicția genelor și adnotarea
- numere de aderare la secvența nucleotidelor
- rezultate
- proiectare experimentală și secvențiere
- adnotare taxonomică
- Adunarea Metagenomică, Predicția genei și adnotarea funcțională
- discuție
- concluzie
- contribuțiile autorului
- Declarație privind conflictul de interese
- recunoaștere
- material suplimentar
Introducere
Koumiss, numit și chige, chigo, arrag, sau airag, în limba mongolă, este un tip de produs lactat fermentat tradițional. A fost un aliment popular în Mongolia și Mongolia Interioară a Chinei de secole (Zhang și Zhang, 2011). Oamenii din aceste regiuni obișnuiau să consume koumiss în timpul marilor festivități și ofrande de sacrificiu (Zhang și Zhang, 2011). Cea mai veche înregistrare a producției koumiss poate fi urmărită înapoi la Dinastia Han (BC202-AD202). Acest produs a atins popularitate pe scară largă în timpul dinastiei Yuan (AD1271-AD1368) (Zhang și colab., 2010b). În zilele noastre, koumiss este un aliment comun pentru localnicii din Mongolia și Mongolia Interioară, deși numai în puține dintre aceste zone este fabricat la scară industrială. Koumiss nu numai că furnizează substanțe nutritive bogate, inclusiv conținut ridicat de aminoacizi esențiali și vitamine, dar se crede, de asemenea, că ameliorează o gamă largă de afecțiuni medicale și este benefic pentru îngrijirea postoperatorie (Jagielski, 1877; Thompson, 1879).
în mod tradițional, koumiss este produs în mod obișnuit în butoaie de lemn, recipiente din piele de animal sau urne. Fermentarea are loc în mod natural la temperatura ambiantă după adăugarea laptelui filtrat de iapă în recipientul cu koumiss vechi, care servește ca cultură de început (Zhang și Zhang, 2011). Koumiss este o sursă bună de bacterii noi de potențial biotehnologic (Zhang și colab., 2010a; Pan și colab., 2011). Prin urmare, este de interes intens să explorăm și să conservăm cât mai multe bacterii koumiss asociate fermentației. În ultimele decenii, au fost efectuate o serie de studii pentru a investiga comunitatea bacteriană koumiss (Wu și colab., 2009; Hao și colab., 2010), studiat în principal prin metode bazate pe cultură, Biologie Moleculară și pirosecvențe (Sun și colab., 2010). Printre aceste abordări diferite, metoda bazată pe pirosecvențe a oferit cel mai cuprinzător profil microbiot al koumiss independent de trăsăturile fenotipice și problemele de cultivabilitate ale microbilor individuali. Cu toate acestea, spectrul genelor funcționale codificate de bacteriile koumiss și capacitățile lor fermentative rămân slab caracterizate, în special pentru populațiile microbiene rare.
studiul de față a folosit tehnica genomică cu o singură celulă pentru a analiza metagenomii bacterieni a 10 probe de koumiss colectate din Mongolia și Mongolia Interioară din China. Lucrarea actuală a aplicat tehnologii de ultimă oră în investigarea diversității bacteriene din produsele lactate. Munca noastră a demonstrat fezabilitatea descoperirii taxonilor cu abundență redusă prin aplicarea abordării metagenomice cu o singură celulă. Rezultatele încurajatoare ar promova dezvoltarea și aplicarea unor abordări noi în abordarea problemelor într-un domeniu tradițional de cercetare.
materiale și metode
pregătirea probelor
un total de 10 probe koumiss au fost colectate din Mongolia (MG14, MG15, MG16, MG17 și MG18) și Mongolia Interioară din China (NM17, NM18, NM19, NM20 și NM21) pentru studiul metagenomică. Probele au fost colectate aseptic și au fost transportate în gheață uscată.
un mililitru din fiecare probă a fost pretratat conform metodologiei descrise în Ward și colab. (2013) cu unele modificări. Pe scurt, probele au fost dezghețate într-o baie de gheață timp de 3-5 minute. După ce probele s-au topit, au fost supuse centrifugării cu viteză mică pentru a îndepărta impuritățile și aglomerările de celule eucariote. Celulele procariote au fost apoi granulate din serurile de lapte prin centrifugare la 13.000 de grame pentru 15 min. Peletele au fost Re-suspendate în 2 mL soluție salină tamponată cu fosfat (PBS) cu 1% Triton X-100 și incubate timp de 2 ore la 37 centicc pentru a Liza orice celule eucariote rămase. Ulterior, bacteriile au fost peletate prin centrifugare la 13.000 de centi g timp de 15 min, iar peletele au fost Re-suspendate în 500 de centi PBS. În cele din urmă, etapa de centrifugare a fost repetată încă o dată pentru a spăla celulele bacteriene.
diluare în Gradient și amplificare cu deplasare multiplă
pentru a detecta bacteriile cu abundență redusă, suspensia bacteriană derivată din fiecare probă de koumiss a fost diluată în serie pentru reacția de amplificare ulterioară. Numărul de celule din fiecare probă a fost estimat aproximativ la microscop (Nikon, Tokyo, Japonia) folosind o cameră de numărare a celulelor (Qiujing, Shanghai, China). Etapa de diluare a fost continuată până când numărul de celule din fiecare suspensie bacteriană a ajuns la aproximativ 100. Amplificarea cu deplasare multiplă a celulelor diluate a fost efectuată utilizând kitul cu o singură celulă REPLI-g (Qiagen, Germantown, MD, SUA) conform instrucțiunilor producătorului.
construcția bibliotecii și secvențierea
ADN-ul amplificat a fost tăiat aleatoriu și au fost selectate fragmentele de aproximativ 500 bp. După construirea bibliotecii, PerkinElmer LabChip GX touch și sistemul PCR StepOnePlusTM în timp real au fost utilizate pentru inspecția calității bibliotecii. În cele din urmă, citirile 125-BP asociate au fost secvențiate pe platforma Illumina HiSeq 2500 conform instrucțiunilor producătorului.
analiza datelor
verificarea calității secvenței și filtrarea
citirile brute generate de secvențiator pot conține citiri artificiale ale contaminării adaptorului în timpul construcției bibliotecii. Prin urmare, au fost efectuate trei etape pentru a obține un set de date de citire curată de înaltă calitate: (1) eliminarea citirilor cauzate de contaminarea adaptorului; (2) eliminarea citirilor cu un scor mediu sub un scor phred de Q30, care a fost considerat cel mai mic cutoff pentru o bază de înaltă calitate; (3) eliminarea citirilor cu un exces semnificativ de „n” (5% din citire). Analiza din aval s-a bazat pe date curate. Mai mult, s-a calculat calitatea bazei statistice, bazată pe Q30 și conținutul GC.
au fost efectuate alinieri împotriva genomului gazdă pentru a elimina secvențele contaminante originare din gazdă. Orice citire originară din gazdă a fost aruncată înainte de o comparație suplimentară cu secvențele de referință ale genomului bacteriilor (sau virusurilor). Pentru a obține rezultate mai precise, Modelul mem Burrows–Wheeler aligner (BWA) (versiunea 0.97 a) a fost utilizat în alinieri (Li și Durbin, 2009).
atribuire taxonomică și diversitate
software-ul web Metaphlan a fost utilizat pentru atribuirea taxonomică la niveluri de gen și specii (Segata și colab., 2012). Pentru a compara diversitatea speciilor în cadrul și între probe, am analizat diversitatea alfa și beta prin pachetul legat de R.
citiți asamblarea, Predicția genelor și adnotarea
pentru a obține informații mai cuprinzătoare, am asamblat fragmentele tăiate în genom (contigs). Cu toate acestea, datorită prezenței mai multor specii, care este comună în probele metagenomice, am îmbunătățit metoda de asamblare a genomului bioinformatic utilizată în mod normal pentru analiza unei singure specii prin integrarea SPAdes (versiunea 3.6.2), scripturi interne și baze de date metagenomice (Zerbino și Birney, 2008; Nurk și colab., 2013).
software-ul MetaGeneMark a fost utilizat pentru predicția genelor contigilor asamblați (Noguchi și colab., 2006). Genele redundante au fost eliminate folosind CD-HIT cu acoperirea identității de 90 și 95% (Li și Godzik, 2006; Fu și colab., 2012). Abundențele relative ale genelor au fost determinate prin alinierea citirilor de secvențiere de înaltă calitate în catalogul genelor folosind aceeași procedură. Analiza discrepanței din aval s-a bazat pe abundențele relative ale genei. Adnotarea genelor a fost realizată prin alinierea secvențelor de înaltă calitate la mai multe baze de date publice (și anume baza de date non-redundantă NCBI, NR; clustere de grupuri Ortologice de proteine, rotițe; Enciclopedia Kyoto a genelor și genomilor, KEGG) folosind BLAST (Altschul și colab., 1997). O căutare de domeniu a fost efectuată utilizând Interproscan (Mulder și Apweiler, 2008).
numere de aderare la secvența nucleotidelor
datele de secvență raportate în acest studiu au fost depozitate în baza de date SRA (acces nr.SRP083102).
rezultate
proiectare experimentală și secvențiere
pentru a detecta bacteriile slab abundente din koumiss, tehnica de amplificare cu o singură celulă a fost utilizată pentru a analiza metagenomii probelor. Trei suspensii bacteriene, fiecare din aproximativ 100 de celule, au fost derivate dintr-o probă independentă de koumiss prin diluare în serie. Au fost analizate în total 30 de suspensii diluate. Fiecărui eșantion diluat i s-a dat un cod de eșantion diferit, adică numărul de identitate al eșantionului urmat de 1, 2 sau 3, reprezentând cele trei diluții separate. Pe baza premisei că unele specii rare vor fi prezente într-una dintre diluții, s-a efectuat amplificarea deplasării multiple a celulelor; și au fost generate date de aproximativ 5 Gb pentru fiecare suspensie bacteriană koumiss.
un total de 1.040.323.864 de citiri brute au fost generate din cele 10 probe de koumiss (un total de 30 de suspensii bacteriene). Numărul mediu de citiri pentru fiecare suspensie de 100 de celule a fost de 34.677.462 (tabelul suplimentar S1). După tunderea și filtrarea secvențelor necalificate, am obținut 1.018.381.702 citiri curate pentru toate probele (tabelul suplimentar S1). Valorile indicelui Shannon, indicele Simpson, indicele Chao1 și numărul speciilor observate (figurile 1-4) au arătat că majoritatea probelor de koumiss au o biodiversitate bacteriană ridicată. Curbele de diversitate Shannon-Wiener au arătat că adâncimea secvenței a fost adecvată pentru toate probele (Figura 1).
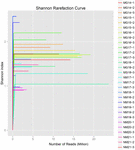
FIGURA 1. Curbele de rarefiere Shannon care estimează diversitatea microbiană a probelor koumiss.
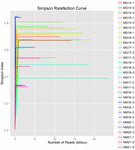
FIGURA 2. Curbele de rarefiere Simpson care estimează diversitatea microbiană a probelor koumiss.
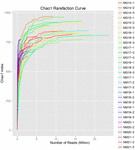
FIGURA 3. Curbe de rarefiere Chao1 care estimează diversitatea microbiană a probelor koumiss.
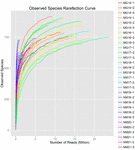
FIGURA 4. Indicele de rarefiere a speciilor observate care estimează diversitatea microbiană a probelor koumiss.
adnotare taxonomică
secvențele de înaltă calitate au fost atribuite diferitelor niveluri taxonomice pentru a permite o analiză aprofundată a comunităților bacteriene eșantion. Cu referire la unele studii publicate privind biodiversitatea koumiss (Wu și colab., 2009; Hao și colab., 2010; Sun și colab., 2010), am clasificat bacteriile asociate koumiss cunoscute și care nu au fost raportate anterior ca taxoni obișnuiți și, respectiv, rari.
secvențele de înaltă calitate au reprezentat 13 genuri diferite (Figura 5). Trei dintre ei au avut o abundență relativă medie de peste 1%, inclusiv Lactobacillus (L.), Lactococcus și Streptococcus. În special, lactobacilul și Lactococul au fost cele două genuri cele mai abundente găsite în koumiss. Proporția Lactobacilului în probe a variat de la 52,72 la 99,96%. Doi membri ai acestui gen, L. helveticus și L. kefiranofaciens, au fost dominați printre majoritatea probelor de koumiss (Figura 6). Specia L. buchneri a fost prezent mai ales în probele mongole, în timp ce Lactococcus lactis a fost detectat în majoritatea probelor, indiferent de regiunea de eșantionare.
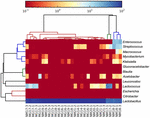
FIGURA 5. Harta de căldură care arată abundența relativă a bacteriilor detectate în probele koumiss la nivel de gen.
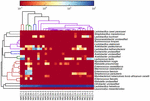
FIGURA 6. Harta de căldură care arată abundența relativă a bacteriilor detectate în probele de koumiss la nivel de specie.
Adunarea Metagenomică, Predicția genei și adnotarea funcțională
asamblarea citirilor a dus la o lungime de asamblare de 614.392.623 bp. Valorile N50 pentru ansambluri au variat de la 5.596 la 35.200 bp (tabelul suplimentar S2). Numărul de gene prezise în probele de koumiss a variat de la 10.347 la 34.547, cu o lungime medie cuprinsă între 647,82 și 985,33 bp (tabelul suplimentar S3). Deși analizele funcționale ale genelor empirice au depășit domeniul de aplicare al studiului actual, am adnotat microbiomii bacterieni koumiss folosind bazele de date COG și KEGG, care au prezis funcția genică bazată în mare parte pe omologia secvenței.
un total de 545 de potriviri în rezultatul adnotării au arătat o omologie ridicată la genele de utilizare a lactozei (categoria COGs de carbohidrați și metabolism, G), iar unele dintre ele au fost localizate în același contig (Tabelul 1).
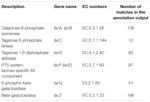
TABELUL 1. Genele legate de metabolismul lactozei adnotate în metagenomul bacterian koumiss.
alte secvențe ar putea codifica genele presupuse din categoria COGs a transportului și metabolismului aminoacizilor (E), inclusiv secvențe care corespundeau proteinazelor degradante de cazeină, sistemului Opp pentru preluarea oligopeptidelor de 4-18 reziduuri și aminopeptidazelor (de exemplu, leucil aminopeptidază, peptidil-dipeptidază A, aminopeptidază N, prolină iminopeptidază endopeptidază). Deși unele secvențe au avut asemănări ridicate cu aminotransferazele specifice argininei, aspartatului, metioninei și izoleucinei, a fost identificată o singură potrivire semnificativă, care corespundea unui presupus S. macedonicus-originea clasei I/Clasa II domeniu (IPR004839)-conținând aminotransferază specifică pentru tirozină și fenilalanină. În cele din urmă, o serie de secvențe au împărtășit o omologie ridicată cu aminoacizii liaze, inclusiv s-ribozilhomocisteină liază, argininosuccinat liază, aspartat amoniac-liază, cistationină gamma-liază, histidină amoniac-liază și o-acetilhomoserină (tiol)-liază.
discuție
Koumiss este un produs lactat tradițional fermentat popular în Mongolia și Mongolia Interioară a Chinei. Deși au fost efectuate o serie de studii pentru a investiga diversitatea bacteriană în koumiss, s-au obținut puține informații cu privire la capacitatea genetică a microbiotei koumiss. Aici, am aplicat tehnica de amplificare cu o singură celulă pentru a analiza metagenomii bacterieni koumiss, concentrându-ne în special pe populația bacteriană cu abundență redusă.
abordarea metagenomică tipică a fost aplicată anterior pentru a descrie microbiota koumiss. Cu toate acestea, datorită costului ridicat al realizării unei secvențe profunde, populația rară de microbiotă din probe este adesea acoperită inadecvat. Astfel, atât metagenomii filogeni, cât și cei funcționali ai bacteriilor minoritare rămân limitați. Metoda noastră de amplificare cu o singură celulă a implicat diluarea în serie a probelor la suspensii de 100 de celule. Datorită numărului redus de celule prezente în probele de koumiss diluate, ar fi extrasă doar o cantitate limitată de ADN. Etapa de amplificare a crescut cantitatea de materiale ADN care urmează să fie analizate și, prin urmare, a facilitat analiza metagenomului probelor care conțin cantități minime de ADN. Un dezavantaj al acestei metode a fost dificultatea de a se asigura că secvențele fiecărui taxon vor fi amplificate în mod egal în acest proces; prin urmare, rezultatele obținute aici ar putea reflecta doar abundența relativă a secvențelor, dar nu și cantitățile absolute de taxoni identificați sau gene funcționale. Cu toate acestea, acest lucru ar avea un efect redus asupra analizei noastre, deoarece studiul de față diferă de alte lucrări publicate în concentrarea populației bacteriene rare. Datele generate de această lucrare oferă informații complementare populației subreprezentate. Considerăm că abordarea actuală este potrivită pentru analiza viitoare a diversității bacteriilor pentru diferite tipuri de medii ecologice.
microbiota bacteriană koumiss este formată în principal din bacterii de acid lactic (LAB) și unele bacterii de acid acetic (Zhang și Zhang, 2011). Așa cum era de așteptat, setul nostru de date conținea în mare parte secvențe reprezentând bacteriile de laborator și acid acetic. Secvențele corespunzătoare Lactobacillus helveticus au dominat toate probele. În plus, secvențe reprezentând specia, L. kefiranofaciens. L. buchneri. L. kefiranofaciens. Enterococcus (E.) casseliflavus. E. faecalis. E. fecale. Leuconostoc mesenteroides. Lactococcus lactis și Acetobacter pasteurianus au fost, de asemenea, găsite. Identificarea secvențelor de taxoni diferiți poate să nu fie suficientă pentru a arăta viabilitatea bacteriilor, totuși reflectă comunitatea bacteriană la un moment dat al procesului de fermentare. Miyamoto și colab. (2010) sugerează că prevalența bacteriană în produsele fermentate finale este legată de toleranța lor la stres acid. În general, lactobacilii au o toleranță mai mare la acid decât lactococii, ceea ce poate explica abundența relativă ridicată a secvențelor de lactobacili prezente în setul nostru de date. Pe de altă parte, apariția frecventă a L. helveticus în koumiss a coincis cu observarea dominanței secvențelor L. helveticus.
în setul nostru de date, unele secvențe corespundeau L. otakiensis, care este o specie rară care nu a fost niciodată raportată în koumiss sau în alte nișe lactate. Specia a fost descrisă mai întâi și izolată de soluția de decapare nesărată utilizată la producerea sunki, o murătură tradițională japoneză (Watanabe și colab., 2009). A fost descoperit prin profilarea polimorfismului lungimii fragmentelor amplificate pe baza genei recA. De atunci, această specie nu a fost raportată a fi asociată cu alte nișe legate de alimente. Astfel, este probabil ca această bacterie să aparțină florei autohtone a murăturilor. Cu toate acestea, nu putem exclude posibilitatea ca aceasta să nu fi fost găsită pur și simplu datorită sensibilității metodei de detectare utilizate. Lactobacillus otakiensis poate produce aminoacizi cu catenă ramificată d, cum ar fi d-leucina, d-Alo-izoleucina și D-valina (Doi și colab., 2013). Are potențialul de a fi utilizat în îmbunătățirea caracteristicilor de producție ale anumitor alimente fermentate (Kato și colab., 2011).
studiul nostru a identificat, de asemenea, secvențe reprezentând specia S. macedonicus, care nu a fost niciodată raportată ca o bacterie asociată cu koumiss. În schimb, este o cultură inițială prezentă în brânzeturile grecești de oaie și capră (Georgalaki și colab., 2000). Unii membri ai acestei specii sunt capabili să producă exopolizaharide, bacteriocine (Vincent și colab., 2001; Anastasiou și colab., 2015) și acid gamma-aminobutiric (Franciosi și colab., 2015). Chiar dacă această specie este frecvent izolată de alimentele fermentate, nișa originală a lui S. macedonicus a fost controversată (Guarcello și colab., 2016). Nu până de curând, Papadimitriou și colab. (2015) a identificat o plasmidă dobândită, pSMA198, de la Lactococcus lactis. Plasmida a fost probabil transferată printr-un eveniment de schimb genetic ancestral într-un mediu de produse lactate, sugerând originea lactată a lui S. macedonicus (Papadimitriou și colab., 2015). Similar cu S. thermophilus. S. macedonicus este strâns legat de comensalii și agenții patogeni oportuniști ai complexului S. bovis/S. equinus.
identificarea secvențelor reprezentând cuplurile de Ruminococcus a fost neașteptată, deoarece această bacterie se găsește de obicei în mediul intestinal. Este un microb normal al intestinului uman care poate degrada oligozaharidele mucinice prin producerea constitutivă a glicozidazelor secretoare (Hoskins și colab., 1985). Dovezile clinice recente arată că abundența fecală a acestei specii este modificată la copiii cu tulburare de spectru autist; cu toate acestea, rolul său în tulburare rămâne neclar (Wang și colab., 2013).
în mod surprinzător, unele dintre secvențe au reprezentat potențiali agenți patogeni. De exemplu, pneumonia Klebsiella este un agent patogen oportunist uman care se află în aproximativ 40% din intestinele umane și animale. Mycobacterium orygis, numit anterior bacilul oryx, este un membru al complexului Mycobacterium tuberculosis care poate provoca tuberculoză umană (Dawson și colab., 2012). Aceste două specii au fost raportate în laptele crud, dar nu în koumiss. Prin urmare, prezența lor s – ar putea atribui contaminării în timpul producției convenționale de koumiss, în special în condiții de manipulare non-sau slab aseptice.
metabolismul bacterian joacă un rol important în formarea caracteristicilor și calității koumiss. Procesele bazate pe microbiene, cum ar fi lipoliza și proteoliza, sunt necesare pentru sintetizarea compușilor aromatici și aromatici koumiss (Gesudu și colab., 2016). În mod consecvent, metagenomul bacterian conținea secvențe care pot codifica degradarea lactozei și sistemele proteolitice. Spre deosebire de căile catabolice relativ simple ale lactozei, sistemele proteolitice de laborator sunt alcătuite dintr-o gamă diversă de enzime (Chen și colab., 2014). În comparație cu alte laboratoare koumiss detectate, specia dominantă L. helveticus este caracterizată cu o activitate proteolitică ridicată (Zhang și colab., 2015). Majoritatea laboratoarelor posedă o singură proteinază celulară care inițiază hidroliza cazeinei din lapte, în timp ce L. helveticus conține cel puțin două dintre aceste enzime, și anume PrtH și PrtH2 (Zhao și colab., 2011). Astfel, proporțiile ridicate de secvențe corespunzătoare speciilor proteolitice puternice ale L. helveticus și genelor legate de proteoliză se pot lega de conținutul relativ ridicat de peptide și aminoacizi liberi din koumiss.
o dificultate în producția de koumiss la scară industrială este controlul percepției aromelor, deoarece koumiss a fost realizat în mod tradițional prin fermentație naturală. Astfel, a fost dificil să se definească aroma și profilul componentelor cheie ale aromei, în special în prezența contaminanților naturali. Producerea componentelor cheie de aromă este rezultatul degradării fermentative și enzimatice a aminoacizilor, cum ar fi aminoacizii cu catenă ramificată, metionina și aminoacizii aromatici (Ardo, 2006). Exemple de componente de aromă includ aldehide, acizi organici și esteri, care sunt formați prin calea transaminazei (AT) (Helinck și colab., 2004). Această cale este inițiată de o transaminază care catalizează conversia unui aminoacid în acidul său corespunzător-ceto (Helinck și colab., 2004). Unele secvențe din setul nostru de date corespundeau aminoacizilor aminotransferazelor cu catenă ramificată și aromatică. În special, am găsit un aminoacid aromatic presupus aminotransferază I în contigul speciei asociate koumiss, S. macedonicus. Deoarece lucrarea noastră actuală a adnotat doar microbiomul in silico, datele noastre nu sunt suficiente pentru a arăta că aceste secvențe genetice identificate au fost într-adevăr funcționale pentru a produce compușii de aromă koumiss menționați mai sus. Prezența acestor gene sugerează totuși că sunt unii candidați posibili pentru astfel de activități fermentative; totuși, ar fi necesare lucrări experimentale suplimentare pentru a elucida rolurile lor funcționale exacte.
mai mult, am găsit secvențe corespunzătoare altor căi de conversie a aminoacizilor, inclusiv aminoacizii liaze și treonină aldolază. Fosta enzimă catalizează conversia metioninei în metanetiol (Irmler și colab., 2008), rezultând astfel formarea disulfurii de dimetil și a trisulfurii de dimetil (Fernandez și colab., 2000), în timp ce acesta din urmă catalizează conversia treoninei în glicină și acetaldehidă (Ott și colab., 2000). În mod similar, simpla localizare a acestor secvențe în microbiomul koumiss nu poate servi drept dovadă certă în rolurile lor biologice reale; va fi necesară o confirmare experimentală viitoare.
concluzie
studiul de față a utilizat o metodă metagenomică modificată pentru a analiza microbiomul bacterian al probelor koumiss colectate din Mongolia și Mongolia Interioară a Chinei. Am caracterizat atât metagenomii filogeni, cât și cei funcționali ai speciilor rare din koumiss; iar setul nostru de date Reflectă trăsături care prezintă interes și potențial biotehnologic. Studiul nostru a demonstrat pentru prima dată fezabilitatea încorporării tehnicilor de amplificare cu o singură celulă în detectarea microbiotei bacteriene koumiss, precum și a contaminanților microbieni. Tehnicile dezvoltate aici pot fi utilizate în studii viitoare pentru a monitoriza modificările microbiomului koumiss de-a lungul procesului de fermentare, cu accent pe populația microbiană minoritară. Mai mult, alte tehnici omics, cum ar fi transcriptomica, metabolomica, pot fi cuplate la analiza metagenomică actuală pentru a confirma funcțiile și capacitatea metabolică a microbiomului koumiss.
din punct de vedere tehnic, următorul pas al acestei lucrări ar fi optimizarea metodei actuale. De exemplu, prin creșterea diluției probei înainte de amplificarea ADN-ului, șansa de a descoperi bacterii rare și noi poate fi îmbunătățită. Pe de altă parte, o tehnică alternativă de secvențiere care poate genera citiri lungi, cum ar fi Pacific Biosciences singură moleculă, tehnologia de secvențiere în timp real, poate fi utilizată pentru a îmbunătăți procesul de asamblare a genomului.
contribuțiile autorului
WZ și HZ au proiectat studiul. WZ, GY, JY și L-YK au scris manuscrisul. QH, WH, WL, BM și TS au efectuat experimente. WZ și QH au analizat datele. Toți autorii au revizuit manuscrisul.
Declarație privind conflictul de interese
autorii declară că cercetarea a fost realizată în absența oricăror relații comerciale sau financiare care ar putea fi interpretate ca un potențial conflict de interese.
recunoaștere
această cercetare a fost susținută de Fundația Națională de științe Naturale din China (Grant Nr.31571815).
material suplimentar
materialul suplimentar pentru acest articol poate fi găsit online la: https://www.frontiersin.org/article/10.3389/fmicb.2017.00165/full#supplementary-material
Altschul, S. F., Madden, T. L., Schaffer, A. A., Zhang, J., Zhang, Z., Miller, W., și colab. (1997). Gapped BLAST și PSI-BLAST: o nouă generație de programe de căutare de baze de date de proteine. Acizi Nucleici Res. 25, 3389-3402. doi: 10.1093/nar / 25.17.3389
PubMed rezumat / CrossRef text integral / Google Scholar
Anastasiou, R., Driessche, G. V., Boutou, E., Kazou, M., Alexandraki, V., Vorgias, C. E. și colab. (2015). Tulpinile proiectate de Streptococcus macedonicus către un fenotip rezistent la stres osmotic își păstrează capacitatea de a produce bacteriocina macedocină în condiții hiperosmotice. J. Biotehnol. 212, 125–133. doi: 10.1016 / j. jbiotec.2015.08.018
PubMed Rezumat / CrossRef Text Integral / Google Scholar
Ardo, Y. (2006). Formarea aromelor prin catabolismul aminoacizilor. Biotehnol. ADV. 24, 238-242. doi: 10.1016 / j. biotechadv.2005.11.005
PubMed rezumat / CrossRef text integral / Google Scholar
de asemenea, este important să se țină cont de faptul că, în cazul în care nu există nici un fel de probleme de sănătate, este necesar să se ia în considerare faptul că, în cazul în care nu există probleme de sănătate, este necesar să se ia în considerare. (2014). Analiza proteomică a Lactobacillus helveticus H9 în timpul creșterii laptelui degresat. J. Lactate Sci. 97, 7413–7425. doi: 10.3168 / jds.2014-8520
rezumat PubMed / CrossRef Text Complet / Google Scholar
Dawson, K. L., Bell, A., Kawakami, R. P., Coley, K., Yates, G. și Collins, D. M. (2012). Transmiterea Mycobacterium orygis (specii complexe M. tuberculosis) de la un pacient cu tuberculoză la o vacă de lapte din Noua Zeelandă. J. Clin. Microbiol. 50, 3136–3138. doi: 10.1128 / JCM.01652-12
PubMed rezumat / CrossRef Text Complet / Google Scholar
Doi, K., Mori, K., Mutaguchi, Y., Tashiro, K., Fujino, Y., Ohmori, T. și colab. (2013). Proiect de secvență genomică a producătorului de aminoacizi cu lanț ramificat D Lactobacillus otakiensis JCM 15040t, izolat dintr-o murătură tradițională japoneză. Announc Genomului. 1, e546-e513. doi: 10.1128 / genomeA.00546-13
PubMed rezumat / CrossRef text integral / Google Scholar
Fernandez, M., van Doesburg, W., Rutten, G. A., Marugg, J. D., Alting, A. C., van Kranenburg, R. și colab. (2000). Analize moleculare și funcționale ale genei metC a Lactococcus lactis, care codifică cistationina beta-lyase. Appl. Environ. Microbiol. 66, 42–48. doi: 10.1128 / AEM.66.1.42-48.2000
rezumat PubMed / CrossRef Text Complet / Google Scholar
Franciosi, E., Carafa, I., Nardin, T., Schiavon, S., Poznanski, E., Cavazza, A. și colab. (2015). Biodiversitatea și producția de acid gamma-aminobutiric de către bacteriile acidului lactic izolate din brânzeturile tradiționale alpine din lapte de vacă crud. Biomed Res. Int. 2015, 625740. doi: 10.1155/2015/625740
PubMed rezumat / CrossRef Text Complet / Google Scholar
Fu, L., Niu, B., Zhu, Z., Wu, S. și Li, W. (2012). CD-HIT: accelerat pentru gruparea datelor de secvențiere de generație următoare. Bioinformatics 28, 3150–3152. doi: 10.1093/bioinformatics/bts565
PubMed Abstract | CrossRef Full Text | Google Scholar
Georgalaki, M. D., Sarantinopoulos, P., Ferreira, E. S., De Vuyst, L., Kalantzopoulos, G., and Tsakalidou, E. (2000). Biochemical properties of Streptococcus macedonicus strains isolated from Greek Kasseri cheese. J. Appl. Microbiol. 88, 817–825. doi: 10.1046/j.1365-2672.2000.01055.x
PubMed Abstract | CrossRef Full Text | Google Scholar
Gesudu, Q. M., Zheng, Y., Xi, X. X., Hou, Q. C., Xie, H. Y., Huang, W. Q. și colab. (2016). Investigarea structurii și dinamicii populației bacteriene în koumiss tradițional din Mongolia Interioară folosind secvențierea în timp real a unei singure molecule. J. Lactate Sci. 99, 7852–7863. doi: 10.3168 / jds.2016-11167
rezumat PubMed / CrossRef Text Complet / Google Scholar
Guarcello, R., Carpino, S., Gaglio, R., Pino, A., Rapisarda, T., Caggia, C. și colab. (2016). O aplicație la scară largă din fabrică a unor bacterii autohtone de acid lactic selectate pentru producția de brânză PDO Pecorino siciliano. Microbiol Alimentar. 59, 66–75. doi: 10.1016 / j. fm. 2016.05.011
rezumat PubMed / CrossRef Text Complet / Google Scholar
Hao, Y., Zhao, L., Zhang, H., Zhai, Z., Huang, Y., Liu, X. și colab. (2010). Identificarea biodiversității bacteriene în koumiss prin denaturarea electroforezei în gel de gradient și a reacției în lanț a polimerazei specifice speciilor. J. Lactate Sci. 93, 1926–1933. doi: 10.3168 / jds.2009-2822
rezumat PubMed / CrossRef Text Complet / Google Scholar
Helinck, S., Le Bars, D., Moreau, D. și Yvon, M. (2004). Capacitatea bacteriilor termofile de acid lactic de a produce compuși aromatici din aminoacizi. Appl. Environ. Microbiol. 70, 3855–3861. doi: 10.1128 / AEM.70.7.3855-3861.2004
PubMed rezumat / CrossRef text integral / Google Scholar
Hoskins, L. C., Agustines, M., McKee, W. B., Boulding, E. T., Kriaris, M. și Niedermeyer, G. (1985). Degradarea mucinei în ecosistemele colonului uman. Izolarea și proprietățile tulpinilor fecale care degradează antigenele grupului sanguin ABH și oligozaharidele din glicoproteinele mucinei. J. Clin. Investește. 75, 944–953. doi: 10.1172 / JCI111795
PubMed rezumat / CrossRef text integral / Google Scholar
Irmler, S., Raboud, S., Beisert, B., Rauhut, D. și Berthoud, H. (2008). Clonarea și caracterizarea a două gene Lactobacillus casei care codifică o cistationină liază. Appl. Environ. Microbiol. 74, 99–106. doi: 10.1128 / AEM.00745-07
PubMed Rezumat / CrossRef Text Complet / Google Scholar
Jagielski, V. (1877). Valoarea koumiss în tratamentul greaței, vărsăturilor și incapacității de a reține alte alimente pe stomac. Br. Med. J. 2, 919-921. doi: 10.1136 / bmj.2.887.919
rezumat PubMed / CrossRef Text Complet / Google Scholar
Kato, S., Ishihara, T., Hemmi, H., Kobayashi, H. și Yoshimura, T. (2011). Modificări ale concentrațiilor de D-aminoacizi și ale structurilor comunității microbiene în timpul fermentării vinurilor roșii și albe. J. Biosci. Bioeng. 111, 104–108. doi: 10.1016 / j. jbiosc.2010.08.019
PubMed rezumat / CrossRef text integral / Google Scholar
Li, H. și Durbin, R. (2009). Aliniere rapidă și precisă de citire scurtă cu transformarea Burrows-Wheeler. Bioinformatica 25, 1754-1760. doi: 10.1093 / Bioinformatica / btp324
PubMed rezumat / CrossRef textul integral / Google Scholar
Li, W. și Godzik, A. (2006). Cd-hit: un program rapid pentru gruparea și compararea seturi mari de secvențe de proteine sau nucleotide. Bioinformatica 22, 1658-1659. doi: 10.1093 / Bioinformatica / btl158
PubMed rezumat / CrossRef text integral / Google Scholar
Miyamoto, M., Seto, T., Nakajima, H., Burenjargal, S., Gombojav, A., Demberei, S. și colab. (2010). Denaturarea gel gradient electroforeză analiza bacteriilor de acid lactic și drojdii în lapte fermentat tradițional mongol. Alimente Sci. Tehnologie. Res. 16, 319-326. doi: 10.3136 / fstr.16.319
CrossRef Text Complet / Google Scholar
Mulder, N. J. și Apweiler, R. (2008). Baza de date InterPro și instrumente pentru analiza domeniului proteinelor. Curr. Protocolul. Bioinformatica Capitolul 2, Unit2. 7. doi: 10.1002 / 0471250953. bi0207s21
PubMed rezumat / CrossRef Text Complet / Google Scholar
Noguchi, H., Park, J. și Takagi, T. (2006). MetaGene: găsirea genei procariote din secvențele de pușcă ale genomului de mediu. Acizi Nucleici Res. 34, 5623-5630. doi: 10.1093 / nar / gkl723
PubMed rezumat / CrossRef Text Complet / Google Scholar
Nurk, S., Bankevich, A., Antipov, D., Gurevich, A. A., Korobeynikov, A., Lapidus, A. și colab. (2013). Asamblarea genomilor cu o singură celulă și a mini-metagenomilor din produsele himeric MDA. J. Comput. Biol. 20, 714–737. doi: 10.1089 / cmb.2013.0084
PubMed rezumat / CrossRef text integral / Google Scholar
Ott, A., Germond, J. E. și Chaintreau, A. (2000). Originea acetaldehidei în timpul fermentării laptelui folosind (13) precursori marcați cu C. J. Agric. Chimie Alimentară. 48, 1512–1517. doi: 10.1021 / jf9904867
PubMed rezumat / CrossRef Text Complet / Google Scholar
Pan, D. D., Zeng, X. Q. și Yan, Y. T. (2011). Caracterizarea Lactobacillus fermentum SM – 7 izolat din koumiss, o potențială bacterie probiotică cu efecte de scădere a colesterolului. J. Sci. Mâncare. Agric. 91, 512–518. doi: 10.1002 / jsfa.4214
Rezumat PubMed / CrossRef Text Complet / Google Scholar
Papadimitriou, K., Anastasiou, R., Maistrou, E., Plakas, T., Papandreou, N. C., Hamodrakas, S. J. și colab. (2015). Achiziționarea prin transfer orizontal de gene a plasmidei pSMA198 de către Streptococcus macedonicus ACA-DC 198 indică originea lactată a speciei. PLoS Unul 10: e0116337. doi: 10.1371/jurnal.pone.0116337
PubMed rezumat / CrossRef Text Complet / Google Scholar
Segata, N., Waldron, L., Ballarini, A., Narasimhan, V., Jousson, O. și Huttenhower, C. (2012). Profilarea comunității microbiene metagenomice folosind gene marker unice specifice cladei. Nat. Metode 9, 811-814. doi: 10.1038 / nmeth.2066
rezumat PubMed / CrossRef Text Complet / Google Scholar
Sun, Z., Liu, W., Zhang, J., Yu, J., Zhang, W., Cai, C. și colab. (2010). Identificarea și caracterizarea lactobacililor dominanți izolați din koumiss în China. J. Gen. Appl. Microbiol. 56, 257–265. doi: 10.2323 / jgam.56.257
Rezumat PubMed / CrossRef Text Complet / Google Scholar
Thompson, J. (1879). Valoarea koumiss în irosirea bolilor. Br. Med. J. 1, 270. doi: 10.1136 / bmj.1.1466.270-c
rezumat PubMed / CrossRef Text Complet / Google Scholar
Vincent, S. J., Faber, E. J., Neeser, J. R., Stingele, F. și Kamerling, J. P. (2001). Structura și proprietățile exopolizaharidei produse de Streptococcus macedonicus Sc136. Glicobiologie 11, 131-139. doi: 10.1093 / glycob / 11.2.131
PubMed rezumat / CrossRef text integral / Google Scholar
Wang, L., Christophersen, C. T., Sorich, M. J., Gerber, J. P., Angley, M. T. și Conlon, M. A. (2013). Abundență crescută de Sutterella spp. și cuplurile de Ruminococcus în fecalele copiilor cu tulburare de spectru autist. Mol. Autism 4, 42. doi: 10.1186/2040-2392-4-42
PubMed rezumat / CrossRef Text Complet / Google Scholar
Ward, T. L., Hosid, S., Ioshikhes, I. și Altosaar, I. (2013). Metagenomul laptelui uman: o analiză a capacității funcționale. BMC Microbiol. 13:116. doi: 10.1186/1471-2180-13-116
PubMed rezumat / CrossRef Text Complet / Google Scholar
Watanabe, K., Fujimoto, J., Tomii, Y., Sasamoto, M., Makino, H., Kudo, Y. și colab. (2009). Lactobacillus kisonensis sp. nov., Lactobacillus otakiensis sp. nov., Lactobacillus rapi sp. nov. și Lactobacillus sunkii sp. nov., specii heterofermentative izolate de sunki, o murătură tradițională japoneză. Int. J. Syst. Evol. Microbiol. 59 (Pt 4), 754-760. doi: 10.1099/ijs.0.004689-0
PubMed rezumat / CrossRef Text Complet / Google Scholar
Wu, R., Wang, L., Wang, J., Li, H., Menghe, B., Wu, J. și colab. (2009). Izolarea și selecția probiotică preliminară a lactobacililor din koumiss în Mongolia Interioară. J. Microbiol De Bază. 49, 318–326. doi: 10.1002 / jobm.200800047
PubMed rezumat / CrossRef text integral / Google Scholar
Zerbino, D. R. și Birney, E. (2008). Catifea: algoritmi pentru asamblare de novo scurtă citire folosind grafice de Bruijn. Genomul Res. 18, 821-829. doi: 10.1101 / gr.074492.107
PubMed rezumat / CrossRef Text Complet / Google Scholar
Zhang, W., soare, Z., soare, T. și Zhang, H. (2010a). Screeningul PCR și analiza secvențială a clusterelor iol la tulpinile de Lactobacillus casei izolate din koumiss. Folia Microbiol. (Praha) 55, 603-606. doi: 10.1007 / s12223-010-0097-3
PubMed rezumat / CrossRef Text Complet / Google Scholar
Zhang, W., Yu, D., soare, Z., Wu, R., Chen, X., Chen, W. și colab. (2010b). Secvența completă a genomului Lactobacillus casei Zhang, o nouă tulpină probiotică izolată din koumiss-ul tradițional de casă din Mongolia Interioară, China. J. Bacteriol. 192, 5268–5269. doi: 10.1128 / JB.00802-10
PubMed rezumat / CrossRef text integral / Google Scholar
Zhang, W. și Zhang, H. (2011). „Fermentarea și koumiss”, în manualul tehnologiei de fermentare a alimentelor și băuturilor, 2nd Edn, ed. Y. H. Hui( Boca Raton, FL: CRC Press), 165-172.
Google Scholar
Zhang, W. Y., Chen, Y. F., Zhao, W. J., Kwok, L. Y. și Zhang, hp (2015). Expresia genetică a sistemului proteolitic al Lactobacillus helveticus H9 în timpul fermentării laptelui. Ann. Microbiol. 65, 1171–1175. doi: 10.3168 / jds.2014-8520
rezumat PubMed / CrossRef Text Complet / Google Scholar
Zhao, W., Chen, Y., soare, Z., Wang, J., Zhou, Z., soare, T. și colab. (2011). Secvența completă a genomului Lactobacillus helveticus H10. J. Bacteriol. 193, 2666–2667. doi: 10.1128 / JB.00166-11
PubMed Rezumat / CrossRef Text Complet / Google Scholar