- introduktion
- materialer og metoder
- forberedelse af prøver
- Gradientfortynding og multipelt Forskydningsamplifikation
- Bibliotekskonstruktion og sekventering
- dataanalyse
- Sekvenskvalitetskontrol og filtrering
- taksonomisk opgave og mangfoldighed
- Læs samling, Genforudsigelse og Annotation
- Nukleotidsekvenstiltrædelsesnumre
- resultater
- eksperimentelt Design og sekventering
- taksonomisk kommentar
- Metagenomisk samling, Genforudsigelse og funktionel Annotation
- Diskussion
- konklusion
- Forfatterbidrag
- interessekonflikt Erklæring
- anerkendelse
- supplerende materiale
introduktion
Koumiss, også kaldet chige, chigo, arrag eller airag, på det mongolske sprog, er en type traditionelt gæret mejeriprodukt. Det har været en populær mad i Mongoliet og Det Indre Mongoliet i Kina i århundreder (2011). Folk i disse regioner plejede at forbruge koumiss under store festligheder og ofre (Jang og Jang, 2011). Den tidligste registrering af koumiss-produktion kan spores tilbage til Han-dynastiet (BC202-AD202). Dette produkt havde opnået udbredt popularitet under Yuan-dynastiet (AD1271-AD1368) (Jang et al., 2010b). I dag er koumiss en almindelig mad til lokalbefolkningen i Mongoliet og Indre Mongoliet, men kun i få af disse områder fremstilles den i industriel skala. Koumiss leverer ikke kun rige næringsstoffer, herunder højt indhold af essentielle aminosyrer og vitaminer, men menes også at lindre en lang række medicinske tilstande og er gavnlig for postoperativ pleje (Jagielski, 1877; Thompson, 1879).
traditionelt produceres koumiss almindeligvis i træfade, containere lavet af dyrehud eller urner. Fermentering forekommer naturligt ved omgivelsestemperatur efter tilsætning af filtreret hoppemælk i beholderen med gammel koumiss, der tjener som startkultur (Jang og Jang, 2011). Koumiss er en god kilde til nye bakterier af bioteknologisk potentiale (Jang et al., 2010A; Pan et al., 2011). Derfor er det af intens interesse at udforske og bevare så mange gæringsassocierede koumiss-bakterier som muligt. I løbet af de sidste årtier blev der udført en række undersøgelser for at undersøge koumiss bakterielle samfund., 2009; Hao et al., 2010), hovedsageligt studeret ved Kultur -, molekylærbiologi-og pyrosekvensbaserede metoder (Sun et al., 2010). Blandt disse forskellige tilgange, den pyrosekvensbaserede metode har givet den mest omfattende mikrobiotaprofil for koumiss uafhængig af fænotypiske træk og problemer med dyrkbarhed af de enkelte mikrober. Spektret af funktionelle gener kodet af koumiss-bakterierne og deres fermentative kapacitet forbliver imidlertid dårligt karakteriseret, især for de sjældne mikrobielle populationer.
denne undersøgelse anvendte genomik-teknikken med en enkelt celle til at analysere bakteriemetagenomer af 10 koumiss-prøver indsamlet fra Mongoliet og Indre Mongoliet i Kina. Det nuværende arbejde har anvendt avancerede teknologier til at undersøge den bakterielle mangfoldighed i mejeriprodukter. Vores arbejde har vist muligheden for at opdage lav-rigelige taksa ved at anvende enkelt celle metagenomics tilgang. De opmuntrende resultater vil fremme udviklingen og anvendelsen af nye tilgange til at tackle problemer inden for et traditionelt forskningsområde.
materialer og metoder
forberedelse af prøver
i alt 10 koumiss-prøver blev indsamlet fra Mongoliet (MG14, MG15, MG16, MG17 og MG18) og Kinas indre mongoliet (Nm17, NM18, NM19, NM20 og NM21) til metagenomics-undersøgelsen. Prøver blev indsamlet aseptisk og blev transporteret i tøris.
en milliliter af hver prøve blev forbehandlet ifølge metoden beskrevet i afdeling et al. (2013) med nogle ændringer. Kort fortalt blev prøverne optøet i et isbad i 3-5 minutter. Efter at prøverne var smeltet, blev de udsat for centrifugering med lav hastighed for at fjerne urenheder og eukaryote celleklumper. Prokaryote celler blev derefter pelleteret fra mælkeseraen ved centrifugering ved 13.000 liter g i 15 minutter. Pellets blev igen suspenderet i 2 mL phosphatbufret saltvand (PBS) med 1% Triton H-100 og inkuberet i 2 timer ved 37 liter C for at lyse eventuelle resterende eukaryote celler. Derefter blev bakterier pelleteret ved centrifugering ved 13.000 liter g i 15 minutter, og pellets blev suspenderet igen i 500 liter PBS. Endelig blev centrifugeringstrinnet gentaget igen for at vaske bakteriecellerne.
Gradientfortynding og multipelt Forskydningsamplifikation
for at detektere de lave rigelige bakterier blev bakteriesuspensionen afledt af hver koumiss-prøve fortyndet serielt til efterfølgende amplifikationsreaktion. Celletallet i hver prøve blev groft estimeret under et mikroskop (Nikon, Tokyo, Japan) ved hjælp af et celletællingskammer (Chiujing, Shanghai, Kina). Fortyndingstrinnet blev fortsat, indtil celletallet i hver bakteriesuspension nåede ca.100. Multipelt forskydningsforstærkning af de fortyndede celler blev udført under anvendelse af REPLI-g-Enkeltcellesættet (CHIAGEN, MD, USA) i henhold til producentens anvisninger.
Bibliotekskonstruktion og sekventering
forstærket DNA blev klippet tilfældigt, og fragmenterne på cirka 500 bp blev valgt. Efter bibliotekets konstruktion blev PerkinElmer LabChip touch og StepOnePlusTM real-time PCR-System brugt til bibliotekskvalitetskontrol. Endelig blev 125-bp-parrede slutlæsninger sekventeret på Illumina 2500-platformen i henhold til producentens anvisninger.
dataanalyse
Sekvenskvalitetskontrol og filtrering
Rålæsninger genereret af sekvenseren kan indeholde kunstige aflæsninger af adapterforurening under bibliotekets konstruktion. Derfor blev der udført tre trin for at opnå et rent læsedatasæt af høj kvalitet: (1) eliminering af læsninger forårsaget af adapterforurening; (2) Fjernelse af læsninger med en gennemsnitlig score under en phred-score på 30.kvartal, som blev betragtet som den laveste cutoff for en base af høj kvalitet; (3) Fjernelse af læsninger med et betydeligt overskud på “N” (5% af læsningen). Nedstrømsanalysen var baseret på de rene data. Desuden blev den statistiske basiskvalitet baseret på 30.kvartal og GC-indholdet beregnet.
justeringer mod værtsgenomet blev udført for at fjerne de værtsoprindede forureningssekvenser. Enhver vært-Oprindelse læser blev kasseret før yderligere sammenligning med bakterier (eller vira) genomreferencesekvenser. For at opnå mere nøjagtige resultater blev Gravhjulets aligner (Version 0.97 a) mem–modellen brugt i justeringerne (Li og Durbin, 2009).
taksonomisk opgave og mangfoldighed
internetprogrammet Metaflan blev brugt til taksonomisk tildeling til slægts-og artsniveauer (Segata et al., 2012). For at sammenligne mangfoldigheden af arter inden for og mellem prøver analyserede vi alfa – og beta-mangfoldigheden ved hjælp af den R-relaterede pakke.
Læs samling, Genforudsigelse og Annotation
for at få mere omfattende information samlede vi de klippede fragmenter i genom (contigs). På grund af tilstedeværelsen af flere arter, som er almindelig i metagenomiske prøver, forbedrede vi imidlertid bioinformatikgenomsamlingsmetoden, der normalt blev brugt til analyse af enkeltarter ved at integrere Spar (Version 3.6.2), interne scripts og metagenomiske databaser (nul og Birney, 2008; Nurk et al., 2013).
MetaGeneMark-programmet blev brugt til genforudsigelse af de samlede contigs (Noguchi et al., 2006). Redundante gener blev fjernet ved hjælp af CD-HIT med dækning af 90 og 95% identitet (Li og Godsik, 2006; Fu et al., 2012). Relative mængder af generne blev bestemt ved at tilpasse sekventering af høj kvalitet til genkataloget ved hjælp af den samme procedure. Den nedstrøms uoverensstemmende analyse var baseret på genets relative overflod. Genanmærkning blev udført ved at tilpasse sekvenserne af høj kvalitet mod flere offentlige databaser (nemlig NCBI ikke-redundant database, NR; klynger af Ortologe grupper af proteiner, tandhjul; Kyoto Encyclopedia of Genes and Genomes, KEGG) ved hjælp af BLAST (Altschul et al., 1997). En domænesøgning blev udført ved hjælp af Interproscan (2008).
Nukleotidsekvenstiltrædelsesnumre
sekvensdata rapporteret i denne undersøgelse er deponeret i Sra-databasen (Tiltrædelsesnr.SRP083102).
resultater
eksperimentelt Design og sekventering
for at detektere de lave rigelige bakterier i koumiss blev enkeltcelleforstærkningsteknikken brugt til at analysere metagenomerne af prøverne. 100 celler, blev afledt af en uafhængig koumiss-prøve ved seriel fortynding. I alt 30 fortyndede suspensioner blev analyseret. Hver fortyndet prøve fik en anden prøvekode, dvs. prøveidentitetsnummeret efterfulgt af 1, 2 eller 3, der repræsenterer de tre separate fortyndinger. Baseret på en forudsætning om, at nogle sjældne arter vil være til stede i en af fortyndingerne, blev der udført flere forskydningsforstærkning af cellerne; og omkring 5 Gb data blev genereret for hver koumiss bakteriesuspension.
i alt 1.040.323.864 rålæsninger blev genereret fra de 10 koumiss-prøver (i alt 30 bakteriesuspensioner). Det gennemsnitlige antal aflæsninger for hver 100-cellesuspension var 34.677.462 (supplerende tabel S1). Efter trimning og filtrering af de ukvalificerede sekvenser opnåede vi 1.018.381.702 rene aflæsninger for alle prøver (supplerende tabel S1). Værdierne for Shannon-indeks, Simpson-indeks, Chao1-indeks og antallet af observerede arter (figur 1-4) viste, at de fleste koumiss-prøver havde en høj bakteriel biodiversitet. Shannon-viener-mangfoldighedskurverne viste, at sekvensdybden var tilstrækkelig til alle prøver (Figur 1).
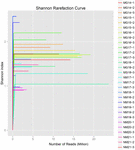
FIGUR 1. Shannon rarefaction kurver, der estimerer den mikrobielle mangfoldighed af koumiss prøver.
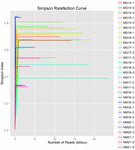
FIGUR 2. Simpson rarefaction kurver, der estimerer den mikrobielle mangfoldighed af koumiss prøver.
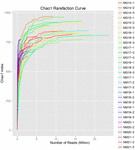
FIGUR 3. Chao1 sjældne kurver, der estimerer den mikrobielle mangfoldighed af koumiss-prøver.
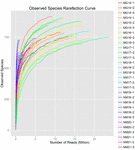
FIGUR 4. Observeret Art rarefaction indeks, der estimerer den mikrobielle mangfoldighed af koumiss prøver.
taksonomisk kommentar
sekvenserne af høj kvalitet blev tildelt forskellige taksonomiske niveauer for at muliggøre en dybdegående analyse af prøvebakteriesamfundene. Med henvisning til nogle offentliggjorte undersøgelser af koumiss biodiversitet (vu et al., 2009; Hao et al., 2010; Sun et al., 2010), klassificerede vi de kendte og tidligere ikke rapporterede koumiss-associerede bakterier som henholdsvis almindelige og sjældne takser.
sekvenserne af høj kvalitet repræsenterede 13 forskellige slægter (figur 5). Tre af dem havde en gennemsnitlig relativ overflod på over 1%, inklusive Lactobacillus (L.), Lactococcus og Streptococcus. Især Lactobacillus og Lactococcus var de to mest udbredte slægter fundet i koumiss. Andelen af Lactobacillus i prøverne varierede fra 52,72 til 99,96%. To medlemmer af denne slægt, L. helveticus og L. kefiranofaciens, blev domineret blandt de fleste koumiss-prøver (figur 6). Arten L. buchneri var hovedsagelig til stede i de mongolske prøver, mens Lactococcus lactis blev påvist i de fleste prøver uanset prøveudtagningsregionen.
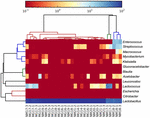
FIGUR 5. Heatmap, der viser den relative overflod af bakterier, der er påvist i koumiss-prøverne på slægtsniveau.
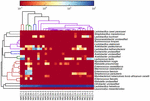
FIGUR 6. Heatmap, der viser den relative overflod af bakterier, der er påvist i koumiss-prøverne på artsniveau.
Metagenomisk samling, Genforudsigelse og funktionel Annotation
samling af læsningerne resulterede i en samlelængde på 614.392.623 bp. N50-værdierne for samlingerne varierede fra 5.596 til 35.200 bp (supplerende tabel S2). Antallet af forudsagte gener i koumiss-prøverne varierede fra 10.347 til 34.547 med en gennemsnitlig længde fra 647,82 til 985,33 bp (supplerende tabel S3). Selvom empiriske genfunktionelle analyser var uden for rammerne af den aktuelle undersøgelse, vi kommenterede koumiss bakterielle mikrobiomer ved hjælp af COG-og KEGG-databaserne, som forudsagde genfunktion stort set baseret på sekvenshomologi.
i alt 545 kampe i annotationsudgangen viste høj homologi til laktoseudnyttelsesgenerne (COGs kategori af kulhydrat og metabolisme, G), og nogle af dem var placeret inden for samme contig (tabel 1).
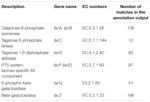
TABEL 1. Laktosemetabolismerelaterede gener kommenteret i koumiss bakteriemetagenom.
nogle andre sekvenser kan kode for formodede gener inden for COGs-kategorien af aminosyretransport og metabolisme (E), herunder sekvenser, der svarede til kaseinnedbrydende proteinaser, Opp-systemet til optagelse af oligopeptider på 4-18 rester og aminopeptidaser (f.eks. leucylaminopeptidase, peptidyl-dipeptidase a, aminopeptidase N, proliniminopeptidase og aminopeptidase endopeptidase). Selvom nogle sekvenser delte høje ligheder med de aminotransferaser, der var specifikke for arginin, aspartat, methionin og isoleucin, blev der kun identificeret en signifikant match, hvilket svarede til en formodet S. macedonicus-oprindelig klasse i/klasse II domæne (IPR004839)-indeholdende aminotransferase specifik for tyrosin og phenylalanin. Endelig delte et antal sekvenser høj homologi med aminosyrelyaserne, herunder S-ribosylhomocysteinlyase, argininosuccinatlyase, aspartat ammoniak-lyase, cystathionin gamma-lyase, histidin ammoniak-lyase og O-acetylhomoserin (thiol)-lyase.
Diskussion
Koumiss er et populært traditionelt gæret mejeriprodukt i Mongoliet og Indre Mongoliet i Kina. Selvom der er udført en række undersøgelser for at undersøge bakteriediversiteten i koumiss, er der kun opnået lidt information om koumiss mikrobiotas genetiske kapacitet. Her anvendte vi enkeltcelleforstærkningsteknikken til at analysere koumiss bakterielle metagenomer, især med fokus på den lave rigelige bakteriepopulation.
den typiske metagenomiske tilgang er tidligere blevet anvendt til at beskrive koumiss mikrobiota. På grund af de høje omkostninger ved at opnå en dyb sekvens dækkes den sjældne mikrobiotapopulation i prøverne imidlertid ofte utilstrækkeligt. Således forbliver både de fylogene og funktionelle metagenomer af minoritetsbakterierne begrænsede. Vores encellede amplifikationsmetode involverede seriel fortynding af prøver til 100-cellesuspensioner. På grund af det lave antal celler, der er til stede i de fortyndede koumiss-prøver, ville kun en begrænset mængde DNA blive ekstraheret. Amplifikationstrinnet øgede mængden af DNA-materialer, der skulle analyseres, og letter dermed metagenomanalysen af prøver indeholdende små DNA-mængder. En ulempe ved denne metode var vanskeligheden ved at sikre, at sekvenser af hver takson ville blive lige forstærket i processen; derfor kunne resultaterne opnået her kun afspejle de relative mængder af sekvenser, men ikke absolutte mængder af identificerede taksa eller funktionelle gener. Alligevel ville dette have ringe effekt på vores Analyse, da den nuværende undersøgelse adskiller sig fra andre offentliggjorte værker med hensyn til at fokusere den sjældne bakteriepopulation. Data genereret af dette arbejde giver supplerende oplysninger til den underrepræsenterede befolkning. Vi mener, at den nuværende tilgang er egnet til fremtidig analyse af bakteriediversitet for forskellige typer økologiske miljøer.
koumiss bakteriemikrobiota består hovedsageligt af mælkesyrebakterier (LAB) og nogle eddikesyrebakterier (Chang and Chang, 2011). Som forventet indeholdt vores datasæt for det meste sekvenser, der repræsenterede laboratoriet og eddikesyrebakterierne. Sekvenser svarende til Lactobacillus helveticus dominerede på tværs af alle prøver. Desuden sekvenser, der repræsenterer arten, L. kefiranofaciens. L. buchneri. L. kefiranofaciens. Enterococcus (E.) casseliflavus. E. faecalis. E. faecium. Leuconostoc mesenteroides. Lactococcus lactis og Acetobacter pasteurianus blev også fundet. Identifikationen af sekvenser af forskellige takser er muligvis ikke nok til at vise bakteriens levedygtighed, det afspejler ikke desto mindre bakteriesamfundet på et eller andet tidspunkt i fermenteringsprocessen. Miyamoto et al. (2010) antyder, at bakterieprævalensen i de endelige fermenterede produkter er relateret til deres syrestresstolerance. Generelt har lactobaciller en højere syretolerance end lactokokker, hvilket kan forklare den høje relative overflod af lactobaciller-sekvenser, der findes i vores datasæt. På den anden side er den hyppige forekomst af L. helveticus i koumiss faldt sammen med observationen af dominans af L. helveticus sekvenser.
i vores datasæt svarede nogle sekvenser til L. otakiensis, som er en sjælden art, der aldrig er rapporteret i koumiss eller andre mejeri nicher. Arten blev først beskrevet og isoleret fra den ikke-saltede picklingopløsning, der blev brugt til fremstilling af sunki, en traditionel japansk pickle., 2009). Det blev opdaget ved amplificeret fragment længde polymorfisme profilering baseret på recA genet. Siden da er denne art ikke rapporteret at være forbundet med andre fødevarerelaterede nicher. Det er således sandsynligt, at denne bakterie tilhører den autoktone flora af pickles. Vi kan dog ikke udelukke muligheden for, at den ikke blev fundet blot på grund af følsomheden af den anvendte detektionsmetode. Lactobacillus otakiensis kan producere D-forgrenede aminosyrer, såsom d-leucin, d-allo-isoleucin og d-valin (Doi et al., 2013). Det har potentiale til at blive brugt til at forbedre produktionsegenskaberne for visse gærede fødevarer (Kato et al., 2011).
vores undersøgelse identificerede også sekvenser, der repræsenterer arten S. macedonicus, som aldrig er blevet rapporteret som en koumiss-associeret bakterie. I stedet er det en startkultur, der findes i græske får-og gedeost (Georgalaki et al., 2000). Nogle medlemmer af denne art er i stand til at producere eksopolysaccharider, bakteriociner (Vincent et al., 2001; Anastasiou et al., 2015) og gamma-aminosmørsyre (Franciosi et al., 2015). Selvom denne art ofte er isoleret fra gærede fødevarer, har den oprindelige niche af S. macedonicus været kontroversiel (Guarcello et al., 2016). Først for nylig, Papadimitriou et al. (2015) identificerede et erhvervet plasmid, pSMA198, fra Lactococcus lactis. Plasmidet blev sandsynligvis overført via en forfædres genetisk udvekslingshændelse inden for et mejeriproduktmiljø, hvilket antydede mejerioprindelsen af S. macedonicus (Papadimitriou et al., 2015). Svarende til S. thermophilus. S. macedonicus er nært beslægtet med de kommensale og opportunistiske patogener i S. bovis/S. hestekomplekset.
identifikationen af sekvenser, der repræsenterer arten Ruminococcus-drejningsmomenter, var uventet, da denne bakterie normalt findes i tarmmiljøet. Det er en normal human tarmmikrobe, der kan nedbryde mucinoligosaccharider ved konstitutiv produktion af sekretoriske glycosidaser (Hoskins et al., 1985). Nylige kliniske beviser viser, at den fækale overflod af denne art ændres hos børn med autismespektrumforstyrrelse; dens rolle i lidelsen forbliver imidlertid uklar., 2013).
overraskende repræsenterede nogle af sekvenserne potentielle patogener. For eksempel er Klebsiella lungebetændelse et opportunistisk humant patogen, der ligger i omkring 40% af menneskers og dyrs tarm. Mycobacterium orygis, tidligere kaldet oryks bacillus, er medlem af Mycobacterium tuberculosis-komplekset, der kan forårsage human tuberkulose., 2012). Disse to arter er blevet rapporteret i rå mælk, men ikke koumiss. Derfor kunne deres tilstedeværelse tilskrives forureningen under konventionel koumiss – produktion, især under ikke-eller lavaseptiske manipulationsbetingelser.
bakteriel metabolisme spiller en vigtig rolle i dannelsen af koumiss egenskaber og kvalitet. Mikrobielle baserede processer såsom lipolyse og proteolyse er nødvendige for at syntetisere koumiss aromatiske og smagsforbindelser (Gesudu et al., 2016). Konsekvent indeholdt det bakterielle metagenom sekvenser, der potentielt koder for laktosedbrydning og proteolytiske systemer. I modsætning til de relativt enkle laktosekataboliske veje består LABORATORIEPROTEOLYTISKE systemer af en bred vifte af f.eks., 2014). Sammenlignet med andre detekterede koumiss LAB er den dominerende art L. helveticus karakteriseret med en høj proteolytisk aktivitet (Jang et al., 2015). De fleste laboratorier har kun en cellehylsterproteinase, der initierer mælkekaseinhydrolysen, mens L. helveticus indeholder mindst to af disse stoffer, nemlig PrtH og PrtH2., 2011). Således kan de høje andele af sekvenser svarende til de stærke proteolytiske arter af L. helveticus og proteolyserelaterede gener forbinde med det relativt høje indhold af peptider og frie aminosyrer i koumiss.
et problem i industriel skala koumiss produktion er kontrollen med smag opfattelse, som koumiss er traditionelt blevet gjort ved naturlig gæring. Det har således været svært at definere smagen og profilen af vigtige smagskomponenter, især i nærværelse af de naturlige forurenende stoffer. Produktionen af vigtige smagskomponenter er et resultat af fermentativ og fermentativ nedbrydning af aminosyrer, såsom de forgrenede aminosyrer, methionin og aromatiske aminosyrer (Ardo, 2006). Eksempler på smagskomponenter inkluderer aldehyder, organiske syrer og estere, som dannes af transaminase (at)-Vej (Helinck et al., 2004). Denne vej initieres af en transaminase, der katalyserer omdannelsen af en aminosyre til dens tilsvarende Purpur-keto-syre (Helinck et al., 2004). Nogle sekvenser i vores datasæt svarede til formodede forgrenede og aromatiske aminosyreaminotransferaser. Især fandt vi en formodet aromatisk aminosyre aminotransferase i i contig af koumiss-associerede arter, S. macedonicus. Da vores nuværende arbejde kun kommenterede mikrobiomet i silico, er vores data ikke nok til at vise, at disse identificerede gensekvenser faktisk var funktionelle til at producere de førnævnte koumiss smagsforbindelser. Tilstedeværelsen af disse gener antyder ikke desto mindre, at de er nogle mulige kandidater til sådanne fermentative aktiviteter; endnu, yderligere eksperimentelt arbejde ville være nødvendigt for at belyse deres nøjagtige funktionelle roller.
desuden fandt vi sekvenser svarende til andre aminosyrekonverteringsveje inklusive aminosyrelyaser og threonin aldolase. En af de mest almindelige årsager til methionin er methanethiol (Irmler et al., 2008), hvilket resulterer i dannelse af dimethyldisulfid og dimethyl trisulfid., 2000), mens sidstnævnte katalyserer omdannelsen af threonin til glycin og acetaldehyd (Ott et al., 2000). Tilsvarende kan blot lokalisering af disse sekvenser inden for koumiss-mikrobiomet ikke tjene som konkret bevis i deres faktiske biologiske roller; fremtidig eksperimentel bekræftelse vil være påkrævet.
konklusion
den nuværende undersøgelse anvendte en modificeret metagenomik-metode til at analysere bakteriemikrobiomet af koumiss-prøver indsamlet fra Mongoliet og Indre Mongoliet i Kina. Vi karakteriserede både de fylogene og funktionelle metagenomer af de sjældne arter i koumiss; og vores datasæt afspejler træk, der er af bioteknologisk interesse og potentiale. Vores undersøgelse har for første gang vist muligheden for at inkorporere enkeltcelleforstærkningsteknikkerne til påvisning af koumiss bakteriel mikrobiota såvel som mikrobielle forurenende stoffer. De teknikker, der er udviklet heri, kan bruges i fremtidige undersøgelser til at overvåge ændringer i koumiss-mikrobiomet langs fermenteringsprocessen, med fokus på den mikrobielle minoritetspopulation. Desuden kan andre omics-teknikker, såsom transcriptomics, metabolomics, kobles til den aktuelle metagenomics-analyse for at bekræfte koumiss-mikrobiomets funktioner og metaboliske kapacitet.
teknisk set ville det næste trin i dette arbejde være at optimere den nuværende metode. For eksempel ved at øge prøvefortyndingen før DNA-amplifikation kan chancen for at afdække sjældne og nye bakterier forbedres. På den anden side kan en alternativ sekventeringsteknik, der kan generere lange læsninger, såsom Pacific Biosciences single molecule, realtids sekventeringsteknologi, anvendes til at forbedre genomsamlingsprocessen.
Forfatterbidrag
designet undersøgelsen. Jy, jy og L-YK skrev manuskriptet. H, H, H, BM og TS udførte eksperimenter. DF og DF har analyseret data. Alle forfattere gennemgik manuskriptet.
interessekonflikt Erklæring
forfatterne erklærer, at forskningen blev udført i mangel af kommercielle eller økonomiske forhold, der kunne fortolkes som en potentiel interessekonflikt.
anerkendelse
denne forskning blev støttet af National Natural Science Foundation of China (Grant No. 31571815).
supplerende materiale
det supplerende materiale til denne artikel kan findes online på: https://www.frontiersin.org/article/10.3389/fmicb.2017.00165/full#supplementary-material
Altschul, S. F., Madden, T. L., Schaffer, A. A., J., J., J., Miller, v., et al. (1997). Gapped BLAST og PSI-BLAST: en ny generation af protein database søgeprogrammer. Nukleinsyrer Res. 25, 3389-3402. doi: 10.1093/nar / 25.17.3389
PubMed abstrakt / CrossRef Fuld tekst / Google Scholar
det er en af de mest almindelige årsager til denne sygdom. (2015). Konstruerede stammer af Streptococcus macedonicus mod en osmotisk stressresistent fænotype bevarer deres evne til at producere bacteriocin macedocin under hyperosmotiske forhold. J. Biotechnol. 212, 125–133. doi: 10.1016 / j. jbiotec.2015.08.018
PubMed Abstrakt / CrossRef Fuld Tekst / Google Scholar
Ardo, Y. (2006). Smagsdannelse ved aminosyrekatabolisme. Biotechnol. Adv. 24, 238-242. doi: 10.1016 / j.biotechadv.2005.11.005
PubMed abstrakt / CrossRef Fuld tekst / Google Scholar
J. J., J. J., J. N., Sun, J. H., J. Y., J. C., Et Al. (2014). Proteomanalyse af Lactobacillus helveticus H9 under vækst i skummetmælk. J. Mejeri Sci. 97, 7413–7425. doi: 10.3168 / jds.2014-8520
PubMed abstrakt / CrossRef Fuld tekst / Google Scholar
han er en af de mest kendte i verden, og han er en af de bedste i verden (2012). Overførsel af Mycobacterium orygis (M. tuberculosis komplekse arter) fra en tuberkulosepatient til en malkeko i Danmark. J. Clin. Mikrobiol. 50, 3136–3138. doi: 10.1128 / JCM.01652-12
PubMed abstrakt / CrossRef Fuld tekst / Google Scholar
Doi, K., Mori, K., Mutaguchi, Y., Tashiro, K., Fujino, Y., Ohmori, T., et al. (2013). Udkast til genomsekvens af D-forgrenet aminosyreproducent Lactobacillus otakiensis JCM 15040t, isoleret fra en traditionel japansk pickle. Genom Announc. 1, e546-e513. doi: 10.1128 / genomeA.00546-13
PubMed abstrakt / CrossRef Fuld tekst / Google Scholar
det er en af de mest populære måder at gøre dette på. (2000). Molekylære og funktionelle analyser af Metc-genet af Lactococcus lactis, der koder for cystathionin beta-lyase. Appl. Environ. Mikrobiol. 66, 42–48. doi: 10.1128 / AEM.66.1.42-48.2000
PubMed abstrakt / CrossRef Fuld tekst / Google Scholar
I., Nardin, T., Schiavon, S., Posnanski, E., Carafa, A., et al. (2015). Biodiversitet og gamma-aminosmørsyre produktion af mælkesyrebakterier isoleret fra traditionelle alpine rå komælk oste. Biomed Res. Int. 2015, 625740. doi: 10.1155/2015/625740
PubMed abstrakt / CrossRef Fuld tekst / Google Scholar
Fu, L., Niu, B., Hu, S., og Li, H. (2012). CD-HIT: accelereret til gruppering af næste generations sekventeringsdata. Bioinformatics 28, 3150–3152. doi: 10.1093/bioinformatics/bts565
PubMed Abstract | CrossRef Full Text | Google Scholar
Georgalaki, M. D., Sarantinopoulos, P., Ferreira, E. S., De Vuyst, L., Kalantzopoulos, G., and Tsakalidou, E. (2000). Biochemical properties of Streptococcus macedonicus strains isolated from Greek Kasseri cheese. J. Appl. Microbiol. 88, 817–825. doi: 10.1046/j.1365-2672.2000.01055.x
PubMed Abstract | CrossRef Full Text | Google Scholar
Gesudu, Q. M., Zheng, Y., Xi, X. X., Hou, Q. C., Xie, H. Y., Huang, et al. (2016). Undersøgelse af bakteriepopulationsstruktur og dynamik i traditionel koumiss fra Indre Mongoliet ved hjælp af enkeltmolekyle realtidsekventering. J. Mejeri Sci. 99, 7852–7863. doi: 10.3168 / jds.2016-11167
PubMed abstrakt / CrossRef Fuld tekst / Google Scholar
Guarcello, R., Carpino, S., Gaglio, R., Pino, A., Rapisarda, T., Caggia, C., Et Al. (2016). En stor fabrik-skala anvendelse af udvalgte autoktone mælkesyrebakterier til produktion af PDO Pecorino Siciliano ost. Fødevarer Mikrobiol. 59, 66–75. doi: 10.1016 / j.fm. 2016.05.011
PubMed abstrakt / CrossRef Fuld tekst / Google Scholar
Hao, Y., Y., L., Jang, H., Jai, Y., Huang, Y., Liu, et al. (2010). Identifikation af den bakterielle biodiversitet i koumiss ved denaturering af gradientgelelektroforese og artsspecifik polymerasekædereaktion. J. Mejeri Sci. 93, 1926–1933. doi: 10.3168 / jds.2009-2822
PubMed abstrakt / CrossRef fuldtekst / Google Scholar
Helinck, S., Le Bars, D., Moreau, D. og Yvon, M. (2004). Evne af termofile mælkesyrebakterier til at producere aromaforbindelser fra aminosyrer. Appl. Environ. Mikrobiol. 70, 3855–3861. doi: 10.1128 / AEM.70.7.3855-3861.2004
PubMed abstrakt / CrossRef fuldtekst / Google Scholar
Hoskins, L. C., Agustines, M., McKee, V. B., Boulding, E. T., Kriaris, M. Og Niedermeyer, G. (1985). Mucinnedbrydning i humane kolonøkosystemer. Isolering og egenskaber af fækale stammer, der nedbryder ABH blod gruppe antigener og oligosaccharider fra mucin glycoproteiner. J. Clin. Investere. 75, 944–953. doi: 10.1172 / JCI111795
PubMed abstrakt / CrossRef Fuld tekst / Google Scholar
Irmler, S., Raboud, S., Beisert, B., Rauhut, D. og Berthoud, H. (2008). Kloning og karakterisering af to Lactobacillus casei-gener, der koder for en cystathioninlyase. Appl. Environ. Mikrobiol. 74, 99–106. doi: 10.1128 / AEM.00745-07
PubMed Abstrakt / CrossRef Fuld Tekst / Google Scholar
Jagielski, V. (1877). Værdien af koumiss i behandlingen af kvalme, opkastning og manglende evne til at bevare anden mad på maven. Br. Middelhavs. J. 2, 919-921. doi: 10.1136 / bmj.2.887.919
PubMed abstrakt / CrossRef Fuld tekst / Google Scholar
Kato, S., Ishihara, T., Hemmi, H., Kobayashi, H. og Yoshimura, T. (2011). Ændringer i d-aminosyrekoncentrationer og mikrobielle samfundsstrukturer under fermentering af røde og hvide vine. J. Biosci. Bioeng. 111, 104–108. doi: 10.1016 / j. jbiosc.2010.08.019
PubMed abstrakt / CrossRef Fuld tekst / Google Scholar
Li, H. og Durbin, R. (2009). Hurtig og præcis kortlæsningsjustering med huller-Hjultransformation. Bioinformatik 25, 1754-1760. doi: 10.1093 / bioinformatik / btp324
PubMed abstrakt / CrossRef Fuld tekst / Google Scholar
(2006). Cd-hit: et hurtigt program til klyngedannelse og sammenligning af store sæt protein-eller nukleotidsekvenser. Bioinformatik 22, 1658-1659. doi: 10.1093 / bioinformatik / btl158
PubMed abstrakt / CrossRef Fuld tekst / Google Scholar
Miyamoto, M., Seto, T., Nakajima, H., Burenjargal, S., Gombojav, A., Demberei, S., et al. (2010). Denaturering gradient gel elektroforese analyse af mælkesyrebakterier og gær i traditionel Mongolsk fermenteret mælk. Mad Sci. Technol. Res. 16, 319-326. doi: 10.3136 / fstr.16.319
CrossRef Fuld tekst | Google Scholar
Mulder, N. J. og J., R. (2008). InterPro database og værktøjer til protein domæne analyse. Curr. Protoco. Bioinformatik Kapitel 2, Unit2. 7. doi: 10.1002/0471250953. bi0207s21
PubMed abstrakt / CrossRef Fuld tekst / Google Scholar
Noguchi, H., Park, J. og Takagi, T. (2006). MetaGene: prokaryotisk genfinding fra miljøgenom haglgeværsekvenser. Nukleinsyrer Res. 34, 5623-5630. doi: 10.1093 / nar / gkl723
PubMed abstrakt / CrossRef Fuld tekst / Google Scholar
Nurk, S., Bankevich, A., Antipov, D., Gurevich, A. A., Korobeynikov, A., Lapidus, A., et al. (2013). Samling af encellede genomer og mini-metagenomer fra kimære MDA-produkter. J. Comput. Biol. 20, 714–737. doi: 10.1089 / cmb.2013.0084
PubMed abstrakt / CrossRef Fuld tekst / Google Scholar
Ott, A., Germond, J. E. Og Chaintreau, A. (2000). Oprindelse af acetaldehyd under mælkefermentering ved anvendelse af (13) C-mærkede prækursorer. J. Agric. Mad Chem. 48, 1512–1517. doi: 10.1021 / jf9904867
PubMed abstrakt / CrossRef Fuld tekst / Google Scholar
Yan, Y. T. (2011). Karakterisering af Lactobacillus fermentum SM – 7 isoleret fra koumiss, en potentiel probiotisk bakterie med kolesterolsænkende virkninger. J. Sci. Fødevare. Agric. 91, 512–518. doi: 10.1002 / jsfa.4214
PubMed Abstrakt / CrossRef Fuld Tekst / Google Scholar
Papadimitriou, K., Anastasiou, R., Maistrou, E., Plakas, T., Papandreou, N. C., Hamodrakas, S. J., et al. (2015). Erhvervelse gennem vandret genoverførsel af plasmid pSMA198 af Streptococcus macedonicus ACA-DC 198 peger mod artens mejerioprindelse. PLoS en 10: e0116337. doi: 10.1371 / tidsskrift.pone.0116337
PubMed abstrakt / CrossRef Fuld tekst / Google Scholar
han er en af de mest kendte i verden, og han er en af de bedste i verden (2012). Metagenomisk mikrobiel samfundsprofilering ved hjælp af unikke kladespecifikke markørgener. Nat. Metoder 9, 811-814. doi: 10.1038 / nmeth.2066
PubMed abstrakt / CrossRef Fuld tekst / Google Scholar
J., Yu, J., J., Cai, C., Et Al. (2010). Identifikation og karakterisering af de dominerende lactobaciller isoleret fra koumiss i Kina. J. Gen. Appl. Mikrobiol. 56, 257–265. doi: 10.2323 / jgam.56.257
PubMed Abstrakt / CrossRef Fuld Tekst / Google Scholar
Thompson, J. (1879). Værdien af koumiss i spild af sygdomme. Br. Middelhavs. J. 1, 270. doi: 10.1136 / bmj.1.1466.270-c
PubMed abstrakt / CrossRef Fuld tekst / Google Scholar
Vincent, S. J., Faber, E. J., Neeser, J. R., Stingele, F. og Kamerling, J. P. (2001). Struktur og egenskaber af eksopolysaccharidet produceret af Streptococcus macedonicus Sc136. Glykobiologi 11, 131-139. doi: 10.1093 / glycob / 11.2.131
PubMed abstrakt / CrossRef Fuld tekst / Google Scholar
C. T., Sorich, M. J., Gerber, J. P., Angley, M. T. og Conlon, M. A. (2013). Øget overflod af Sutterella spp. og Ruminococcus momenter i afføring af børn med autismespektrumforstyrrelse. Mol. Autisme 4, 42. doi: 10.1186/2040-2392-4-42
PubMed abstrakt / CrossRef Fuld tekst / Google Scholar
afdeling, T. L., Hosid, S., Ioshikhes, I. og Altosaar, I. (2013). Human mælk metagenome: en funktionel kapacitetsanalyse. BMC Microbiol. 13:116. doi: 10.1186/1471-2180-13-116
PubMed abstrakt / CrossRef Fuld tekst / Google Scholar
K., Fujimoto, J., Tomii, Y., Sasamoto, M., Makino, H., Kudo, Y., et al. (2009). Lactobacillus kisonensis sp. nov., Lactobacillus otakiensis sp. nov., Lactobacillus rapi sp. nov. og Lactobacillus sunkii sp. nov., heterofermentative arter isoleret fra sunki, en traditionel japansk pickle. Int. J. Syst. Evol. Mikrobiol. 59 (Pt 4), 754-760. doi: 10.1099 / ijs.0.004689-0
PubMed abstrakt / CrossRef Fuld tekst / Google Scholar
J., Li, H., Menghe, B., Vu, J., et al. (2009). Isolering og foreløbig probiotisk udvælgelse af lactobaciller fra koumiss i Indre Mongoliet. J. Grundlæggende Mikrobiol. 49, 318–326. doi: 10.1002 / jobm.200800047
PubMed abstrakt / CrossRef Fuld tekst / Google Scholar
D. R. Og Birney, E. (2008). Velvet: algoritmer til de novo kortlæst samling ved hjælp af de Bruijn grafer. Genom Res. 18, 821-829. doi: 10.1101 / gr.074492.107
PubMed abstrakt / CrossRef Fuld tekst / Google Scholar
H., H., H., H., H., H. (2010a). PCR-screening og sekvensanalyse af IOL-klynger i Lactobacillus casei-stammer isoleret fra koumiss. Folia Microbiol. (Praha) 55, 603-606. doi: 10.1007 / s12223-010-0097-3
PubMed abstrakt / CrossRef Fuld tekst / Google Scholar
det er en af de mest almindelige måder at gøre det på. (2010b). Komplet genomsekvens af Lactobacillus casei Jang, en ny probiotisk stamme isoleret fra traditionel hjemmelavet koumiss i Indre Mongoliet, Kina. J. Bakteriol. 192, 5268–5269. doi: 10.1128 / JB.00802-10
PubMed abstrakt / CrossRef Fuld tekst / Google Scholar
Jørgen, J. Og Jørgen, J. (2011). “Fermentering og koumiss,” i Handbook of Food and Beverage fermenteringsteknologi, 2.udgave, Red. Y. H. Hui (Boca Raton, FL: CRC Press), 165-172.
Google Scholar
H. P. (2015). Genekspression af proteolytisk system af Lactobacillus helveticus H9 under mælkefermentering. Ann. Mikrobiol. 65, 1171–1175. doi: 10.3168 / jds.2014-8520
PubMed abstrakt / CrossRef Fuld tekst / Google Scholar
J., J., J., S., T., Et Al. (2011). Komplet genomsekvens af Lactobacillus helveticus H10. J. Bakteriol. 193, 2666–2667. doi: 10.1128 / JB.00166-11
PubMed Abstrakt / CrossRef Fuld Tekst / Google Scholar