- Introduction
- materiaalit ja menetelmät
- näytteiden valmistelu
- Gradienttilaimennus ja Monisiirtymävahvistus
- kirjaston rakenne ja sekvensointi
- Data-analyysi
- sekvenssin laadun tarkistus ja suodatus
- taksonominen jako ja monimuotoisuus
- Read Assembly, Gene Prediction, and Annotation
- Nukleotidisekvenssit Liittymisnumerot
- tulokset
- kokeellinen suunnittelu ja sekvensointi
- taksonominen Huomautus
- Metagenominen kokoonpano, geenien ennustaminen ja funktionaalinen Huomautus
- Keskustelu
- johtopäätös
- Tekijäosuudet
- Eturistiriitalausunto
- tunnustus
- lisäaineisto
Introduction
Koumiss, toiselta nimeltään chige, chigo, arrag, or airag, in the mongolin language, is a type of traditional fermented dairy product. Se on ollut suosittu ruoka Mongoliassa ja Kiinan Sisä-Mongoliassa vuosisatojen ajan (Zhang and Zhang, 2011). Näillä alueilla ihmisillä oli tapana kuluttaa koumissia suurten juhlallisuuksien ja uhrilahjojen aikana (Zhang and Zhang, 2011). Varhaisin merkintä koumissin tuotannosta voidaan jäljittää Han-dynastian aikaan (BC202-AD202). Tämä tuote oli saavuttanut laajaa suosiota Yuan-dynastian aikana (AD1271-AD1368) (Zhang et al., 2010b). Nykyään koumissi on Mongolian ja Sisä-Mongolian paikallisväestön yhteinen ruoka, joskin vain harvoilla näistä alueista sitä valmistetaan teollisessa mittakaavassa. Komiss ei ainoastaan tarjoa runsaasti ravintoaineita, kuten suuria välttämättömiä aminohappoja ja vitamiineja, vaan sen uskotaan myös helpottavan monenlaisia sairauksia ja olevan hyödyllinen leikkauksen jälkeisessä hoidossa (Jagielski, 1877; Thompson, 1879).
perinteisesti koumissia valmistetaan yleisesti puutynnyreissä, eläinten nahasta tehdyissä astioissa tai uurnissa. Käyminen tapahtuu luonnollisesti ympäristön lämpötilassa sen jälkeen, kun astiaan on lisätty suodatettua tammamaitoa vanhalla koumissilla, joka toimii aloitusviljelynä (Zhang and Zhang, 2011). Komiss on hyvä bioteknisesti potentiaalisten uusien bakteerien lähde (Zhang et al., 2010a; Pan et al., 2011). Siksi on erittäin kiinnostavaa tutkia ja säilyttää mahdollisimman monta käymiseen liittyvää koumiss-bakteeria. Viime vuosikymmeninä koumissin bakteerikuntaa selvitettiin useilla tutkimuksilla (Wu et al., 2009; Hao et al., 2010), jota on tutkittu pääasiassa kulttuuri -, molekyylibiologia – ja pyrosekvenssipohjaisilla menetelmillä (Sun et al., 2010). Näistä erilaisista lähestymistavoista pyrosekvenssiin perustuva menetelmä on tarjonnut kattavimman koumissin mikrobiomiprofiilin riippumatta yksittäisten mikrobien fenotyyppisistä piirteistä ja viljelyongelmista. Komiss-bakteerien koodaamien funktionaalisten geenien kirjo ja niiden fermentointikyky ovat kuitenkin edelleen huonosti luonnehdittuja erityisesti harvinaisille mikrobipopulaatioille.
tässä tutkimuksessa käytettiin yksisoluista genomitekniikkaa analysoimaan 10 koumiss-näytteen bakteerimetagenomeja, jotka oli kerätty Kiinan Mongoliasta ja Sisä-Mongoliasta. Nykyisessä työssä on sovellettu uusinta teknologiaa maitotuotteiden bakteeriston monimuotoisuuden tutkimisessa. Työmme on osoittanut, että on mahdollista löytää vähän runsas taksonia soveltamalla yksisoluista metagenomiikan lähestymistapaa. Rohkaisevat tulokset edistäisivät uudenlaisten lähestymistapojen kehittämistä ja soveltamista ongelmien ratkaisemisessa perinteisellä tutkimusalalla.
materiaalit ja menetelmät
näytteiden valmistelu
Metagenomics-tutkimusta varten Mongoliasta (MG14, MG15, MG16, MG17 ja Mg18) ja Kiinan Sisä-Mongoliasta (Nm17, NM18, nm19, NM20 ja Nm21) kerättiin yhteensä 10 koumiss-näytettä. Näytteet kerättiin aseptisesti ja kuljetettiin kuivajäässä.
yksi millilitra kutakin näytettä esikäsiteltiin Ward et al-tutkimuksessa kuvatun menetelmän mukaisesti. (2013) muutamin muutoksin. Näytteitä sulatettiin hetken aikaa jääkylvyssä 3-5 minuutin ajan. Kun näytteet olivat sulaneet, niille tehtiin hidas sentrifugointi epäpuhtauksien ja eukaryoottisten solukompressioiden poistamiseksi. Tämän jälkeen prokaryoottisolut pelletoitiin maitoseerumista sentrifugoimalla 13 000 × g: n kokoon 15 minuutin ajan. Pelletit suspendoitiin uudelleen 2 mL: aan fosfaattipuskuroitua suolaliuosta (PBS), jossa oli 1% Triton X-100: aa, ja inkuboitiin 2 tunnin ajan 37°C: ssa jäljellä olevien eukaryoottisten solujen lyseeraamiseksi. Tämän jälkeen bakteereja pelletoitiin sentrifugoimalla 13 000 × g: n tarkkuudella 15 minuutin ajan ja pelletit suspendoitiin uudelleen 500 µL: aan PBS: ää. Lopuksi sentrifugointivaihe toistettiin vielä kerran bakteerisolujen pesemiseksi.
Gradienttilaimennus ja Monisiirtymävahvistus
vähävaraisten bakteerien havaitsemiseksi jokaisesta koumiss-näytteestä saatu bakteerisuspensio laimennettiin sarjamaisesti myöhempää monistusreaktiota varten. Kunkin näytteen solujen määrä arvioitiin karkeasti mikroskoopilla (Nikon, Tokio, Japani) käyttäen solujen laskentakammiota (Qiujing, Shanghai, Kiina). Laimennusvaihetta jatkettiin, kunnes kunkin bakteerisuspension solumäärä oli noin 100. Laimennettujen solujen monisiirtovahvistuksessa käytettiin REPLI-g: n Yksisolupakettia (Qiagen, Germantown, MD, USA) valmistajan ohjeiden mukaisesti.
kirjaston rakenne ja sekvensointi
monistettu DNA kerittiin sattumanvaraisesti, ja noin 500 bp: n pituiset fragmentit valittiin. Kirjaston rakentamisen jälkeen PerkinElmer LabChip® GX Touch-ja StepOnePlusTM-reaaliaikaista PCR-järjestelmää käytettiin kirjaston laadun tarkastamiseen. Lopulta 125 bp: n paripäätylukemat sekvensoitiin Illumina HiSeq 2500-alustalla valmistajan ohjeiden mukaan.
Data-analyysi
sekvenssin laadun tarkistus ja suodatus
sekvensserin tuottamat Raakalukemat saattavat sisältää keinotekoisia lukuja adapterin saastumisesta kirjaston rakentamisen aikana. Siksi suoritettiin kolme vaihetta korkealaatuisen puhtaan lukemisen aineiston saamiseksi: (1) adapterin saastumisen aiheuttamien lukemien poistaminen; (2) sellaisten lukemien poistaminen, joiden keskimääräinen pistemäärä on alle phred-arvon Q30, mitä pidettiin alimpana raja-arvona korkealaatuiselle perusarvolle; (3) sellaisten lukemien poistaminen, joiden merkittävä ylitys on ”N” (≥5% lukemasta). Jatkoanalyysi perustui puhtaisiin tietoihin. Lisäksi laskettiin tilastollinen peruslaatu, joka perustui Q30: een ja GC-pitoisuuteen.
isännän genomia vastaan tehtiin linjauksia isännästä peräisin olevien kontaminaatiosekvenssien poistamiseksi. Kaikki isännästä peräisin olevat lukemat hylättiin ennen kuin niitä verrattiin bakteerien (tai virusten) genomivertailusekvensseihin. Tarkempien tulosten saamiseksi alligmenteissa käytettiin Burrows-Wheeler aligner (BWA) (versio 0.97 a) mem-mallia (Li and Durbin, 2009).
taksonominen jako ja monimuotoisuus
web-ohjelmistoa Metaflania käytettiin taksonomiseen luokitteluun suku-ja lajitasoille (Segata et al., 2012). Vertailemme lajien monimuotoisuutta näytteiden sisällä ja välillä analysoimalla alfa – ja beeta-monimuotoisuutta R: ään liittyvällä paketilla.
Read Assembly, Gene Prediction, and Annotation
to get more comprehensive information, we Assembly The sheared fragments into genome (contigs). Koska metagenomisissa näytteissä esiintyy useita lajeja, paransimme bioinformatiikan genomin kokoamismenetelmää, jota tavallisesti käytetään yhden lajin analyysissä integroimalla pata (versio 3.6.2), sisäiset skriptit ja metagenomiset tietokannat (Zerbino and Birney, 2008; Nurk et al., 2013).
metagenemark-ohjelmistoa käytettiin koottujen kontigien (Noguchi et al., 2006). Redundantit geenit poistettiin CD-hitin avulla 90 ja 95% identiteetin kattavuudella (Li and Godzik, 2006; Fu et al., 2012). Geenien suhteelliset pitoisuudet määritettiin kohdistamalla laadukkaat sekvensointilukemat geeniluetteloon samaa menetelmää käyttäen. Loppupään eroavaisuusanalyysi perustui geenien suhteellisiin pitoisuuksiin. Geenimerkinnät tehtiin kohdistamalla laadukkaat sekvenssit useisiin julkisiin tietokantoihin (NCBI non-redundant database, NR; Ortologisten proteiiniryhmien klusterit, COGs; Kyoto Encyclopedia of Genes and Genomes, KEGG) käyttäen BLAST (Altschul et al., 1997). Verkkotunnuksen haku suoritettiin Interproscan (Mulder and Apweiler, 2008) avulla.
Nukleotidisekvenssit Liittymisnumerot
tässä tutkimuksessa raportoidut sekvenssitiedot on talletettu SRA-tietokantaan (Liittymisnumero.SRP083102).
tulokset
kokeellinen suunnittelu ja sekvensointi
koumissin vähävaraisten bakteerien havaitsemiseksi näytteiden metagenomien analysoinnissa käytettiin yksisoluista monistustekniikkaa. Kolme bakteerisuspensiota, kukin noin 100 solusta, johdettiin riippumattomasta koumiss-näytteestä sarjalaimennuksella. Laimennettuja suspensioita analysoitiin yhteensä 30. Kullekin laimennetulle näytteelle annettiin eri näytekoodi eli näytteen tunnusnumero, jota seurasi 1, 2 tai 3, joka edustaa kolmea erillistä laimennosta. Perustuen olettamukseen, että joitakin harvinaisia lajeja esiintyy yhdessä laimennoksista, solujen Siirtymä monistettiin; ja noin 5 Gb: n tiedot tuotettiin jokaisesta koumiss-bakteerisuspensiosta.
10 koumissin näytteestä (yhteensä 30 bakteerisuspensiota) saatiin yhteensä 1 040 323 864 raakalukemaa. Jokaisen 100-soluisen suspension lukema oli keskimäärin 34 677 462 (täydentävä taulukko S1). Kun trimmaus ja suodatus varauksettomat sekvenssit, saimme 1,018,381,702 puhdas lukee kaikille näytteille (täydentävä taulukko S1). Shannon-indeksin, Simpson-indeksin, Chao1-indeksin ja havaittujen lajien lukumäärän (Luvut 1-4) arvot osoittivat, että useimpien koumiss-näytteiden bakteeristo oli hyvin monimuotoinen. Shannonin-Wienerin diversiteettikäyrät osoittivat, että sekvenssin syvyys oli riittävä kaikille näytteille (Kuva 1).
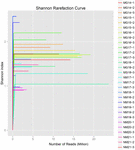
KUVA 1. Shannon rarefaction-käyrät, jotka arvioivat koumiss-näytteiden mikrobiston monimuotoisuutta.
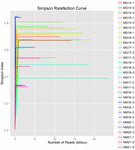
KUVA 2. Simpsonin harvinaistumiskäyrät, jotka arvioivat koumiss-näytteiden mikrobiston monimuotoisuutta.
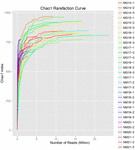
KUVA 3. Chao1 rarefaction-käyrät, jotka arvioivat koumiss-näytteiden mikrobiston monimuotoisuutta.
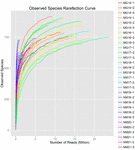
KUVA 4. Havaitut lajien harvinaistumisindeksit, jotka arvioivat koumiss-näytteiden mikrobiston monimuotoisuutta.
taksonominen Huomautus
korkealuokkaiset sekvenssit jaettiin eri taksonomisille tasoille, jotta näytteen bakteeriyhteisöt voitiin analysoida perusteellisesti. Viitaten joihinkin julkaistuihin tutkimuksiin koumissin biologisesta monimuotoisuudesta (Wu et al., 2009; Hao et al., 2010; Sun ym., 2010) luokittelimme tunnetut ja aiemmin raportoimattomat koumiss-bakteerit yleisiksi ja harvinaisiksi taksoneiksi.
kookkaat suvut edustivat 13 eri sukua (kuva 5). Kolmessa niistä oli keskimäärin yli 1%: n suhteellinen määrä, mukaan lukien Lactobacillus (L.), Lactococcus ja Streptococcus. Erityisesti Lactobacillus ja Lactococcus olivat kaksi runsainta koumissukua. Lactobacillus-bakteerin osuus näytteistä vaihteli välillä 52,72-99,96%. Kaksi tämän suvun jäsentä, L. helveticus ja L. kefiranofaciens, hallitsivat useimpia koumiss-näytteitä (kuva 6). Laji L. buchneria esiintyi lähinnä Mongolian näytteissä, kun taas Lactococcus lactis-bakteeria havaittiin useimmissa näytteissä näytteenottoalueesta riippumatta.
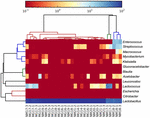
KUVA 5. Heatmap osoittaa koumiss-näytteissä Havaittujen bakteerien suhteellisen runsauden sukutasolla.
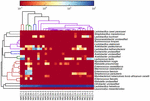
KUVA 6. Heatmap osoittaa koumiss-näytteissä Havaittujen bakteerien suhteellisen runsauden lajitasolla.
Metagenominen kokoonpano, geenien ennustaminen ja funktionaalinen Huomautus
lukujen kokoaminen johti 614 392 623 bp: n pituuteen. Kokoonpanojen N50-arvot vaihtelivat välillä 5,596-35,200 bp (täydentävä taulukko S2). Ennustettujen geenien määrä koumiss-näytteissä vaihteli välillä 10 347-34 547, ja keskimääräinen pituus vaihteli välillä 647,82-985,33 bp (täydentävä taulukko S3). Vaikka empiiriset geenifunktioanalyysit eivät kuuluneet nykyisen tutkimuksen piiriin, yhdistimme koumiss-bakteerin mikrobiomit COG-ja KEGG-tietokantojen avulla, jotka ennustivat geenien toimintaa pitkälti sekvenssihomologian perusteella.
yhteensä 545 osumaa annotointilähdössä osoitti laktoosin käyttögeenien korkean homologian (COGs-Luokka hiilihydraatti ja aineenvaihdunta, G), ja osa niistä sijaitsi samassa kontigissa (Taulukko 1).
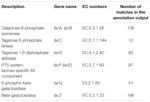
TAULUKKO 1. Koumiss-bakteerin metagenomiin merkityt laktoosi-aineenvaihduntaan liittyvät geenit.
eräät muut sekvenssit saattavat koodata aminohappojen kuljetuksen ja metabolian (E) COGS-luokkaan kuuluvia oletettuja geenejä, mukaan lukien sekvenssit, jotka vastasivat kaseiinia hajottavia proteinaaseja, 4-18 jäämän oligopeptidejä ja aminopeptidaaseja (esim. leusyyliaminopeptidaasi, peptidyylidipeptidaasi A, aminopeptidaasi N, proliini-iminopeptidaasi, ja endopeptidaasi). Vaikka joillakin sekvensseillä oli suuria yhtäläisyyksiä arginiinille, aspartaatille, metioniinille ja isoleusiinille spesifisten aminotransferaasien kanssa, havaittiin vain yksi merkittävä vastaavuus, joka vastasi tyrosiinille ja fenyylialaniinille spesifistä S. macedonicuksesta peräisin olevaa luokan I/II domeenia (IPR004839) sisältävää aminotransferaasia. Lopuksi useat sekvenssit jakoivat korkean homologian aminohappolyaaseille, mukaan lukien s-ribosyylihomokysteiinilyaasi, argininosukkinaattilyaasi, aspartaatti ammoniakkilyaasi, kystationiini gammalyaasi, histidiini ammoniakkilyaasi ja O-asetyylihomoseriini (tioli)-lyaasi.
Keskustelu
Koumiss on suosittu perinteinen fermentoitu maitotuote Mongoliassa ja Kiinan Sisä-Mongoliassa. Koumissin bakteeriston monimuotoisuutta on tutkittu useissa tutkimuksissa, mutta koumissin mikrobiston geneettisestä kapasiteetista on saatu vain vähän tietoa. Tässä sovellimme yksisoluista monistustekniikkaa analysoidaksemme koumiss-bakteerin metagenomeja, keskittyen erityisesti vähän runsaaseen bakteerikantaan.
tyypillistä metagenomiikkaa on aiemmin sovellettu koumissin mikrobiston kuvaamiseen. Syvän sekvenssin saavuttamisen kalleuden vuoksi näytteiden harvinainen mikrobipopulaatio on kuitenkin usein peitetty puutteellisesti. Vähemmistöbakteerien sekä fylogeeniset että toiminnalliset metagenomit jäävät siis vähäisiksi. Yksisoluisella vahvistusmenetelmällämme näytteet laimennettiin 100-soluisiksi suspensioiksi. Koska laimennetuissa koumiss-näytteissä on vähän soluja, vain rajallinen määrä DNA: ta saataisiin talteen. Monistusvaihe lisäsi analysoitavien DNA-materiaalien määrää ja helpotti siten metagenomin analysointia näytteistä, jotka sisälsivät pieniä DNA-määriä. Yksi haittapuoli tässä menetelmässä oli vaikeus varmistaa, että sekvenssit kunkin taksonin olisi yhtä monistetaan prosessissa; tästä syystä saadut tulokset saattoivat kuvastaa vain sekvenssien suhteellista runsautta, mutta eivät tunnistettujen taksonien tai funktionaalisten geenien absoluuttisia määriä. Tällä ei kuitenkaan olisi suurta vaikutusta analyysiimme, sillä nyt julkaistu tutkimus eroaa muista julkaistuista teoksista harvinaisen bakteerikannan tarkentamisessa. Tämän työn tuottamat tiedot antavat täydentävää tietoa aliedustetulle väestölle. Uskomme, että nykyinen lähestymistapa soveltuu tulevaisuudessa erilaisten ekologisten ympäristöjen bakteerien monimuotoisuuden analysointiin.
koumiss-bakteerin mikrobisto koostui pääasiassa maitohappobakteereista (LAB) ja joistakin etikkahappobakteereista (Zhang and Zhang, 2011). Odotetusti aineistossamme oli lähinnä sekvenssejä, jotka edustivat laboratoriota ja etikkahappobakteereja. Lactobacillus helveticusta vastaavat sekvenssit dominoivat kaikissa näytteissä. Lisäksi, sekvenssit edustavat lajia, L. kefiranofaciens. L. buchneri. L. kefiranofaciens. Enterococcus (E.) casseliflavus. E. faecalis. E. faecium. Leuconostoc mesenteroides. Myös Lactococcus lactis ja Acetobacter pasteurianus löydettiin. Eri taksonien sekvenssien tunnistaminen ei välttämättä riitä osoittamaan bakteerien elinkelpoisuutta, mutta se kuitenkin heijastaa bakteeriyhteisöä jossakin käymisprosessin vaiheessa. Miyamoto ym. (2010) viittaa siihen, että bakteerien esiintyvyys lopullisissa fermentoiduissa tuotteissa liittyy niiden happamaan stressinsietokykyyn. Laktobasilleilla on yleensä suurempi haponsietokyky kuin laktokokilla, mikä saattaa selittää laktobasillisekvenssien suuren suhteellisen runsauden aineistossamme. Toisaalta usein esiintyvä L. helveticus koumississa tapahtui samaan aikaan havainto L. helveticus-sekvenssien valta-asemasta.
aineistossamme eräät sekvenssit vastasivat L. otakiensista, joka on harvinainen laji, jota ei ole koskaan raportoitu koumississa tai muissa maitiaisnesteissä. Laji kuvattiin ensin ja eristettiin suolaamattomasta peittausliuoksesta, jota käytettiin perinteisen japanilaisen suolakurkun (Watanabe et al., 2009). Se löydettiin amplified fragmentti pituus polymorfismi profilointi perustuu recA geeni. Tämän jälkeen lajin ei ole raportoitu liittyvän muihin ravintoon liittyviin lokeroihin. On siis todennäköistä, että tämä bakteeri kuuluu suolakurkkujen omakustanteiseen kasvistoon. Emme kuitenkaan voi sulkea pois sitä mahdollisuutta, että sitä ei löydetty pelkästään käytetyn havaitsemismenetelmän herkkyyden vuoksi. Lactobacillus otakiensis voi tuottaa D-haaraketjuisia aminohappoja, kuten d-leusiinia, d-allo-isoleusiinia ja d-valiinia (Doi et al., 2013). Sitä voidaan käyttää tiettyjen fermentoitujen elintarvikkeiden (Kato et al., 2011).
tutkimuksessamme tunnistettiin myös sekvenssejä, jotka edustivat lajia S. macedonicus, josta ei ole koskaan raportoitu koumiss-bakteeria. Sen sijaan se on kreikkalaisissa lammas-ja vuohenjuustoissa (Georgalaki et al., 2000). Jotkut tämän lajin jäsenet pystyvät tuottamaan eksopolysakkarideja, bakteriosiineja (Vincent et al., 2001; Anastasiou et al., 2015) ja gamma-aminovoihappo (Franciosi et al., 2015). Vaikka tämä laji on usein eristetty fermentoiduista elintarvikkeista, S. macedonicus-lajin alkuperäinen markkinarako on ollut kiistanalainen (Guarcello et al., 2016). Vasta äskettäin, Papadimitriou et al. (2015) tunnistettu hankittu plasmidi, pSMA198, Lactococcus lactis. Plasmidi siirrettiin todennäköisesti esi-isien geneettisen vaihtotapahtuman kautta maitotuoteympäristössä, mikä viittaa S. macedonicuksen (Papadimitriou et al., 2015). Samanlainen kuin S. thermophilus. S. macedonicus on läheistä sukua S. bovis/S. equinus-kompleksin kommensaalisille ja opportunistisille taudinaiheuttajille.
Ruminococcus torques-lajia edustavien sekvenssien tunnistaminen oli odottamatonta, sillä kyseistä bakteeria tavataan yleensä suolistoympäristössä. Se on normaali ihmisen suolistomikrobi, joka voi hajottaa mucin oligosakkarideja sekretoristen glykosidaasien konstitutiivisen tuotannon (Hoskins et al., 1985). Viimeaikaiset kliiniset todisteet osoittavat, että ulosteen runsaus tämän lajin muuttuu lapsilla autismikirjon häiriö; kuitenkin, sen rooli häiriö jää epäselväksi (Wang et al., 2013).
yllättäen osa sekvensseistä edusti potentiaalisia taudinaiheuttajia. Esimerkiksi Klebsiella keuhkokuume on opportunistinen ihmisen patogeeni, joka asuu noin 40% ihmisen ja eläimen suolet. Mycobacterium orygis, aiemmin nimeltään oryx bacillus, kuuluu Mycobacterium tuberculosis-ryhmään, joka voi aiheuttaa ihmisen tuberkuloosia (Dawson et al., 2012). Näitä kahta lajia on raportoitu raakamaidosta, mutta ei koumissista. Sen vuoksi niiden esiintyminen voisi johtua kontaminaatiosta tavanomaisessa komiss-tuotannossa, erityisesti ei-tai vähäaseptisissa käsittelyolosuhteissa.
bakteerien aineenvaihdunnalla on tärkeä rooli koumissin ominaisuuksien ja laadun muodostamisessa. Komissin aromaattisten ja aromiyhdisteiden syntetisointiin tarvitaan mikrobipohjaisia prosesseja, kuten lipolyysiä ja proteolyysiä (Gesudu et al., 2016). Bakteerin metagenomi sisälsi johdonmukaisesti sekvenssejä, jotka mahdollisesti koodaavat laktoosin hajoamista ja proteolyyttisiä järjestelmiä. Toisin kuin suhteellisen yksinkertainen laktoosi catabolic reitit, LAB proteolyyttiset järjestelmät koostuvat monipuolinen joukko entsyymejä (Chen et al., 2014). Verrattuna muihin havaittuihin koumiss-laboratorioihin, dominoivalle lajille L. helveticus on ominaista korkea proteolyyttinen aktiivisuus (Zhang et al., 2015). Useimmissa laboratorioissa on vain yksi solukuoriproteinaasi, joka käynnistää maidon kaseiinihydrolyysin, kun taas L. helveticus sisältää vähintään kaksi näistä entsyymeistä, prth: n ja PrtH2: n (Zhao et al., 2011). Näin ollen L. helveticuksen vahvaa proteolyyttistä lajia vastaavien sekvenssien ja proteolyysiin liittyvien geenien suuret osuudet voivat liittyä koumissin suhteellisen korkeaan peptidien ja vapaiden aminohappojen pitoisuuteen.
teollisen mittakaavan koumissin valmistuksen yksi vaikeus on makuhavainnon hallinta, sillä koumiss on perinteisesti valmistettu luonnollisella käymisellä. Siksi on ollut vaikea määritellä maku ja Profiilin tärkeimmät makukomponentit, varsinkin kun läsnä on luonnon epäpuhtauksia. Keskeisten makuaineiden tuotanto on seurausta aminohappojen, kuten haaraketjuisten aminohappojen, metioniinin ja aromaattisten aminohappojen, fermentatiivisesta ja entsymaattisesta hajoamisesta (Ardo, 2006). Esimerkkejä makukomponenteista ovat aldehydit, orgaaniset hapot ja esterit, joita muodostuu transaminaasi (AT)-reitillä (Helinck et al., 2004). Tämän reitin aloittaa transaminaasi, joka katalysoi aminohapon muuntumista vastaavaksi α-ketohapoksi (Helinck et al., 2004). Jotkin sekvenssit aineistossamme vastasivat oletettuja haaraketjuisia ja aromaattisia aminohappoaminotransferaaseja. Erityisesti koumissin sukulaislajin S. macedonicuksen kontigista löytyi putatiivista aromaattista aminohappoa aminotransferaasi I: tä. Koska nykyisessä työssämme vain huomautettiin mikrobiomista silicossa, tietomme eivät riitä osoittamaan, että nämä tunnistetut geenisekvenssit olisivat todella toimivia tuottamaan edellä mainittuja komiss-makuyhdisteitä. Näiden geenien läsnäolo kuitenkin viittaa siihen, että ne ovat mahdollisia ehdokkaita tällaiseen fermentatiiviseen toimintaan; kuitenkin tarvitaan lisää kokeellista työtä niiden täsmällisten toiminnallisten roolien selventämiseksi.
lisäksi löysimme sekvenssejä, jotka vastaavat muita aminohappojen muuntumisreittejä, mukaan lukien aminohappolyaasit ja treoniinialdolaasi. Entinen entsyymi katalysoi metioniinin muuntumista metaanitioliksi (Irmler et al., 2008), jolloin muodostuu dimetyylidisulfidia ja dimetyylitrisulfidia (Fernandez et al., 2000), kun taas jälkimmäinen katalysoi treoniinin muuntumista glysiiniksi ja asetaldehydiksi (Ott et al., 2000). Vastaavasti pelkästään näiden sekvenssien paikantaminen koumissin mikrobiomiin ei voi olla varmaa näyttöä niiden varsinaisista biologisista rooleista, vaan tarvitaan tulevaisuudessa kokeellinen vahvistus.
johtopäätös
tässä tutkimuksessa käytettiin muokattua metagenomiikan menetelmää, jolla analysoitiin koumissin näytteiden mikrobiomia, joka oli kerätty Mongoliasta ja Kiinan Sisä-Mongoliasta. Olemme luonnehtineet koumissin harvinaisten lajien fylogeenisiä ja toiminnallisia metagenomeja; ja aineistomme heijastaa piirteitä, jotka ovat bioteknisesti kiinnostavia ja potentiaalisia. Tutkimuksemme on osoittanut ensimmäistä kertaa, että on mahdollista käyttää yksisoluista monistustekniikkaa koumiss-bakteerin mikrobiomin sekä mikrobikontaminanttien havaitsemiseen. Tässä kehitettyjä tekniikoita voidaan käyttää tulevissa tutkimuksissa koumissin mikrobiomin muutosten seuraamiseen käymisprosessin varrella keskittyen vähemmistömikrobipopulaatioon. Lisäksi muut omics-tekniikat, kuten transkriptomiikka, metabolomiikka, voidaan yhdistää nykyiseen metagenomiikan analyysiin koumissin mikrobiomin toimintojen ja metabolisen kapasiteetin vahvistamiseksi.
teknisesti seuraava askel tässä työssä olisi nykyisen menetelmän optimointi. Esimerkiksi lisäämällä näytelaimennosta ennen DNA: n vahvistusta voidaan parantaa mahdollisuutta löytää harvinaisia ja uusia bakteereja. Toisaalta, vaihtoehtoinen sekvensointitekniikka, joka voi tuottaa pitkiä lukemia, kuten Pacific Biosciences single molecule, reaaliaikainen sekvensointitekniikka, voidaan käyttää parantamaan genomin kokoamisprosessia.
Tekijäosuudet
WZ ja HZ suunnittelivat tutkimuksen. WZ, GY, JY ja L-YK kirjoittivat käsikirjoituksen. QH, WH, WL, BM ja TS suorittivat kokeita. WZ ja QH analysoivat dataa. Kaikki kirjoittajat kävivät käsikirjoituksen läpi.
Eturistiriitalausunto
kirjoittajat toteavat, että tutkimus tehtiin ilman kaupallisia tai taloudellisia suhteita, joita voitaisiin pitää mahdollisena eturistiriitana.
tunnustus
tätä tutkimusta tuki Kiinan kansallinen Luonnontieteellinen säätiö (apuraha nro 31571815).
lisäaineisto
tämän artikkelin lisäaineisto löytyy verkosta osoitteesta: https://www.frontiersin.org/article/10.3389/fmicb.2017.00165/full#supplementary-material
Altschul, S. F., Madden, T. L., Schaffer, A. A., Zhang, J., Zhang, Z., Miller, W., et al. (1997). Gapped BLAST ja PSI-BLAST: uuden sukupolven proteiinitietokannan hakuohjelmat. Nucleic Acids Res. 25, 3389-3402. doi: 10.1093/nar/25.17.3389
PubMed Abstract / CrossRef Full Text / Google Scholar
Anastasiou, R., Driessche, G. V., Boutou, E., Kazou, M., Alexandraki, V., Vorgias, C. E., et al. (2015). Streptococcus macedonicus-bakteerin muuntuneet kannat, joilla on osmoottinen stressiresistentti fenotyyppi, säilyttävät kykynsä tuottaa bakteriosiini makedosiinia hyperosmoottisissa olosuhteissa. J. Biotechnol. 212, 125–133. doi: 10.1016 / j.jbiotec.2015.08.018
PubMed Abstract | CrossRef Full Text | Google Scholar
Ardo, Y. (2006). Arominmuodostus aminohappokatabolian avulla. Bioteknologiaa. ADV 24, 238-242. doi: 10.1016 / J.biotechadv.2005.11.005
PubMed Abstract | CrossRef Full Text | Google Scholar
Chen, Y. F., Zhao, W. J., Wu, R. N., Sun, Z. H., Zhang, W. Y., Wang, J. C., et al. (2014). Lactobacillus helveticus H9: n Proteome-analyysi rasvattoman maidon kasvun aikana. J. Dairy Sci. 97, 7413–7425. doi: 10.3168 / jds.2014-8520
PubMed Abstract | CrossRef Full Text | Google Scholar
Dawson, K. L., Bell, A., Kawakami, R. P., Coley, K., Yates, G., and Collins, D. M. (2012). Mycobacterium orygis (M. tuberculosis complex species) – bakteerin tarttuminen tuberkuloosipotilaalta Lypsylehmään Uudessa-Seelannissa. J. Clin. Mikrobiolia. 50, 3136–3138. doi: 10.1128 / JCM.01652-12
PubMed Abstract | CrossRef Full Text | Google Scholar
Doi, K., Mori, K., Mutaguchi, Y., Tashiro, K., Fujino, Y., Ohmori, T., et al. (2013). D-haaraketjuisten aminohappojen tuottajan Lactobacillus otakiensis JCM 15040T genomisekvenssin luonnos, eristetty perinteisestä japanilaisesta suolakurkusta. Genomikuulutus. 1, e546-e513. doi: 10.1128 / genomeA.00546-13
PubMed Abstract | CrossRef Full Text | Google Scholar
Fernandez, M., van Doesburg, W., Rutten, G. A., Marugg, J. D., Alting, A. C., van Kranenburg, R., et al. (2000). Kystationiini-beetalyaasia koodaavan Lactococcus lactisin metC-geenin molekyyli-ja funktionaaliset analyysit. Appl. Environ. Mikrobiolia. 66, 42–48. doi: 10.1128 / AEM.66.1.42-48.2000
PubMed Abstract | CrossRef Full Text | Google Scholar
Franciosi, E., Carafa, I., Nardin, T., Schiavon, S., Poznanski, E., Cavazza, A., et al. (2015). Biodiversiteetti ja gamma-aminovoihapon tuotanto maitohappobakteereilla, jotka on eristetty perinteisistä alpiinisista raa ’ asta lehmänmaidosta valmistetuista juustoista. Biomed Res. Int. 2015, 625740. doi: 10.1155/2015/625740
PubMed Abstrakti / CrossRef kokoteksti / Google Scholar
Fu, L., Niu, B., Zhu, Z., Wu, S., and Li, W. (2012). CD-HIT: kiihdytetty seuraavan sukupolven sekvensointitietojen ryhmittelyyn. Bioinformatics 28, 3150–3152. doi: 10.1093/bioinformatics/bts565
PubMed Abstract | CrossRef Full Text | Google Scholar
Georgalaki, M. D., Sarantinopoulos, P., Ferreira, E. S., De Vuyst, L., Kalantzopoulos, G., and Tsakalidou, E. (2000). Biochemical properties of Streptococcus macedonicus strains isolated from Greek Kasseri cheese. J. Appl. Microbiol. 88, 817–825. doi: 10.1046/j.1365-2672.2000.01055.x
PubMed Abstract | CrossRef Full Text | Google Scholar
Gesudu, Q. M., Zheng, Y., Xi, X. X., Hou, Q. C., Xie, H. Y., Huang, W. Q., et al. (2016). Tutkitaan bakteeripopulaation rakennetta ja dynamiikkaa perinteisessä Koumississa Sisä-Mongoliasta käyttäen yhden molekyylin reaaliaikaista sekvensointia. J. Dairy Sci. 99, 7852–7863. doi: 10.3168 / jds.2016-11167
PubMed Abstract | CrossRef Full Text | Google Scholar
Guarcello, R., Carpino, S., Gaglio, R., Pino, A., Rapisarda, T., Caggia, C., et al. (2016). Valikoitujen kotoperäisten maitohappobakteerien laajamittainen käyttö San Pecorino Siciliano-juuston valmistuksessa. Elintarvikemikrobioli. 59, 66–75. doi: 10.1016 / J.fm. 2016.05.011
PubMed Abstract | CrossRef Full Text | Google Scholar
Hao, Y., Zhao, L., Zhang, H., Zhai, Z., Huang, Y., Liu, X., et al. (2010). Bakteerien biodiversiteetin määrittäminen koumississa denaturoimalla gradienttigeelielektroforeesi ja lajispesifinen polymeraasiketjureaktio. J. Dairy Sci. 93, 1926–1933. doi: 10.3168 / jds.2009-2822
PubMed Abstract | CrossRef Full Text | Google Scholar
Helinck, S., Le Bars, D., Moreau, D., and Yvon, M. (2004). Termofiilisten maitohappobakteerien kyky tuottaa aromiyhdisteitä aminohapoista. Appl. Environ. Mikrobiolia. 70, 3855–3861. doi: 10.1128 / AEM.70.7.3855-3861.2004
PubMed Abstract | CrossRef Full Text | Google Scholar
Hoskins, L. C., Agustines, M., McKee, W. B., Boulding, E. T., Kriaris, M., and Niedermeyer, G. (1985). Mucinin hajoaminen ihmisen paksusuolen ekosysteemeissä. ABH-veriryhmän antigeenejä ja oligosakkarideja hajottavien ulostekantojen eristäminen ja ominaisuudet mucin-glykoproteiineista. J. Clin. Sijoittaa. 75, 944–953. doi: 10.1172 / JCI111795
PubMed Abstract | CrossRef Full Text | Google Scholar
Irmler, S., Raboud, S., Beisert, B., Rauhut, D., and Berthoud, H. (2008). Kahden Lactobacillus casei-geenin Kloonaus ja luonnehdinta, jotka koodaavat kystationiinilyaasia. Appl. Environ. Mikrobiolia. 74, 99–106. doi: 10.1128 / AEM.00745-07
PubMed Abstract | CrossRef Full Text | Google Scholar
Jagielski, V. (1877). Koumissin arvo pahoinvoinnin, oksentelun ja kyvyttömyyden säilyttää muita elintarvikkeita mahassa. Br. Med. J. 2, 919-921. doi: 10.1136 / bmj.2.887.919
PubMed Abstract | CrossRef Full Text | Google Scholar
Kato, S., Ishihara, T., Hemmi, H., Kobayashi, H., and Yoshimura, T. (2011). Muutokset d-aminohappojen pitoisuuksissa ja mikrobiyhteisön rakenteissa puna-ja valkoviinien käymisen aikana. J. Biosci. Bioeng. 111, 104–108. doi: 10.1016 / j.jbiosc.2010.08.019
PubMed Abstract | CrossRef Full Text | Google Scholar
Li, H., and Durbin, R. (2009). Nopea ja tarkka lyhyt lukusuuntaus Burrows-Wheeler-muunnoksella. Bioinformatiikka 25, 1754-1760. doi: 10.1093 / bioinformatiikka / btp324
PubMed Abstract | CrossRef Full Text | Google Scholar
Li, W., and Godzik, A. (2006). Cd-hit: nopea ohjelma suurten proteiini-tai nukleotidisekvenssien ryhmittelyyn ja vertailuun. Bioinformatiikka 22, 1658-1659. doi: 10.1093 / bioinformatiikka / btl158
PubMed Abstract / CrossRef Full Text | Google Scholar
Miyamoto, M., Seto, T., Nakajima, H., Burenjargal, S., Gombojav, A., Demberei, S., et al. (2010). Denaturoiva gradienttigeelielektroforeesianalyysi maitohappobakteereista ja hiivoista perinteisessä mongolialaisessa fermentoidussa maidossa. Food Sci. Technol. Res. 16, 319-326. doi: 10.3136 / fstr.16.319
CrossRef Full Text | Google Scholar
Mulder, N. J., and Apweiler, R. (2008). InterPro-tietokanta ja työkalut proteiinien domain-analyysiin. Kurr. Protokolla. Bioinformatiikka Luku 2, Unit2.7. doi: 10.1002 / 0471250953. bi0207s21
PubMed Abstract / CrossRef Full Text | Google Scholar
Noguchi, H., Park, J., and Takagi, T. (2006). Metageeni: prokaryoottinen geenilöydös ympäristön genomisekvensseistä. Nucleic Acids Res. 34, 5623-5630. doi: 10.1093 / nar/gkl723
PubMed Abstract / CrossRef Full Text | Google Scholar
Nurk, S., Bankevitš, A., Antipov, D., Gurevitš, A. A., Korobeynikov, A., Lapidus, A., et al. (2013). Yksisoluisten genomien ja minimetagenomien kokoaminen kimeerisistä MDA-tuotteista. J. Comput. Biol. 20, 714–737. doi: 10.1089 / cmb.2013.0084
PubMed Abstract | CrossRef Full Text | Google Scholar
Ott, A., Germond, J. E. ja Chaintreau, A. (2000). Asetaldehydin alkuperä maitokäymisen aikana käyttäen (13)C-merkittyjä lähtöaineita. J. Agric. Ruokakemikaalia. 48, 1512–1517. doi: 10.1021 / jf9904867
PubMed Abstract / CrossRef Full Text | Google Scholar
Pan, D. D., Zeng, X. Q., and Yan, Y. T. (2011). Koumissista, joka on mahdollisesti kolesterolia alentava probioottibakteeri, eristetyn Lactobacillus fermentum SM-7: n kuvaus. J. Sci. Elintarvike. Agric. 91, 512–518. doi: 10.1002 / jsfa.4214
PubMed Abstract | CrossRef Full Text | Google Scholar
Papadimitriou, K., Anastasiou, R., Maistrou, E., Plakas, T., Papandreou, N. C., Hamodrakas, S. J., et al. (2015). Streptococcus macedonicus ACA-DC 198: n plasmidi pSMA198: n horisontaalisen geeninsiirron kautta tapahtuva hankkiminen viittaa lajin maitoperäiseen alkuperään. PLoS ONE 10: e0116337. doi: 10.1371 / lehti.pone.0116337
PubMed Abstract | CrossRef Full Text | Google Scholar
Segata, N., Waldron, L., Ballarini, A., Narasimhan, V., Jousson, O., and Huttenhower, C. (2012). Metagenomisten mikrobiyhteisöjen profilointi käyttäen ainutlaatuisia kladispesifisiä merkkigeenejä. Nat. Menetelmät 9, 811-814. doi: 10.1038 / nmeth.2066
PubMed Abstract | CrossRef Full Text | Google Scholar
Sun, Z., Liu, W., Zhang, J., Yu, J., Zhang, W., Cai, C., et al. (2010). Koumissista Kiinassa eristettyjen dominoivien laktobasillien tunnistaminen ja luonnehtiminen. J. Kenr. Appl. Mikrobiolia. 56, 257–265. doi: 10.2323 / jgam.56.257
PubMed Abstract | CrossRef Full Text | Google Scholar
Thompson, J. (1879). Koumissin arvo sairauksien tuhlaamisessa. Br. Med. J. 1, 270. doi: 10.1136 / bmj.1.1466.270-C
PubMed Abstract | CrossRef Full Text | Google Scholar
Vincent, S. J., Faber, E. J., Neeser, J. R., Stingele, F., and Kamerling, J. P. (2001). Streptococcus macedonicus Sc136: n tuottaman eksopolysakkaridin rakenne ja ominaisuudet. Glykobiologia 11, 131-139. doi: 10.1093/glycob / 11.2.131
PubMed Abstract / CrossRef Full Text / Google Scholar
Wang, L., Christophersen, C. T., Sorich, M. J., Gerber, J. P., Angley, M. T., and Conlon, M. A. (2013). Sutterella spp.: n lisääntynyt runsaus ja Ruminococcus torques ulosteessa lasten autismikirjon häiriö. Mol. Autismi 4, 42. doi: 10.1186/2040-2392-4-42
PubMed Abstrakti / CrossRef kokoteksti / Google Scholar
Ward, T. L., Hosid, S., Ioshikhes, I., and Altosaar, I. (2013). Human milk metagenome: a functional capacity analysis. BMC-Mikrobioli. 13:116. doi: 10.1186/1471-2180-13-116
PubMed Abstrakti / CrossRef kokoteksti / Google Scholar
Watanabe, K., Fujimoto, J., Tomii, Y., Sasamoto, M., Makino, H., Kudo, Y., et al. (2009). Lactobacillus kisonensis sp. Marraskuuta., Lactobacillus otakiensis sp. Marraskuuta., Lactobacillus rapi sp. Marraskuuta. Lactobacillus sunkii sp. Marraskuuta., heterofermentatiiviset lajit eristetty sunki, perinteinen japanilainen suolakurkku. Int. J. Syst. Evol. Mikrobiolia. 59 (Pt 4), 754-760. doi: 10.1099 / ijs.0.004689-0
PubMed Abstract | CrossRef Full Text | Google Scholar
Wu, R., Wang, L., Wang, J., Li, H., Menghe, B., Wu, J., et al. (2009). Lactobacillien eristäminen ja alustava probioottivalinta koumissista Sisä-Mongoliassa. J. Perusmikrobioli. 49, 318–326. doi: 10.1002 / jobm.200800047
PubMed Abstract | CrossRef Full Text | Google Scholar
Zerbino, D. R., and Birney, E. (2008). Velvet: de novo short read assembly-algoritmit de Bruijn-kaavioilla. Genome Res. 18, 821-829. doi: 10.1101 / gr.074492.107
PubMed Abstract | CrossRef Full Text | Google Scholar
Zhang, W., Sun, Z., Sun, T., and Zhang, H. (2010a). Koumissista eristettyjen Lactobacillus casei-kantojen IOL-klustereiden PCR-seulonta ja sekvenssianalyysi. Folia Microbiol. (Praha) 55, 603-606. doi: 10.1007 / s12223-010-0097-3
PubMed Abstrakti / CrossRef kokoteksti / Google Scholar
Zhang, W., Yu, D., Sun, Z., Wu, R., Chen, X., Chen, W., et al. (2010b). Lactobacillus casei Zhang on uusi probioottikanta, joka on eristetty perinteisestä kotitekoisesta koumissista Sisä-Mongoliassa Kiinassa. J. Bakterioli. 192, 5268–5269. doi: 10.1128 / JB.00802-10
PubMed Abstract | CrossRef Full Text | Google Scholar
Zhang, W., and Zhang, H. (2011). ”Fermentation and komiss”, teoksessa Handbook of Food and Beverage Fermentation Technology, 2nd Edn, ed. Y. H. Hui (Boca Raton, FL: CRC Press), 165-172.
Google Scholar
Zhang, W. Y., Chen, Y. F., Zhao, W. J., Kwok, L. Y., and Zhang, H. P. (2015). Lactobacillus helveticus H9: n proteolyyttisen järjestelmän geeniekspressio maitokäymisen aikana. Ann. Mikrobiolia. 65, 1171–1175. doi: 10.3168 / jds.2014-8520
PubMed Abstract | CrossRef Full Text | Google Scholar
Zhao, W., Chen, Y., Sun, Z., Wang, J., Zhou, Z., Sun, T., et al. (2011). Lactobacillus helveticus H10: n täydellinen genomisekvenssi. J. Bakterioli. 193, 2666–2667. doi: 10.1128 / JB.00166-11
PubMed Abstract | CrossRef Full Text / Google Scholar