- Bevezetés
- anyagok és módszerek
- minták előkészítése
- gradiens hígítás és többszörös elmozdulás amplifikáció
- Könyvtárépítés és szekvenálás
- Adatelemzés
- szekvencia minőségellenőrzés és szűrés
- taxonómiai hozzárendelés és sokféleség
- olvassa el az összeszerelést, a Génjóslást és az annotációt
- nukleotidszekvencia csatlakozási számok
- eredmények
- kísérleti tervezés és szekvenálás
- taxonómiai annotáció
- Metagenomikai összeszerelés, Génjóslás és funkcionális annotáció
- Vita
- következtetés
- szerzői hozzájárulások
- összeférhetetlenségi nyilatkozat
- elismerés
- Kiegészítő anyag
Bevezetés
Koumiss, más néven chige, chigo, arrag vagy airag, mongol nyelven, a hagyományos erjesztett tejtermékek egyik típusa. Évszázadok óta népszerű étel Mongóliában és Kína belső-Mongóliájában (Zhang and Zhang, 2011). Ezekben a régiókban az emberek nagy ünnepségek és áldozati felajánlások során fogyasztották a koumissot (Zhang and Zhang, 2011). A koumiss termelésének legkorábbi feljegyzése a Han dinasztia (BC202-AD202). Ez a termék széles körben elterjedt a Yuan-dinasztia idején (AD1271-AD1368) (Zhang et al., 2010b). Manapság a koumiss a Mongólia és belső-mongólia helyi lakosainak közös étele, bár csak néhány ilyen területen gyártják ipari méretekben. A Koumiss nemcsak gazdag tápanyagokat biztosít, beleértve az esszenciális aminosavak és vitaminok magas tartalmát, hanem úgy gondolják, hogy enyhíti az orvosi állapotok széles skáláját, és előnyös a posztoperatív ellátásban (Jagielski, 1877; Thompson, 1879).
hagyományosan a koumissot általában fahordókban, állati bőrből vagy urnákból készült tartályokban állítják elő. Az erjedés természetes módon, környezeti hőmérsékleten történik, miután szűrt kancatejet adtak az Old koumiss tartályba, amely kiindulási tenyészetként szolgál (Zhang and Zhang, 2011). A Koumiss jó forrása a biotechnológiai potenciállal rendelkező új baktériumoknak (Zhang et al., 2010a; Pan et al., 2011). Ezért nagy érdeklődésre tart számot, hogy minél több erjedéssel összefüggő koumiss baktériumot tárjunk fel és őrizzünk meg. Az elmúlt évtizedekben számos tanulmányt végeztek a koumiss baktériumközösség vizsgálatára (Wu et al., 2009; Hao et al., 2010), főleg kultúra -, molekuláris biológia – és piroszekvenálás-alapú módszerekkel tanulmányozva (Sun et al., 2010). E különböző megközelítések közül a piroszekvenáláson alapuló módszer szolgáltatta a koumiss legátfogóbb mikrobiota profilját, függetlenül az egyes mikrobák fenotípusos tulajdonságaitól és tenyésztési problémáitól. A koumiss baktériumok által kódolt funkcionális gének spektruma és fermentációs képességük azonban továbbra is rosszul jellemezhető, különösen a ritka mikrobiális populációk esetében.
jelen tanulmány az egysejtű genomikai technikát alkalmazta a Mongóliából és belső-Mongóliából származó 10 koumiss minta bakteriális metagenomjának elemzésére. A jelenlegi munka a legmodernebb technológiákat alkalmazta a tejtermékek bakteriális sokféleségének vizsgálatában. Munkánk bebizonyította az alacsony bőségű taxonok felfedezésének megvalósíthatóságát az egysejtű metagenomikai megközelítés alkalmazásával. A biztató eredmények elősegítenék újszerű megközelítések kidolgozását és alkalmazását a hagyományos kutatási területek problémáinak kezelésében.
anyagok és módszerek
minták előkészítése
összesen 10 koumiss mintát gyűjtöttek Mongóliából (MG14, MG15, MG16, MG17 és MG18) és a kínai Belső-Mongóliából (Nm17, NM18, NM19, NM20 és NM21) a metagenomikai vizsgálathoz. A mintákat aszeptikusan gyűjtöttük és szárazjégben szállítottuk.
mindegyik minta egy milliliterét előkezeltük a Ward et al. (2013) néhány módosítással. Röviden, A mintákat jégfürdőben 3-5 percig felolvasztottuk. Miután a minták megolvadtak, kis sebességű centrifugálásnak vetették alá őket, hogy eltávolítsák a szennyeződéseket és az eukarióta sejtcsomókat. A prokarióta sejteket ezután a tejszérumokból pelletáltuk centrifugálással 13 000 g-on 15 percig. A pelleteket újra szuszpendáltuk 2 mL foszfátpufferelt sóoldatban (PBS) 1% Triton X-100-mal, és 2 órán át inkubáltuk 37cc-n, hogy a fennmaradó eukarióta sejteket lizáljuk. Ezt követően a baktériumokat 13 000 Ft-os centrifugálással 15 percig pelletáltuk, majd a pelleteket 500 Ft-os PBS-ben újra szuszpendáltuk. Végül a centrifugálási lépést még egyszer megismételtük a baktériumsejtek mosásához.
gradiens hígítás és többszörös elmozdulás amplifikáció
az alacsony bőségű baktériumok kimutatásához az egyes koumiss-mintákból származó bakteriális szuszpenziót sorozatosan hígítottuk a későbbi amplifikációs reakcióhoz. Az egyes minták sejtszámát nagyjából mikroszkóp alatt becsülték meg (Nikon, Tokió, Japán) egy sejtszámláló kamrával (Qiujing, Sanghaj, Kína). A hígítási lépést addig folytattuk, amíg az egyes baktériumszuszpenziókban a sejtszám el nem érte a körülbelül 100-at. A hígított sejtek többszörös elmozdulási amplifikációját a REPLI-g egysejtű készlet (Qiagen, Germantown, MD, USA) alkalmazásával hajtottuk végre a gyártó utasításainak megfelelően.
Könyvtárépítés és szekvenálás
az amplifikált DNS-t véletlenszerűen nyírtuk, és a körülbelül 500 bp-es fragmentumokat választottuk ki. A könyvtár építése után a PerkinElmer LabChip (GX Touch) és a StepOnePlusTM (real-Time PCR) rendszert használták a könyvtári minőség ellenőrzéséhez. Végül 125 bp párosított végű leolvasásokat szekvenáltunk az Illumina HiSeq 2500 platformon a gyártó utasításainak megfelelően.
Adatelemzés
szekvencia minőségellenőrzés és szűrés
a szekvenszer által generált nyers olvasmányok tartalmazhatnak mesterséges olvasásokat az adapter szennyeződéséről a könyvtár építése során. Ezért három lépést hajtottunk végre a kiváló minőségű tiszta olvasási adatkészlet elérése érdekében: (1) az adapter szennyeződése által okozott olvasások kiküszöbölése; (2) a Q30 phred-pontszám alatti átlagos pontszámmal rendelkező olvasmányok eltávolítása, amelyet a kiváló minőségű alap legalacsonyabb határértékének tekintettek; (3) az olvasások jelentős “N” – es felesleggel történő eltávolítása (az olvasás 5% – a). A downstream elemzés a tiszta adatokon alapult. Ezenkívül a Q30 és a GC-tartalom alapján kiszámították a statisztikai alapminőséget.
a gazdaszervezet genomjához igazításokat végeztünk a gazdaszervezet eredetű szennyező szekvenciák eltávolítására. Minden gazdaszervezetből származó olvasást elvetettek, mielőtt további összehasonlítást végeztek volna a baktériumokkal (vagy vírusokkal) Genom referencia szekvenciák. A pontosabb eredmények elérése érdekében a Burrows-Wheeler aligner (BWA) (0.97 a verzió) mem modellt használták az igazításokban (Li and Durbin, 2009).
taxonómiai hozzárendelés és sokféleség
a metaflan webes szoftvert használták a nemzetségek és fajok szintjeinek rendszertani hozzárendelésére (Segata et al., 2012). A fajok sokféleségének összehasonlításához a mintákon belül és a minták között az alfa – és béta-sokféleséget elemeztük az R-rel kapcsolatos csomag alapján.
olvassa el az összeszerelést, a Génjóslást és az annotációt
hogy átfogóbb információkat kapjunk, összeállítottuk a nyírt fragmentumokat genomba (contigs). Azonban több faj jelenléte miatt, ami gyakori a metagenomikus mintákban, javítottuk a bioinformatikai Genom-összeszerelési módszert, amelyet általában az egyes fajok elemzésére használnak a SPAdes (3.6.2 verzió), a házon belüli szkriptek és a metagenomikus adatbázisok integrálásával (Zerbino and Birney, 2008; Nurk et al., 2013).
a MetaGeneMark szoftvert használták az összeszerelt összefüggések gén-előrejelzésére (Noguchi et al., 2006). A redundáns géneket CD-HIT alkalmazásával távolítottuk el, 90 és 95% – os identitással (Li and Godzik, 2006; Fu et al., 2012). A gének relatív bőségét úgy határoztuk meg, hogy a kiváló minőségű szekvenálási leolvasásokat a génkatalógushoz igazítottuk ugyanazzal az eljárással. A downstream eltérés elemzése a gén relatív abundanciáin alapult. A gén annotációt úgy hajtottuk végre, hogy a kiváló minőségű szekvenciákat több nyilvános adatbázishoz igazítottuk (nevezetesen NCBI nem redundáns adatbázis, NR; Ortológ fehérjecsoportok klaszterei, fogaskerekek; Kyoto Encyclopedia of Genes and Genomes, KEGG) segítségével robbanás (Altschul et al., 1997). A domain keresés az Interproscan használatával történt (Mulder and Apweiler, 2008).
nukleotidszekvencia csatlakozási számok
az ebben a tanulmányban közölt szekvenciaadatokat az sra adatbázisba helyezték (csatlakozási szám.SRP083102).
eredmények
kísérleti tervezés és szekvenálás
a koumiss kis mennyiségben előforduló baktériumainak kimutatására az egysejtes amplifikációs technikát alkalmazták a minták metagenomjainak elemzésére. Három bakteriális szuszpenziót, amelyek mindegyike körülbelül 100 sejtből áll, független koumiss mintából származtattuk soros hígítással. Összesen 30 hígított szuszpenziót elemeztünk. Minden hígított minta különböző mintakódot kapott, azaz a minta azonosító számát, amelyet 1, 2 vagy 3 követett, amelyek a három különálló hígítást képviselik. Azon feltevés alapján, hogy néhány ritka faj jelen lesz az egyik hígításban, a sejtek többszörös elmozdulási amplifikációját végeztük; és körülbelül 5 Gb adatot generáltunk minden koumiss baktériumszuszpenzióhoz.
összesen 1 040 323 864 nyers olvasatot állítottak elő a 10 koumiss mintából (összesen 30 bakteriális szuszpenzió). Az egyes 100 cellás szuszpenziók olvasásának átlagos száma 34 677 462 volt (S1 kiegészítő táblázat). A nem minősített szekvenciák kivágása és szűrése után minden mintára 1 018 381 702 tiszta leolvasást kaptunk (S1 kiegészítő táblázat). A Shannon-index, A Simpson-index, a Chao1-index és a megfigyelt fajok száma (1-4. ábra) azt mutatta, hogy a legtöbb koumiss-minta Magas bakteriális biodiverzitással rendelkezik. A Shannon-Wiener diversity görbék azt mutatták, hogy a szekvencia mélysége minden minta esetében megfelelő volt (1.ábra).
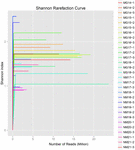
1. ÁBRA. Shannon ritkítási görbék, amelyek becsülik a koumiss minták mikrobiális sokféleségét.
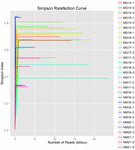
2. ÁBRA. Simpson ritkítási görbék, amelyek becsülik a koumiss minták mikrobiális sokféleségét.
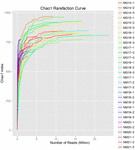
3. ÁBRA. Chao1 ritkítási görbék, amelyek becsülik a koumiss minták mikrobiális sokféleségét.
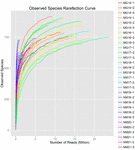
4. ÁBRA. Megfigyelt Fajok ritkulási indexe, amely megbecsüli a koumiss minták mikrobiális sokféleségét.
taxonómiai annotáció
a kiváló minőségű szekvenciákat különböző taxonómiai szintekhez rendeltük, hogy lehetővé tegyék a minta baktériumközösségeinek mélyreható elemzését. A koumiss biodiverzitásáról szóló néhány közzétett tanulmányra hivatkozva (Wu et al., 2009; Hao et al., 2010; Sun et al., 2010), az ismert és korábban nem jelentett koumiss-asszociált baktériumokat gyakori és ritka taxonokként osztályoztuk.
a kiváló minőségű szekvenciák 13 különböző nemzetséget képviseltek (5.ábra). Három közülük átlagosan több mint 1% – os relatív bőséggel rendelkezett, beleértve a lactobacillust (L.), A Lactococcust és a streptococcust. Különösen a lactobacillus és a Lactococcus volt a koumiss két leggyakoribb nemzetsége. A Lactobacillus aránya a mintákban 52,72-99,96% volt. E nemzetség két tagja, a L. helveticus és a L. kefiranofaciens dominált a legtöbb koumiss minta között (6.ábra). A faj L. buchneri főleg a mongol mintákban volt jelen, míg a Lactococcus lactis a mintavételi régiótól függetlenül a legtöbb mintában kimutatható volt.
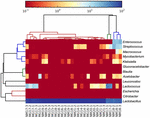
5. ÁBRA. Hőtérkép, amely a koumiss mintákban kimutatott baktériumok relatív bőségét mutatja nemzetség szintjén.
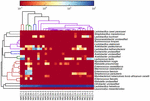
6. ÁBRA. Hőtérkép, amely a koumiss mintákban kimutatott baktériumok relatív bőségét mutatja fajok szintjén.
Metagenomikai összeszerelés, Génjóslás és funkcionális annotáció
a leolvasások összeszerelése 614 392 623 bp összeszerelési hosszt eredményezett. A szerelvények N50 értékei 5596 és 35 200 bp között mozogtak (S2 kiegészítő táblázat). A koumiss-mintákban az előre jelzett gének száma 10 347 és 34 547 között mozgott, átlagos hossza 647,82 és 985,33 bp között volt (S3 kiegészítő táblázat). Bár az empirikus génfunkcionális elemzések túlmutattak a jelenlegi vizsgálat hatókörén, a koumiss bakteriális mikrobiomokat a COG és a KEGG adatbázisok segítségével jegyeztük fel, amelyek nagyrészt a szekvencia homológián alapuló génfunkciót jósoltak.
az annotációs kimeneten összesen 545 egyezés mutatott magas homológiát a laktóz-hasznosító génekkel (cogs szénhidrát-és anyagcserekategória, G), és néhányuk ugyanabban a contig-ben található (1.táblázat).
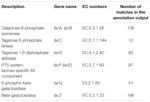
TÁBLÁZAT 1. A koumiss bakteriális metagenómában feljegyzett laktóz-anyagcserével kapcsolatos gének.
néhány más szekvencia kódolhat feltételezett géneket az aminosav-transzport és-metabolizmus (e) COGs kategóriájába, beleértve a kazein-lebontó proteinázoknak, a 4-18 maradék oligopeptidek felvételére szolgáló Opp rendszernek és az aminopeptidázoknak megfelelő szekvenciákat (pl. leucil-aminopeptidáz, peptidil-dipeptidáz A, aminopeptidáz N, prolin iminopeptidáz és endopeptidáz). Bár egyes szekvenciák nagy hasonlóságot mutattak az argininre, aszpartátra, metioninra és izoleucinra specifikus aminotranszferázokkal, csak egy szignifikáns egyezést azonosítottak, amely megfelelt egy feltételezett S. macedonicus eredetű I/II.osztályú doménnek (IPR004839), amely tirozinra és fenilalaninra specifikus aminotranszferázt tartalmazott. Végül számos szekvencia magas homológiát mutatott az aminosav-liázokkal, beleértve az S-ribozil-homocisztein-liázt, az argininoszukcinát-liázt, az aszpartát-ammónia-liázt, a cisztationin-gamma-liázt, a hisztidin-ammónia-liázt és az O-acetil-homozerint (tiol) – liázt.
Vita
a Koumiss népszerű hagyományos erjesztett tejtermék Mongóliában és Kína belső-Mongóliájában. Bár számos vizsgálatot végeztek a koumiss bakteriális sokféleségének vizsgálatára, kevés információt szereztek a koumiss mikrobiota genetikai kapacitásáról. Itt az egysejtű amplifikációs technikát alkalmaztuk a koumiss bakteriális metagenomok elemzésére, különös tekintettel az alacsony bőségű baktériumpopulációra.
a tipikus metagenomikai megközelítést korábban alkalmazták a koumiss mikrobiota leírására. A mély szekvencia elérésének magas költségei miatt azonban a minták ritka mikrobiota populációját gyakran nem fedik le megfelelően. Így mind a kisebbségi baktériumok filogén, mind funkcionális metagenomjai korlátozottak maradnak. Egysejtű amplifikációs módszerünk a minták soros hígítását 100 cellás szuszpenziókká tette. A hígított koumiss mintákban jelen lévő sejtek alacsony száma miatt csak korlátozott mennyiségű DNS-t extrahálnánk. Az amplifikációs lépés növelte az elemzendő DNS-anyagok mennyiségét, ezáltal megkönnyítette a percnyi DNS-mennyiséget tartalmazó minták metagenom-elemzését. Ennek a módszernek az egyik hátránya az volt, hogy nehéz volt biztosítani, hogy az egyes taxonok szekvenciái egyformán felerősödjenek a folyamat során; ezért az itt kapott eredmények csak a szekvenciák relatív bőségét tükrözhetik, de az azonosított taxonok vagy funkcionális gének abszolút mennyiségét nem. Ennek azonban kevés hatása lenne elemzésünkre, mivel a jelen tanulmány a ritka baktériumpopuláció fókuszálásában különbözik a többi publikált munkától. Az e munka által generált adatok kiegészítő információkat nyújtanak az alulreprezentált népesség számára. Úgy gondoljuk, hogy a jelenlegi megközelítés alkalmas a baktériumok sokféleségének jövőbeni elemzésére a különböző típusú ökológiai környezetekben.
a koumiss bakteriális mikrobiota főleg tejsavbaktériumokból (LAB) és néhány ecetsavbaktériumból áll (Zhang and Zhang, 2011). Ahogy az várható volt, az adatkészletünk többnyire a labort és az ecetsavbaktériumokat reprezentáló szekvenciákat tartalmazott. A Lactobacillus helveticusnak megfelelő szekvenciák domináltak az összes mintában. Különben is, szekvenciák képviselő faj, L. kefiranofaciens. L. buchneri. L. kefiranofaciens. Enterococcus (E.) casseliflavus. E. faecalis. E. faecium. Leuconostoc mesenteroides. Lactococcus lactis-t és Acetobacter pasteurianus-t is találtak. A különböző taxonok szekvenciáinak azonosítása nem biztos, hogy elegendő a baktériumok életképességének bemutatásához, mindazonáltal tükrözi a baktériumközösséget a fermentációs folyamat egy bizonyos pontján. Miyamoto et al. (2010) azt sugallja, hogy a baktériumok prevalenciája a végső fermentált termékekben a savas stressztűrésükhöz kapcsolódik. Általában, a laktobacillusok magasabb savtoleranciával rendelkeznek, mint a Lactococcusok, ami megmagyarázhatja az adatkészletünkben jelen lévő laktobacillus szekvenciák magas relatív bőségét. Másrészt az L gyakori előfordulása. a koumiss-I helveticus egybeesett az L. helveticus szekvenciák dominanciájának megfigyelésével.
adatkészletünkben egyes szekvenciák megfeleltek a L. otakiensis-nek, amely ritka faj, amelyet soha nem jelentettek koumissban vagy más tejelő fülkékben. A fajt először a hagyományos japán savanyúság (Watanabe et al., 2009). A recA gén alapján amplifikált fragmentum hosszúságú polimorfizmus profilozással fedezték fel. Azóta arról számoltak be, hogy ez a faj nem kapcsolódik más élelmiszerekkel kapcsolatos fülkékhez. Így valószínű, hogy ez a baktérium a savanyúság őshonos növényvilágához tartozik. Nem zárhatjuk ki azonban annak lehetőségét, hogy nem pusztán az alkalmazott kimutatási módszer érzékenysége miatt találták meg. A Lactobacillus otakiensis d-elágazó láncú aminosavakat képes előállítani, mint például a d-leucin, a D-allo-izoleucin és a d-valin (Doi et al., 2013). Potenciálisan felhasználható bizonyos erjesztett élelmiszerek termelési jellemzőinek javítására (Kato et al., 2011).
Vizsgálatunk az S. macedonicus fajt képviselő szekvenciákat is azonosította, amelyekről soha nem számoltak be koumiss-asszociált baktériumként. Ehelyett a görög juh-és kecskesajtokban (Georgalaki et al., 2000). Ennek a fajnak néhány tagja képes exopoliszacharidokat, bakteriocinokat előállítani (Vincent et al., 2001; Anastasiou et al., 2015) és gamma-amino-vajsav (Franciosi et al., 2015). Annak ellenére, hogy ezt a fajt gyakran elkülönítik erjesztett ételektől, a S. macedonicus eredeti rése ellentmondásos volt (Guarcello et al., 2016). Nem egészen a közelmúltig, Papadimitriou et al. (2015) azonosított egy megszerzett plazmidot, a pSMA198-at a Lactococcus lactis-ból. A plazmid valószínűleg egy ősi genetikai csereeseményen keresztül került át egy tejtermék-környezetben, utalva a S. macedonicus (Papadimitriou et al., 2015). Hasonló a S. thermophilushoz. Az S. macedonicus közeli rokonságban áll az S. bovis/S. equinus komplex kommenzális és opportunista kórokozóival.
a fajokat képviselő szekvenciák azonosítása Ruminococcus torques váratlan volt, mivel ez a baktérium általában a bélkörnyezetben található meg. Ez egy normális emberi bélmikroba, amely lebonthatja a mucin oligoszacharidokat szekréciós glikozidázok konstitutív termelésével (Hoskins et al., 1985). A legújabb klinikai bizonyítékok azt mutatják, hogy ennek a fajnak a székletbősége megváltozik autista spektrumzavarban szenvedő gyermekeknél; a rendellenességben betöltött szerepe azonban továbbra sem tisztázott (Wang et al., 2013).
meglepő módon néhány szekvencia potenciális kórokozókat képviselt. Például a Klebsiella pneumonia egy opportunista humán kórokozó, amely az emberi és állati belek körülbelül 40% – ában található meg. A Mycobacterium orygis, amelyet korábban oryx bacillusnak hívtak, a Mycobacterium tuberculosis komplex tagja, amely emberi tuberkulózist okozhat (Dawson et al., 2012). Ezt a két fajt a nyers tejben jelentették, a koumissot azonban nem. Ezért jelenlétük a hagyományos koumiss termelés során bekövetkező szennyeződésnek tulajdonítható, különösen nem vagy alacsony aszeptikus manipulációs körülmények között.
a baktériumok anyagcseréje fontos szerepet játszik a koumiss tulajdonságainak és minőségének kialakításában. Mikrobiális alapú folyamatok, például lipolízis és proteolízis szükségesek a koumiss aromás és aroma vegyületek szintetizálásához (Gesudu et al., 2016). Következetesen a bakteriális metagenom olyan szekvenciákat tartalmazott, amelyek potenciálisan kódolják a laktóz lebontását és a proteolitikus rendszereket. A viszonylag egyszerű laktóz katabolikus utakkal ellentétben a laboratóriumi proteolitikus rendszerek sokféle enzimből állnak (Chen et al., 2014). Összehasonlítva más észlelt koumiss laboratóriummal, a domináns faj L. helveticus magas proteolitikus aktivitással jellemezhető (Zhang et al., 2015). A legtöbb labor csak egy sejtburok proteinázzal rendelkezik, amely elindítja a tej kazein hidrolízisét, míg az L. helveticus ezen enzimek közül legalább kettőt tartalmaz, nevezetesen a PrtH-t és a PrtH2-t (Zhao et al., 2011). Így az L. helveticus erős proteolitikus fajainak és a proteolízishez kapcsolódó géneknek megfelelő szekvenciák nagy aránya összefügghet a koumiss viszonylag magas peptid-és szabad aminosavtartalmával.
az ipari méretű koumiss termelés egyik nehézsége az ízérzékelés ellenőrzése, mivel a koumissot hagyományosan természetes erjesztéssel készítették. Így nehéz volt meghatározni a legfontosabb ízösszetevők ízét és profilját, különösen a természetes szennyeződések jelenlétében. A kulcsfontosságú ízkomponensek előállítása az aminosavak, például az elágazó láncú aminosavak, a metionin és az aromás aminosavak fermentatív és enzimatikus lebomlásának eredménye (Ardo, 2006). Az ízkomponensek közé tartoznak az aldehidek, szerves savak és észterek, amelyeket transzamináz (AT)-út képez (Helinck et al., 2004). Ezt az utat egy transzamináz indítja el, amely katalizálja az aminosav átalakulását a megfelelőtá (Helinck et al., 2004). Adatkészletünk egyes szekvenciái megfeleltek a feltételezett elágazó láncú és aromás aminosav-aminotranszferázoknak. Különösen egy feltételezett aromás aminosav-aminotranszferáz I-t találtunk a koumiss-asszociált Fajok contig-jében, S. macedonicus. Mivel jelenlegi munkánk csak az In silico mikrobiomot kommentálta, adataink nem elegendőek annak bizonyításához, hogy ezek az azonosított génszekvenciák valóban funkcionálisak voltak a fent említett koumiss ízvegyületek előállításához. Ezeknek a géneknek a jelenléte mindazonáltal arra utal, hogy ezek lehetséges jelöltek az ilyen fermentációs tevékenységekre; mégis további kísérleti munkára lenne szükség pontos funkcionális szerepük tisztázásához.
ezenkívül más aminosav-konverziós útvonalaknak megfelelő szekvenciákat találtunk, beleértve az aminosav-liázokat és a treonin-aldolázt. Az előbbi enzim katalizálja a metionin metántiolná történő átalakulását (Irmler et al., 2008), így dimetil-diszulfid és dimetil-triszulfid képződést eredményezett (Fernandez et al., 2000), míg az utóbbi katalizálja a treonin glicinné és acetaldehiddé történő átalakulását (Ott et al., 2000). Hasonlóképpen, ezeknek a szekvenciáknak a koumiss mikrobiomon belüli elhelyezkedése nem szolgálhat határozott bizonyítékként tényleges biológiai szerepükben; jövőbeli kísérleti megerősítésre lesz szükség.
következtetés
ez a tanulmány módosított metagenomikai módszert használt a Mongóliából és a kínai Belső-Mongóliából gyűjtött koumiss minták bakteriális mikrobiomjának elemzésére. A koumiss ritka fajainak filogén és funkcionális metagenomjait egyaránt jellemeztük; és adatkészletünk olyan tulajdonságokat tükröz, amelyek a biotechnológia szempontjából érdekesek és potenciálisak. Tanulmányunk először bizonyította az egysejtű amplifikációs technikák beépítésének megvalósíthatóságát a koumiss bakteriális mikrobiota kimutatásában, valamint mikrobiális szennyeződések. Az itt kifejlesztett technikák felhasználhatók a jövőbeni tanulmányokban a koumiss mikrobiom változásainak figyelemmel kísérésére a fermentációs folyamat mentén, a kisebbségi mikrobiális populációra összpontosítva. Ezenkívül más omikai technikák, mint például a transzkriptomika, a metabolomika, összekapcsolhatók a jelenlegi metagenomikai elemzéssel a koumiss mikrobiom funkcióinak és metabolikus kapacitásának megerősítése érdekében.
technikailag a munka következő lépése a jelenlegi módszer optimalizálása lenne. Például a minta hígításának növelésével a DNS amplifikáció előtt javulhat a ritka és új baktériumok feltárásának esélye. Másrészt egy alternatív szekvenálási technika, amely hosszú olvasásokat generálhat, például a Pacific Biosciences single molecule, a valós idejű szekvenálási technológia alkalmazható a genom összeszerelési folyamatának javítására.
szerzői hozzájárulások
WZ és HZ tervezte a tanulmányt. WZ, GY, JY és L-YK írta a kéziratot. QH, WH, WL, BM és TS kísérleteket végeztek. A WZ és a QH elemezte az adatokat. Minden szerző áttekintette a kéziratot.
összeférhetetlenségi nyilatkozat
a szerzők kijelentik, hogy a kutatást olyan kereskedelmi vagy pénzügyi kapcsolatok hiányában végezték, amelyek potenciális összeférhetetlenségnek tekinthetők.
elismerés
ezt a kutatást a kínai Nemzeti Természettudományi Alapítvány támogatta (31571815 támogatás).
Kiegészítő anyag
a cikk kiegészítő anyaga megtalálható az interneten:: https://www.frontiersin.org/article/10.3389/fmicb.2017.00165/full#supplementary-material
B. A., B. A., B. A., B. A., B. A., B. A., B. A., B. A., B. A., B. A., B. A., B. A., B. A., B. A., B. A., B. A., B. A., B. A., et al. (1997). Gapped BLAST és PSI-BLAST: egy új generációs fehérje adatbázis kereső programok. Nukleinsavak Res. 25, 3389-3402. doi: 10.1093 / nar / 25.17.3389
PubMed absztrakt / CrossRef teljes szöveg / Google Tudós
Anasztasziou, R., Driessche, G. V., Boutou, E., Kazou, M., Alexandraki, V., Vorgias, C. E., et al. (2015). A streptococcus macedonicus tervezett törzsei az ozmotikus stressz-rezisztens fenotípus felé megtartják képességüket a bakteriocin macedocin hiperozmotikus körülmények között történő előállítására. J. Biotechnol. 212, 125–133. doi: 10.1016 / j. jbiotec.2015.08.018
PubMed Absztrakt / CrossRef Teljes Szöveg / Google Scholar
Ardo, Y. (2006). Ízképződés aminosav katabolizmussal. Biotechnol. ADV. 24, 238-242. doi: 10.1016 / j.biotechadv.2005.11.005
PubMed absztrakt / CrossRef teljes szöveg / Google Scholar
Chen, Y. F., Zhao, W. J., Wu, R. N., Sun, Z. H., Zhang, W. Y., Wang, J. C., et al. (2014). A Lactobacillus helveticus H9 proteom elemzése a sovány tej növekedése során. J. Dairy Sci. 97, 7413–7425. doi: 10.3168 / jds.2014-8520
PubMed absztrakt / CrossRef teljes szöveg / Google Scholar
Dawson, K. L., Bell, A., Kawakami, R. P., Coley, K., Yates, G. és Collins, D. M. (2012). A Mycobacterium orygis (M. tuberculosis komplex Fajok) átvitele egy tuberkulózisos betegből egy tejelő tehénbe Új-Zélandon. J. Clint. Mikrobiol. 50, 3136–3138. doi: 10.1128 / JCM.01652-12
PubMed absztrakt / CrossRef teljes szöveg / Google Tudós
Dóni, K., Mori, K., Mutagucsi, Y., Tashiro, K., Fudzsino, Y., Ohmori, T., et al. (2013). A hagyományos japán savanyúságból izolált Lactobacillus otakiensis JCM 15040T d-elágazó láncú aminosav-termelő genomszekvenciája. Genome Announc. 1, e546-e513. doi: 10.1128 / genomeA.00546-13
PubMed absztrakt / CrossRef teljes szöveg / Google Scholar
Fernandez, M., Van Doesburg, W., Rutten, G. A., Marugg, J. D., Alting, A. C., van Kranenburg, R., et al. (2000). A cisztationin béta-liázt kódoló Lactococcus lactis metC génjének molekuláris és funkcionális analízise. Appl. Environ. Mikrobiol. 66, 42–48. doi: 10.1128 / AEM.66.1.42-48.2000
PubMed absztrakt / CrossRef teljes szöveg / Google Scholar
E. Ferenczi, E., Carafa, I., Nardin, T., Schiavon, S., Poznanski, E., Cavazza, A., et al. (2015). Biodiverzitás és gamma-amino-vajsav termelés a hagyományos alpesi nyers tehéntej sajtokból izolált tejsavbaktériumok által. Biomed Res.Int. 2015, 625740. doi: 10.1155/2015/625740
PubMed absztrakt / CrossRef teljes szöveg / Google Tudós
Fu, L., Niu, B., Zhu, Z., Wu, S. és Li, W. (2012). CD-HIT: gyorsított a következő generációs szekvenálási adatok csoportosításához. Bioinformatics 28, 3150–3152. doi: 10.1093/bioinformatics/bts565
PubMed Abstract | CrossRef Full Text | Google Scholar
Georgalaki, M. D., Sarantinopoulos, P., Ferreira, E. S., De Vuyst, L., Kalantzopoulos, G., and Tsakalidou, E. (2000). Biochemical properties of Streptococcus macedonicus strains isolated from Greek Kasseri cheese. J. Appl. Microbiol. 88, 817–825. doi: 10.1046/j.1365-2672.2000.01055.x
PubMed Abstract | CrossRef Full Text | Google Scholar
Gesudu, Q. M., Zheng, Y., Xi, X. X., Hou, Q. C., Xie, H. Y., Huang, W. Q., et al. (2016). A bakteriális populáció szerkezetének és dinamikájának vizsgálata a belső-mongóliai hagyományos koumiss-ban egyetlen molekula valós idejű szekvenálásával. J. Dairy Sci. 99, 7852–7863. doi: 10.3168 / jds.2016-11167
PubMed absztrakt / CrossRef teljes szöveg / Google Scholar
Guarcello, R., Carpino, S., Gaglio, R., Pino, A., Rapisarda, T., Caggia, C., et al. (2016). Válogatott őshonos tejsavbaktériumok nagy gyári méretű alkalmazása a Pecorino Siciliano sajtgyártáshoz. Élelmiszer Mikrobiol. 59, 66–75. doi: 10.1016 / j.fm. 2016.05.011
PubMed absztrakt / CrossRef teljes szöveg / Google Tudós
Hao, Y., Zhao, L., Zhang, H., Zhai, Z., Huang, Y., Liu, X., et al. (2010). A baktériumok biológiai sokféleségének azonosítása koumiss-ban denaturáló gradiens gélelektroforézissel és fajspecifikus polimeráz láncreakcióval. J. Dairy Sci. 93, 1926–1933. doi: 10.3168 / jds.2009-2822
PubMed absztrakt / CrossRef teljes szöveg / Google Scholar
Helinck, S., Le Bars, D., Moreau, D. és Yvon, M. (2004). A termofil tejsavbaktériumok képessége aromavegyületek előállítására aminosavakból. Appl. Environ. Mikrobiol. 70, 3855–3861. doi: 10.1128 / AEM.70.7.3855-3861.2004
PubMed absztrakt / CrossRef teljes szöveg / Google Tudós
Hoskins, L. C., Agustines, M., McKee, W. B., Boulding, E. T., Kriaris, M. és Niedermeyer, G. (1985). A Mucin lebomlása az emberi vastagbél ökoszisztémákban. A széklet törzsek izolálása és tulajdonságai, amelyek lebontják az ABH vércsoport antigénjeit és oligoszacharidjait a mucin glikoproteinekből. J. Clint. Fektessen be. 75, 944–953. doi: 10.1172 / JCI111795
PubMed absztrakt / CrossRef teljes szöveg / Google Tudós
Irmler, S., Raboud, S., Beisert, B., Rauhut, D. és Berthoud, H. (2008). Két cisztationin-liázt kódoló Lactobacillus casei gén klónozása és jellemzése. Appl. Environ. Mikrobiol. 74, 99–106. doi: 10.1128 / AEM.00745-07
PubMed Absztrakt / CrossRef Teljes Szöveg / Google Scholar
Jagielski, V. (1877). A koumiss értéke hányinger, hányás, valamint a gyomorban lévő egyéb élelmiszerek megtartásának képtelensége kezelésében. Br. Med. J. 2, 919-921. doi: 10.1136 / bmj.2.887.919
PubMed absztrakt / CrossRef teljes szöveg / Google Tudós
Kato, S., Ishihara, T., Hemmi, H., Kobayashi, H. és Yoshimura, T. (2011). A D-aminosav koncentráció és a mikrobiális közösség szerkezetének megváltozása a vörös-és fehérborok erjedése során. J. Biosci. Bioeng. 111, 104–108. doi: 10.1016 / j. jbiosc.2010.08.019
PubMed absztrakt / CrossRef teljes szöveg / Google Scholar
Li, H. és Durbin, R. (2009). Gyors és pontos rövid olvasási igazítás a Burrows-Wheeler transzformációval. Bioinformatika 25, 1754-1760. doi: 10.1093 / bioinformatika / btp324
PubMed absztrakt / CrossRef teljes szöveg / Google Scholar
Li, W. és Godzik, A. (2006). Cd-hit: gyors program a nagy fehérje-vagy nukleotidszekvenciák csoportosítására és összehasonlítására. Bioinformatika 22, 1658-1659. doi: 10.1093 / bioinformatika / btl158
PubMed absztrakt / CrossRef teljes szöveg / Google Scholar
Mijamoto, M., Szeto, T., Nakadzsima, H., Burenjargal, S., Gombojav, A., Demberei, S., et al. (2010). Tejsavbaktériumok és élesztők denaturáló gradiens gélelektroforézis elemzése hagyományos mongol erjesztett tejben. Élelmiszer Sci. Technol. Res. 16, 319-326. doi: 10.3136 / fstr.16.319
CrossRef teljes szöveg / Google Scholar
Mulder, N. J. és Apweiler, R. (2008). Az InterPro adatbázis és a protein domain analízis eszközei. Curr. Protokoll. Bioinformatika 2. Fejezet, Unit2. 7. doi: 10.1002 / 0471250953. bi0207s21
PubMed absztrakt / CrossRef teljes szöveg / Google Tudós
Noguchi, H., Park, J. és Takagi, T. (2006). Metagén: prokarióta génkeresés környezeti Genom sörétes szekvenciákból. Nukleinsavak Res. 34, 5623-5630. doi: 10.1093 / nar / gkl723
PubMed absztrakt / CrossRef teljes szöveg / Google Tudós
Nurk, S., Bankevics, A., Antipov, D., Gurevics, A. A., Korobejnyikov, A., Lapidus, A., et al. (2013). Egysejtű genomok és mini-metagenomok összeállítása kiméra MDA termékekből. J. Comput. Biol. 20, 714–737. doi: 10.1089 / cmb.2013.0084
PubMed absztrakt / CrossRef teljes szöveg / Google Scholar
ott, A., Germond, J. E. és Chaintreau, A. (2000). Az acetaldehid eredete a tej erjesztése során (13)C-jelölt prekurzorok felhasználásával. J. Agric. Élelmiszer-Kémia. 48, 1512–1517. doi: 10.1021 / jf9904867
PubMed absztrakt / CrossRef teljes szöveg / Google Tudós
Pan, D. D., Zeng, X. Q. és Yan, Y. T. (2011). A koumiss-ból izolált Lactobacillus fermentum SM-7 jellemzése, potenciális probiotikus baktérium koleszterinszint-csökkentő hatással. J. Sci. Kaja. Agric. 91, 512–518. doi: 10.1002 / jsfa.4214
PubMed Absztrakt / CrossRef Teljes Szöveg / Google Tudós
Papadimitriou, K., Anastasiou, R., Maistrou, E., Plakas, T., Papandreou, N. C., Hamodrakas, S. J., et al. (2015). A Psma198 plazmid streptococcus macedonicus ACA-DC 198 általi horizontális géntranszfer útján történő megszerzése a faj tejszerű eredete felé mutat. PLoS egy 10: e0116337. doi: 10.1371 / folyóirat.pone.0116337
PubMed absztrakt / CrossRef teljes szöveg / Google Tudós
Segata, N., Waldron, L., Ballarini, A., Narasimhan, V., Jousson, O. és Huttenhower, C. (2012). Metagenomikus mikrobiális közösség profilozása egyedi klád-specifikus marker gének felhasználásával. Nat. Módszerek 9, 811-814. doi: 10.1038 / nmeth.2066
PubMed absztrakt / CrossRef teljes szöveg / Google Tudós
Z., Liu, W., Zhang, J., Yu, J., Zhang, W., Cai, C., et al. (2010). A kínai koumissból izolált domináns laktobacillusok azonosítása és jellemzése. J. Gen. Appl. Mikrobiol. 56, 257–265. doi: 10.2323 / jgam.56.257
PubMed Absztrakt / CrossRef Teljes Szöveg / Google Tudós
Thompson, J. (1879). A koumiss értéke a betegségek pazarlásában. Br. Med. J. 1, 270. doi: 10.1136 / bmj.1.1466.270-C
PubMed absztrakt / CrossRef teljes szöveg / Google Tudós
Vincent, S. J., Faber, E. J., Neeser, J. R., Stingele, F. és Kamerling, J. P. (2001). A Streptococcus macedonicus Sc136 által termelt exopoliszacharid szerkezete és tulajdonságai. Glikobiológia 11, 131-139. doi: 10.1093 / glycob/11.2.131
PubMed absztrakt / CrossRef teljes szöveg / Google Tudós
Wang, L., Christophersen, C. T., Sorich, M. J., Gerber, J. P., Angley, M. T. és Conlon, M. A. (2013). A Sutterella spp. és Ruminococcus nyomaték az autizmus spektrum zavarban szenvedő gyermekek székletében. Mol. Autizmus 4, 42. doi: 10.1186/2040-2392-4-42
PubMed absztrakt / CrossRef teljes szöveg / Google Tudós
Ward, T. L., Hosid, S., Ioshikhes, I. és Altosaar, I. (2013). Humán tej metagenom: funkcionális kapacitás elemzés. BMC Mikrobiol. 13:116. doi: 10.1186/1471-2180-13-116
PubMed absztrakt / CrossRef teljes szöveg / Google Tudós
T. A., J., Tomii, Y., Sasamoto, M., Makino, H., Kudo, Y., et al. (2009). Lactobacillus kisonensis sp. November., Lactobacillus otakiensis sp. November., Lactobacillus rapi sp. November. és a Lactobacillus sunkii sp. November., a sunkiból izolált heterofermentatív fajok, egy hagyományos japán savanyúság. Int. J. Syst. Evol. Mikrobiol. 59 (Pt 4), 754-760. doi: 10.1099 / ijs.0.004689-0
PubMed absztrakt / CrossRef teljes szöveg / Google Tudós
Wu, R., Wang, L., Wang, J., Li, H., Menghe, B., Wu, J., et al. (2009). A belső-mongóliai koumissból származó laktobacillusok izolálása és előzetes probiotikus szelekciója. J. Alapvető Mikrobiol. 49, 318–326. doi: 10.1002 / jobm.200800047
PubMed absztrakt / CrossRef teljes szöveg / Google Scholar
Zerbino, D. R. és Birney, E. (2008). Velvet: algoritmusok de novo rövid olvasási összeállításhoz de Bruijn grafikonok segítségével. Genome Res. 18, 821-829. doi: 10.1101 / g.074492.107
PubMed absztrakt / CrossRef teljes szöveg / Google Tudós
Zhang, W., Sun, Z., Sun, T. és Zhang, H. (2010a). A koumiss-ból izolált Lactobacillus casei törzsek IOL-klasztereinek PCR-szűrése és szekvenciaanalízise. Folia Microbiol. (Praha) 55, 603-606. doi: 10.1007 / s12223-010-0097-3
PubMed absztrakt / CrossRef teljes szöveg / Google Tudós
Csang, W., Yu, D., Sun, Z., Wu, R., Chen, X., Chen, W., et al. (2010b). A Lactobacillus casei Zhang teljes genomszekvenciája, egy új probiotikus törzs, amelyet a hagyományos házi készítésű koumissból izoláltak Belső-Mongóliában, Kínában. J. Bakteriol. 192, 5268–5269. doi: 10.1128 / JB.00802-10
PubMed absztrakt / CrossRef teljes szöveg / Google Tudós
Zhang, W. és Zhang, H. (2011). “Fermentation and koumiss,” in Handbook of Food and Beverage Fermentation Technology, 2. kiadás, Szerk. Y. H. Hui (Boca Raton, FL: CRC Press), 165-172.
Google Scholar
Zhang, W. Y., Chen, Y. F., Zhao, W. J., Kwok, L. Y. és Zhang, H. P. (2015). A Lactobacillus helveticus H9 proteolitikus rendszerének génexpressziója a tej erjedése során. Ann. Mikrobiol. 65, 1171–1175. doi: 10.3168 / jds.2014-8520
PubMed absztrakt / CrossRef teljes szöveg / Google Scholar
Zhao, W., Chen, Y., Sun, Z., Wang, J., Zhou, Z., Sun, T., et al. (2011). A Lactobacillus helveticus H10 teljes genomszekvenciája. J. Bakteriol. 193, 2666–2667. doi: 10.1128 / JB.00166-11
PubMed Absztrakt / CrossRef Teljes Szöveg / Google Tudós